Мотивы в белках
Задание 1
Для работы был взят домен PF01298 - C-lobe and N-lobe beta barrels of Tf-binding protein B. Описание:
Bacterial lipoproteins represent a large group of specialized membrane proteins that perform a variety of functions including maintenance and stabilization of the cell envelope, protein targeting and transit to the outer membrane, membrane biogenesis, and cell adherence [1]. Pathogenic Gram-negative bacteria within the Neisseriaceae and Pasteurellaceae families rely on a specialized uptake system, characterized by an essential surface receptor complex that acquires iron from host transferrin (Tf) and transports the iron across the outer membrane. They have an iron uptake system composed of surface exposed lipoprotein, Tf-binding protein B (TbpB), and an integral outer-membrane protein, Tf-binding protein A (TbpA), that together function to extract iron from the host iron binding glycoprotein (Tf). TbpB is a bilobed (N and C lobe) lipid-anchored protein with each lobe consisting of an eight-stranded beta barrel flanked by a "handle" domain made up of four (N lobe) or eight (C lobe) beta strands [2]. TbpB extends from the outer membrane surface by virtue of an N-terminal peptide region that is anchored to the outer membrane by fatty acyl chains on the N-terminal cysteine and is involved in the initial capture of iron-loaded Tf [3]. This domain family is found in C and N lobe eight stranded beta barrel region of TbpB proteins. The eight-stranded barrel domains in N and C lobe draw comparisons to eight-stranded beta barrel outer-membrane protein W (OmpW). However, the barrel domains of TbpB have the hydrophobic residues line the inner surface of the beta barrels to create a stable hydrophobic core [2].
В выравнивании SEED содержится 54 последовательности. После использования коамнды remove rebundancy их осталось также 54.
Если поставить порог идентичности 100%, то не высвечивается ни одна позиция выравнивания. Если уменьшить до 90%, высвечиваются изолированные позиции. Если уменьшить до 80%, выделяется участок выравнивания с 175 по 180 позиции, в котором высвечивается 4 консервантивных позиции. При уменьшении порога до 55% высвечивается также 182 позиция. На этом я решила остановиться.
Паттерн выбранного мотива имеет вид GN.F.G.A (175-182 позиции).
Поиск в Jalview выдает 15 совпадений. Все они находятся на участке 175-182.
Далее паттерн был переведен в формат Prosite: G-N-x-F-x-G-x-A.
Было найдено 215 совпадений в 213 последовательностях.
Задание 2
Было построено дерево по последовательностям и выбрана ветвь для дальнейшей работы (покрашена в синий на рисунке 1).
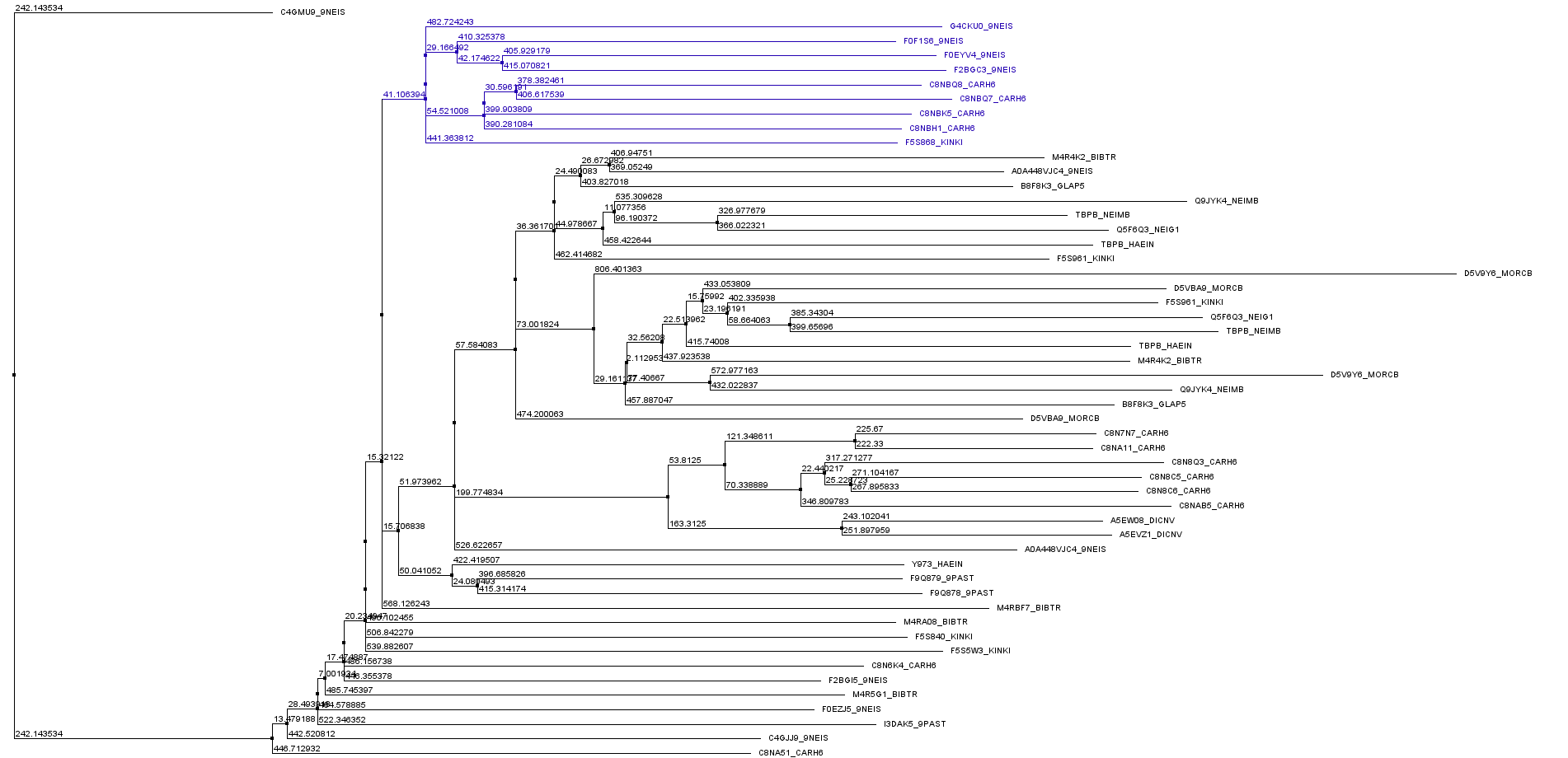
Рисунок 1. Дерево, посторенное по мотивам белков.
Выбранная ветвь содержит 9 последовательностей. Первые 6 позиций выравнивания этих последовательностей консервативны и образуют паттерн [MV]P[TMASG][TSN]G[TKIVS]. Он встречается во всех последовательностях клады и еще 4 за ее пределами. Таким образом, он достаточно специфичен.
Задание 3
Для работы был взят идентификатор P47908. Он принадлежит фактору гибернации рибосомы из организма Picosynechococcus sp. PCC 7002. Его функция заключается в том, чтобы связываться с рибосомой и подавлять трансляцию при определенных условиях. Для него была получена таблица итераций PSI-BLAST:
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 24 | P30334.1 | 0.004 | P47908.2 | 3,00E-143 |
| 2 | 28 | P9WMA8.1 | 3,00E-06 | P47908.2 | 5,00E-88 |
| 3 | 28 | P24694.1 | 4,00E-21 | P47908.2 | 4,00E-82 |
| 4 | 28 | P24694.1 | 4,00E-21 | P47908.2 | 4,00E-82 |
Результат стабилизировался на 4 итерации. Найденные белки являются факторами гибернации рибосом или другими белками, которые связываются с рибосомами. Таким образом, все они с большой вероятностью принадлежат к одному симейству.
Задание 4
Были скачаны последовательности белков, содержащих домен PF01298 и принадлежащих бактериям из семейства Paracoccacea. Всего было получено 113 последовательностей.
Для запуска МЕМЕ использовалась следующая команда:
meme export.fasta -o meme_results -mod anr -minw 4 -maxw 8 -nmotifs 4
Были найдены следующие мотивы:
FYGPBAEE E-value = 1.4e-180
LAACGGGG E-value = 2.5e-177
PTTGSATY E-value = 5.9e-052
DIALTABF E-value = 2.8e-053
Полную выдачу можно посмотреть по ссылке. Полученные диаграммы LOGO показаны на рисунке 2.

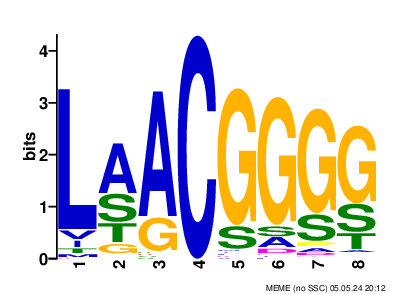

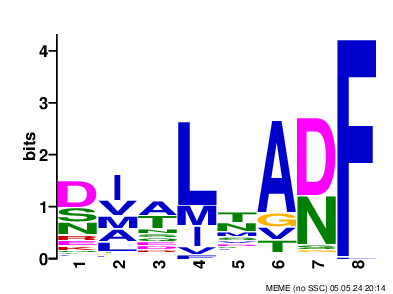
Рисунок 2. Диаграммы LOGO найденных мотивов. Слева-направо: FYGPBAEE, LAACGGGG, PTTGSATY, DIALTABF.
Для запуска FIMO использовалась следующая команда:
fimo meme_results/meme.txt export.fasta
Были получены следующие результаты: FYGPBAEE - 101 находка, LAACGGGG - 113 находок, PTTGSATY - 64 находки, DIALTABF - 110 находок. Полную выдачу можно посмотреть по ссылке.
Задание 5
Для работы был взят референсный геном бактерии Kocuria rhizophila. Был получен файл со сайтами длины 4 без повторений. Была запущена следующая программа:
cbcalc -s list.txt -M -o cbcres.txt rhizophila_reference.fna
Выдачу можно посмотреть по ссылке. Гистограмма, посторенная по результатам, показана на рисунке 3. Видно, что сайт GATC в геноме моей бактерии не очень распространен относительно других сайтов.
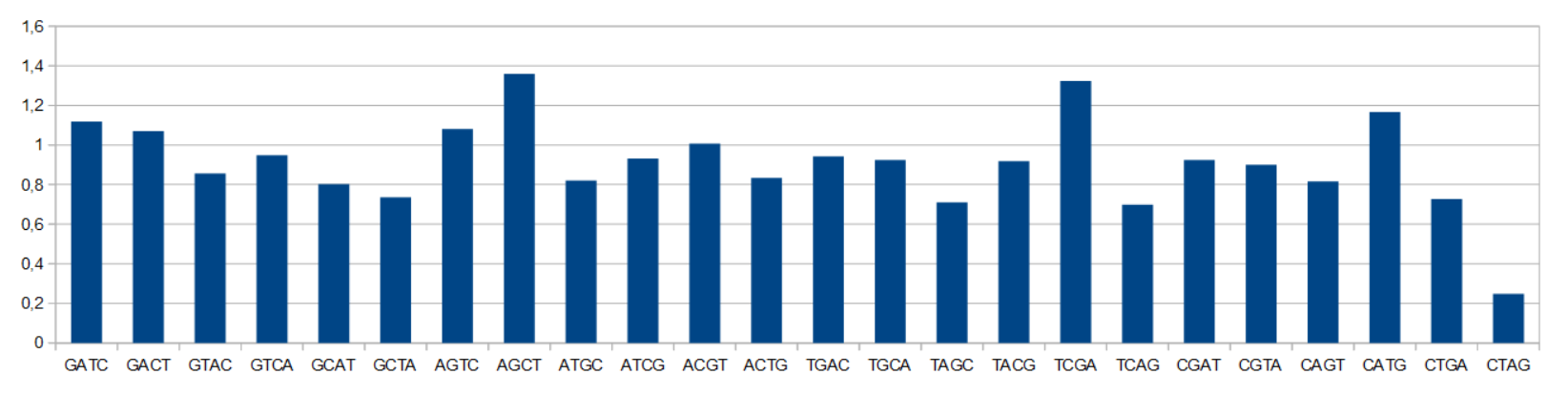
Рисунок 3. Гистограмма контрастов obs/exp для сайтов длины 4.