Составление списка гомологичных белков, включающих паралоги
Для выполнения задания была создана база данных из протеомов восьми бактерий:
makeblastdb -in proteoms.fasta -dbtype prot
Поиск осуществлялся с помощью следующей команды:
blastp -db .\proteoms.fasta -query .\CLPX_ECOLI.fa -evalue 0.001
Были получены следующие находки:
sp|Q3SI99|CLPX_THIDA ATP-dependent Clp protease ATP-binding subun... 642 0.0
sp|Q5P160|CLPX_AROAE ATP-dependent Clp protease ATP-binding subun... 619 0.0
sp|Q92QQ2|CLPX_RHIME ATP-dependent Clp protease ATP-binding subun... 596 0.0
sp|Q6G3Z2|CLPX_BARHE ATP-dependent Clp protease ATP-binding subun... 588 0.0
sp|P44838|CLPX_HAEIN ATP-dependent Clp protease ATP-binding subun... 587 0.0
sp|Q8G0I5|CLPX_BRUSU ATP-dependent Clp protease ATP-binding subun... 586 0.0
sp|A5FX05|CLPX_ACICJ ATP-dependent Clp protease ATP-binding subun... 583 0.0
sp|Q9JTX8|CLPX_NEIMA ATP-dependent Clp protease ATP-binding subun... 557 0.0
sp|Q6G5G0|HSLU_BARHE ATP-dependent protease ATPase subunit HslU O... 97.1 4e-22
sp|Q5P503|HSLU_AROAE ATP-dependent protease ATPase subunit HslU O... 93.6 7e-21
sp|Q92TA7|HSLU_RHIME ATP-dependent protease ATPase subunit HslU O... 92.8 1e-20
tr|A5FYD7|A5FYD7_ACICJ ATP-dependent protease ATPase subunit HslU... 92.0 2e-20
sp|P43773|HSLU_HAEIN ATP-dependent protease ATPase subunit HslU O... 92.0 2e-20
sp|Q8FY12|HSLU_BRUSU ATP-dependent protease ATPase subunit HslU O... 90.9 4e-20
tr|Q3SFW1|Q3SFW1_THIDA ATP-dependent protease ATPase subunit HslU... 86.7 1e-18
tr|A0A0H3GCZ6|A0A0H3GCZ6_BRUSU ATP-dependent zinc metalloprotease... 45.4 3e-05
tr|A0A0H3LXZ4|A0A0H3LXZ4_BARHE ATP-dependent zinc metalloprotease... 45.4 3e-05
tr|Q92M98|Q92M98_RHIME ATP-dependent zinc metalloprotease FtsH OS... 45.4 3e-05
tr|A5FVF9|A5FVF9_ACICJ ATP-dependent zinc metalloprotease FtsH OS... 44.7 6e-05
sp|Q6G5R1|RUVB_BARHE Holliday junction branch migration complex s... 43.5 1e-04
tr|Q3SJR4|Q3SJR4_THIDA ATP-dependent zinc metalloprotease FtsH OS... 43.5 1e-04
sp|Q9JUB0|RUVB_NEIMA Holliday junction branch migration complex s... 42.7 2e-04
sp|P71377|FTSH_HAEIN ATP-dependent zinc metalloprotease FtsH OS=H... 42.7 2e-04
tr|Q3SJH1|Q3SJH1_THIDA ATP-dependent Clp protease, ATP-binding su... 42.7 2e-04
tr|A0A0U1RJ22|A0A0U1RJ22_NEIMA Replication-associated recombinati... 40.8 8e-04
tr|Q3SKL1|Q3SKL1_THIDA Chaperone protein ClpB OS=Thiobacillus den... 40.8 0.001
Реконструкция и визуализация
По полученным последовательностям было построено дерево. Его формулу Newick можно найти по ссылке.
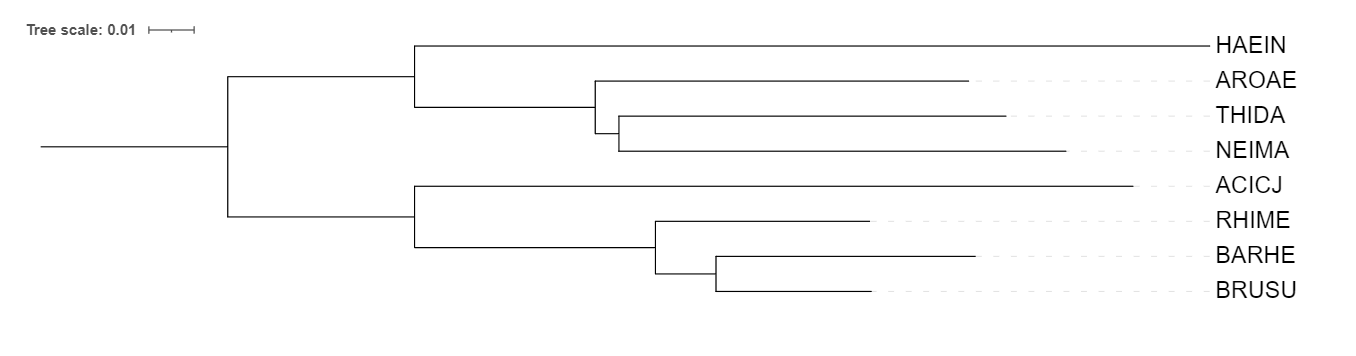
В дереве есть 4 группы ортологов: белки CLPX, HSLU, FTSH и RUVB. Они выделены разными цветами на рисунке 1. Любые два белка из каждой группы являются ортологами. Паралогами являются белки из одной бактерии, например, Q3SKL1_THIDA и Q3SJH1_THIDA; RUVB_BARHE и HSLU_BARHE; CLPX_RHIME и HSLU_RHIME.
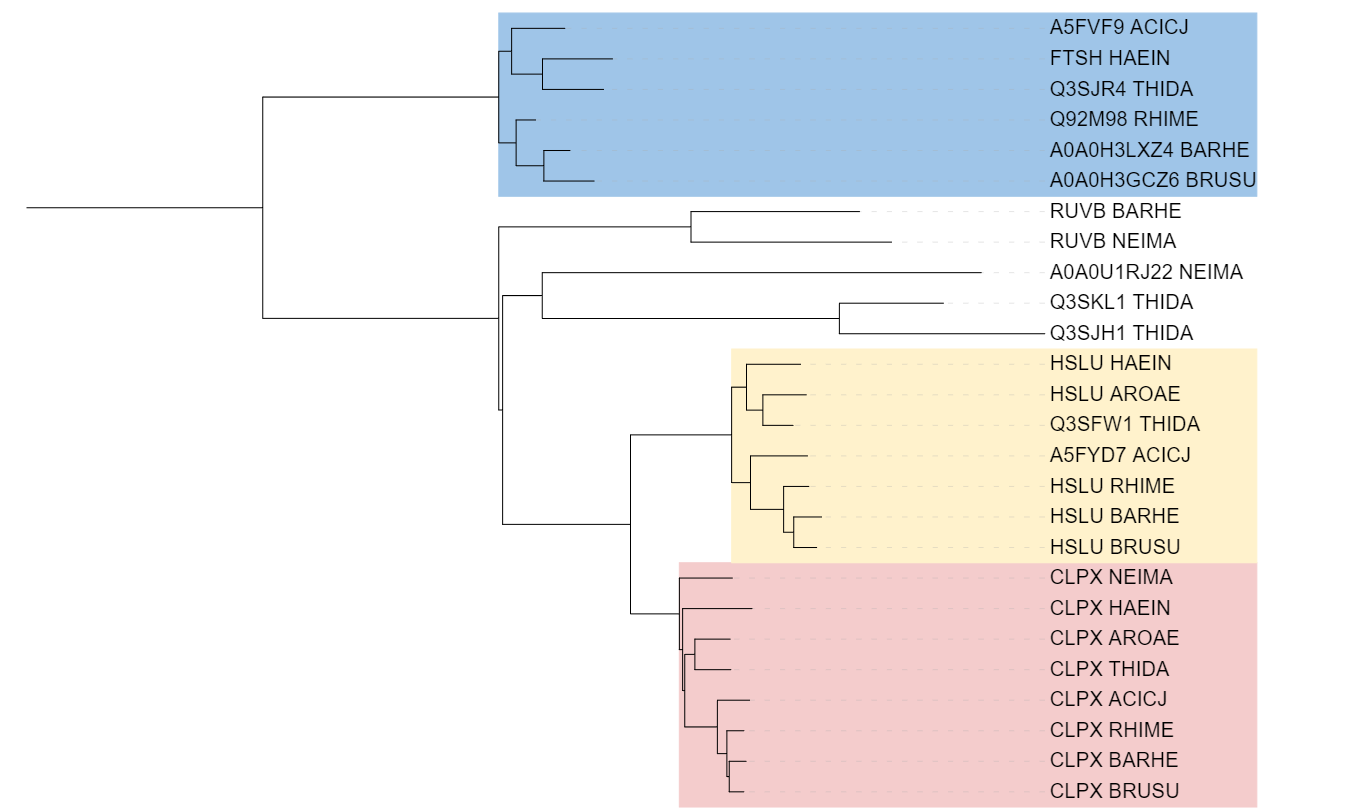
На рисунке 2 показано дерево со схлопнутыми ортологическими группами. В состав ветви CLPX входят белки всех восьми бактерий; в состав ветви HSLU - белки всех бактерий, кроме NEIMA; в состав ветви FTSH - белки всех бактерий, кроме NEIMA и ARORE. Ортологическая группа, содержащая белки RUVB_BARHE и RUVB_NEIMA, показана целиком.
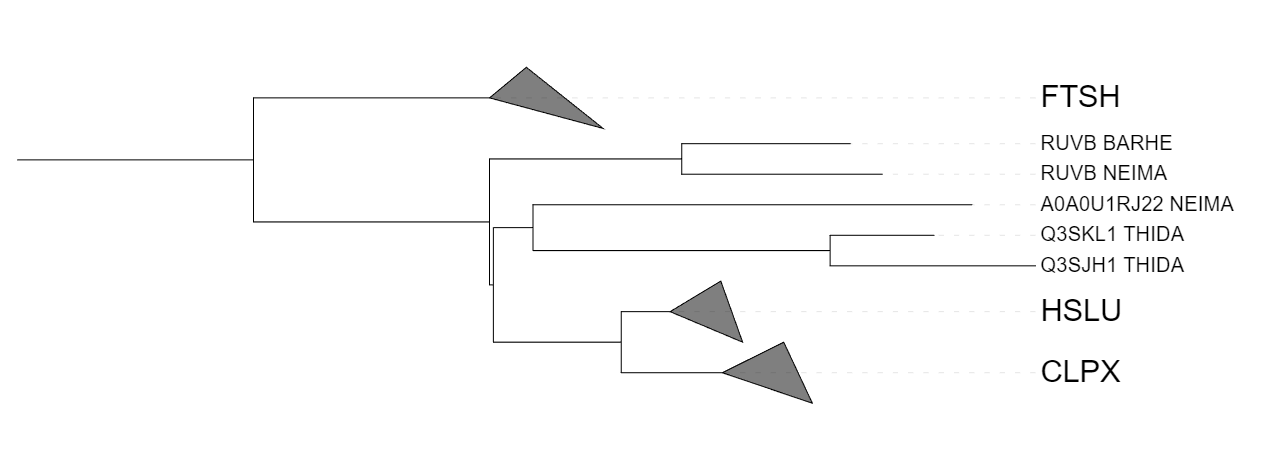
На рисунке 3 показано дерево, построенное по генам 16s рРНК бактерий. Видно, что филогения белков HSLU соответствует филогении бактерий: если учесть, что у NEIMA нет этого белка, дерево по 16s рРНК полностью совпадает с фрагментом дерева, содержащим эту ортологическую группу. Для группы CLPX филогения белков совпадает с филогенией 16s рРНК только для бактерий ACICJ, RHIME, BARHE, BRUSU. Остальные бактерии образуют монофилетическую группу, а их белки - нет. Филогения белков FTSH совпадает с филогенией бактерий, за исключением белка ACICJ, который должен быть ближе к белкам бактерий RHIME, BARHE и BRUSU.