Базы данных KEGG и GO
Входные данные представляли собой список из 133 идентификаторов.
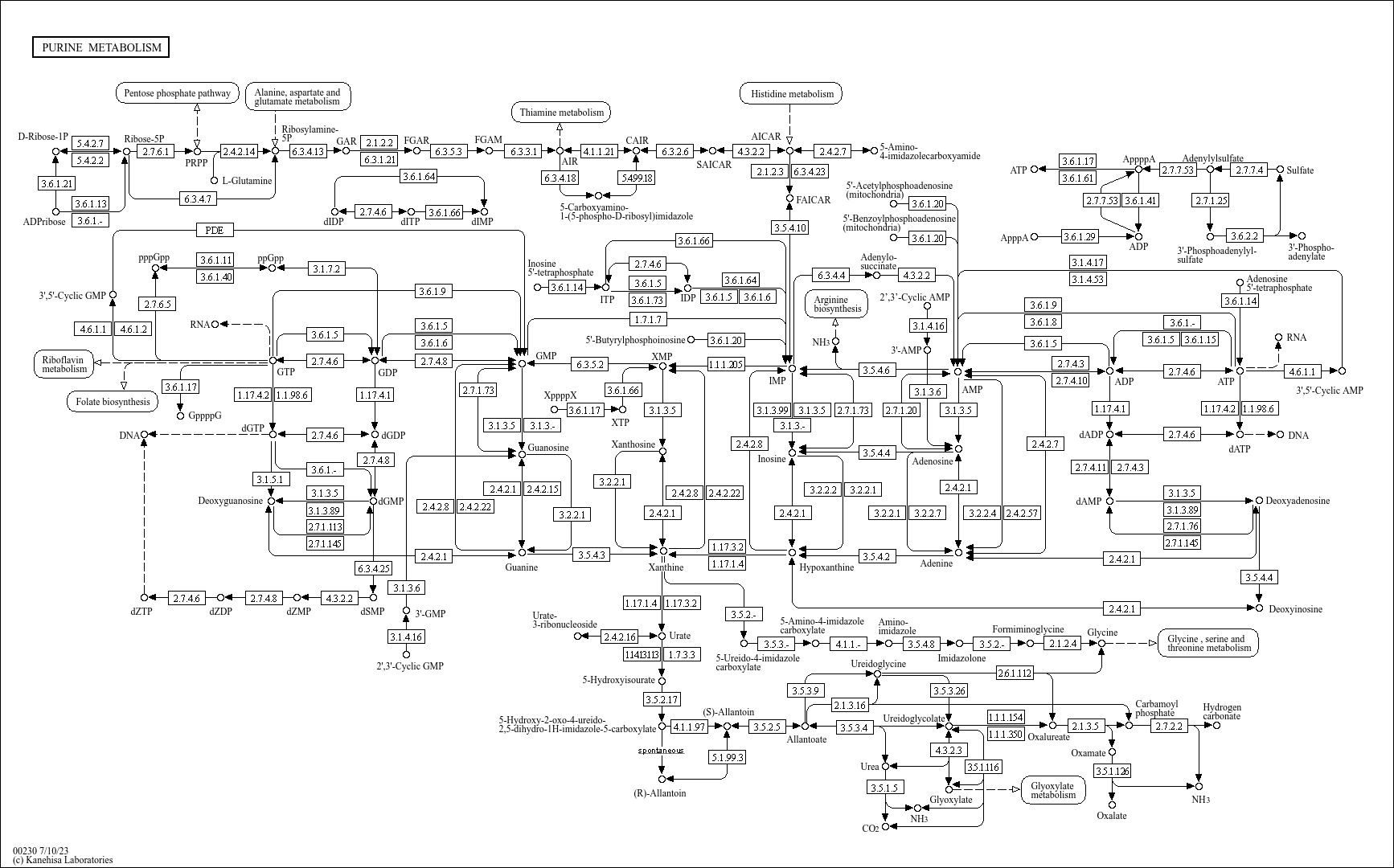
Сначала я проанализировала мои белки с помощью STRING для того, чтобы определить, в каких биологический процессах они участвуют. Функциональное обогащение показало (Gene Ontology), что большая часть белков (102) участвует в метаболизме нуклеотидов (см. рис. 1). Анализ путей KEGG показал, что наиболее полно представленный путь - метаболизм пуринов. Из 127 белков, участвующих в этом пути, 126 входят в список. Карта пути, полученная из базы данных KEGG показана на рисунке 2.
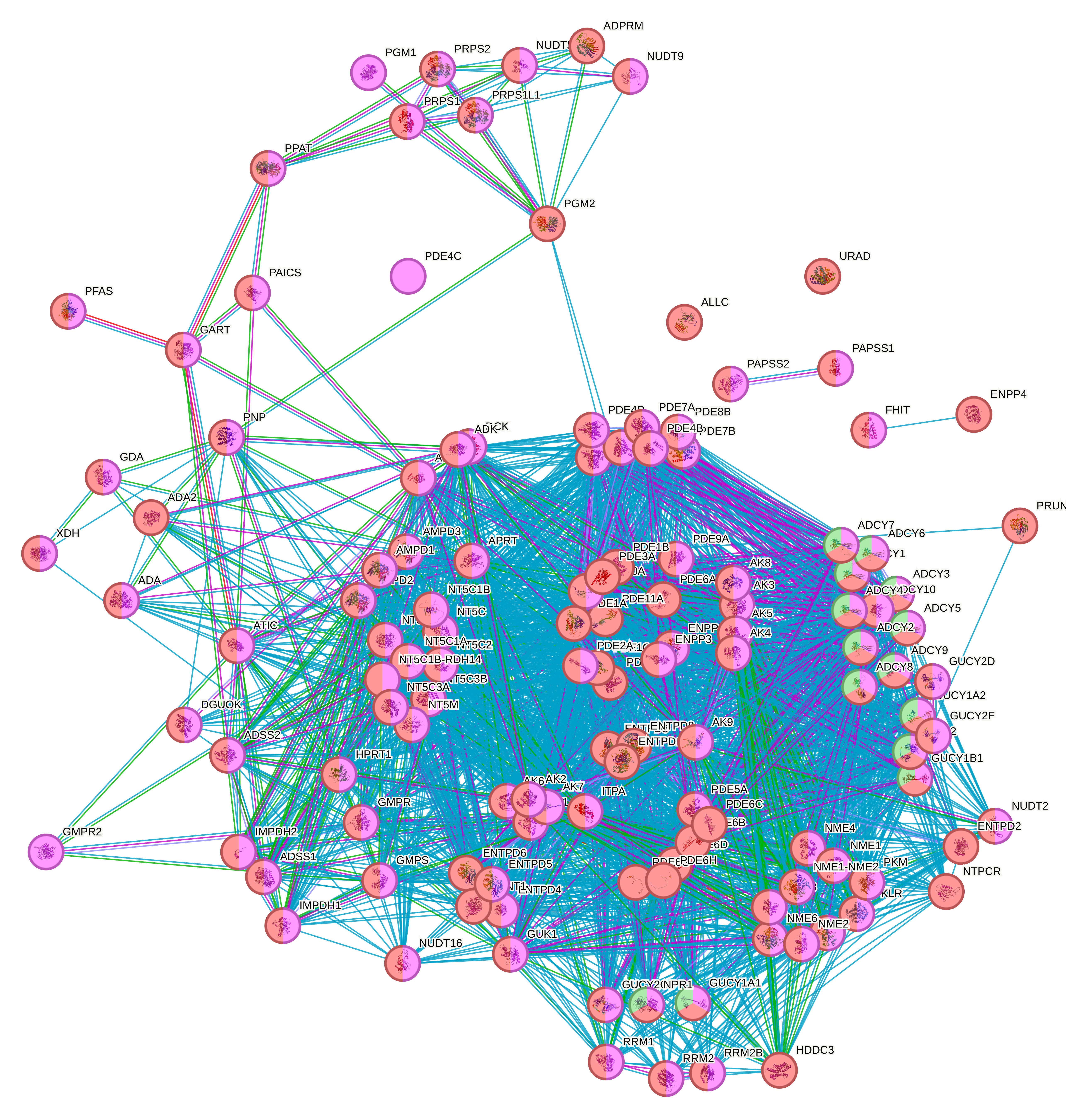
Далее я решила определить функции белков с помощью Reacfoam и сравнить результаты. Анализ подтвердил наличие большого количества белков, связанных с метаболизмом нуклеотидов, но показал также наличие значительно количества белков, выполняющих другие функции, например, участвующих в мышечных сокращениях (см. рис. 3). Я посмотрела, есть ли эта категория в выдаче STRING. Оказалась, что там есть 14 белков, участвующих в сокращении гладких мышц сосудов (всего белков, участвующих в этом пути KEGG 14). Эти белки также относятся метаболизму нуклеотидов. Дело в том, что эти белки участвуют в самых разнообразных процессов. Среди них есть в том числе аденилатциклазы (ADCY), которые входят в огромное множество сигнальных каскадов.
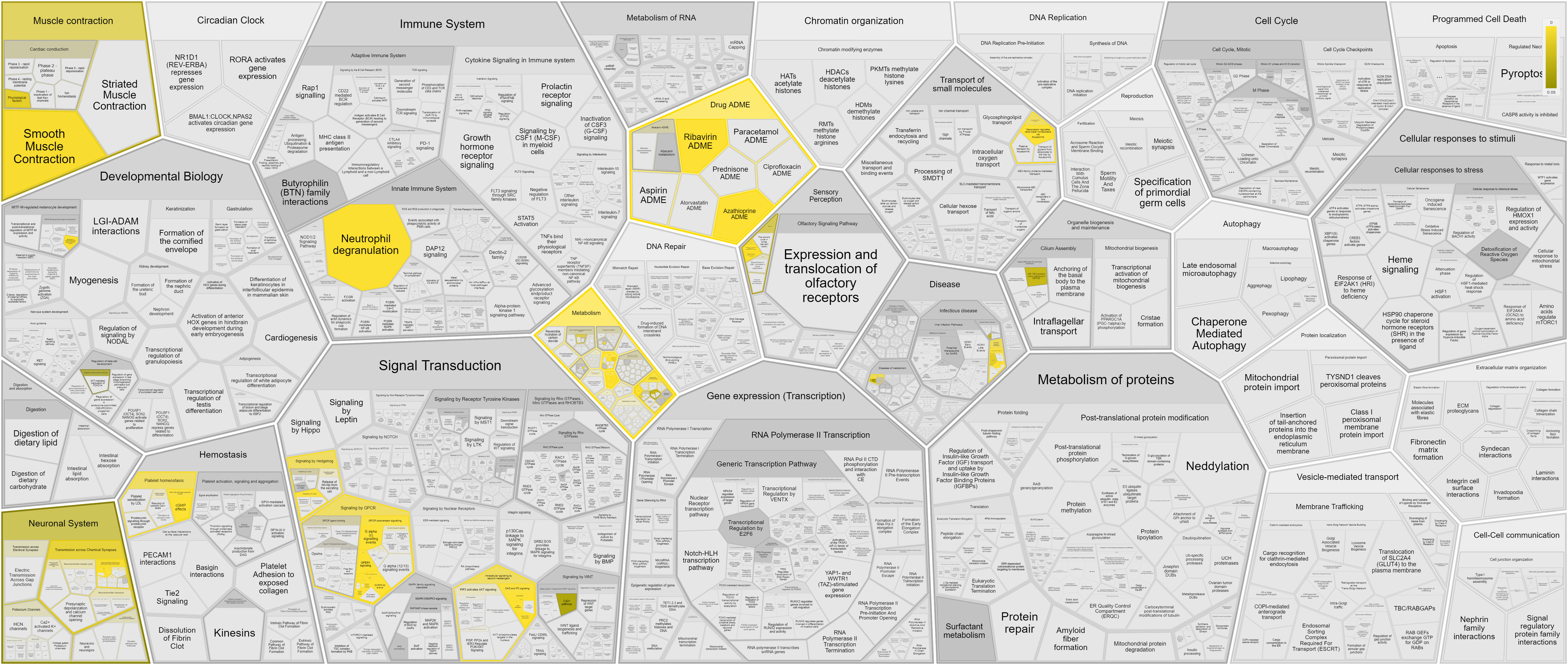
Вывод: некоторые белки не очень показательны для анализа функционального обогащения, т.к. принимают участие в самых разных процессах. Разные программы могут относить такие белки к разным категориям. Нужно смотреть на пути, которые представлены наиболее полно, в том числе белками с узкими функциями. Так в данном случае набор белков отвечает синтезу нуклеотидов, а не мышечным сокращениям.