Задание 1
Для начала был получен список оперонов бактерии Kocuria rhizophila ATCC с помощью программы Operon-mapper. У меня это уже давно было сделано для лабы, что приятно. Для того, чтобы вытащить все промоторы, я использовала скрипт на питоне, который сама написала. Код можно посмотреть по ссылке. Далее я отобрала опероны для получения материала обучения МЕМЕ. Они находятся в этом файле. Участки перед оперонами находятся в файле в формете FASTA. Также я получила последовательности длиной 150 нуклеотидов изнутри каждого оперона, чтобы использовать их в качестве негативного контроля.
Далее я запустила MEME со следующими параметрами:
meme selected_promotorseqs.fa -dna -nmotifs 3eqs.fa
Были найдены 3 мотива:
GTTBWWCGTSW E-value = 4.5e-001
WCVCGSKCAYCGH E-value = 2.3e+000
ATCACCACCCHSTCCGGCMCSGWCACCAC E-value = 6.4e+001
LOGO полученных мотивов показаны на рисунке 1.
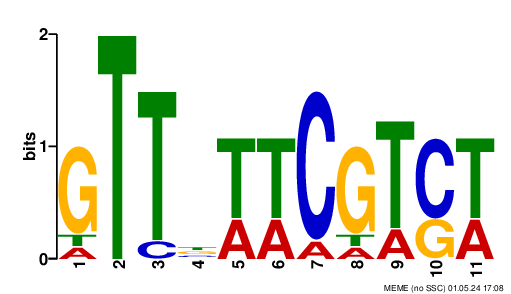

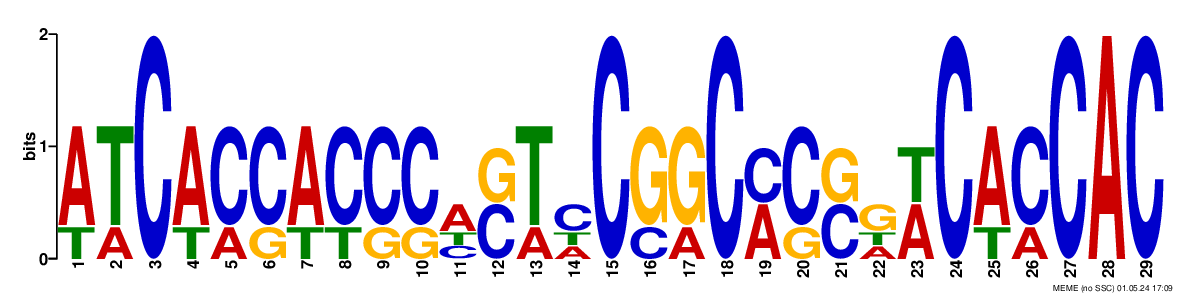
Рисунок 1. Мотивы, выданные МЕМЕ. Слева-направо: GTTBWWCGTSW, WCVCGSKCAYCGH, ATCACCACCCHSTCCGGCMCSGWCACCAC.
Для дальнейшей работы был взят первый мотив, т.к. у других находок слишком больше E-value. Хотя оно и у первой достаточно немаленькое(
Далее была запущена программа FIMO:
fimo --norc meme_out/meme.txt promotorseqs.fa
Результаты представлены в таблицах 1 и 2. Для последовательностей перед оперонами было найдено всего лишь совпадений. При этом у всех, кроме первой, очень большое q-value. То есть, хотя вероятность случайно получить одну такую находку маленькая (это отражает р-value), если учесть, что последовательностей очень много, вероятность значительно возрастает и находки уже нельзя считать значимыми. Для последовательностей, где заведомо нет промоторов, было получено 8 находок. У них у всех огромное q-value. То есть это немного лучше, чем в последовательностях, где должны быть промоторы, но вообще это полный провал.
Таблица 1. Находки мотива 1 в последовательностях промоторных областей.
| motif_id | motif_alt_id | sequence_name | start | stop | strand | score | p-value | q-value | matched_sequence |
| GTTBWWCGTSW | MEME-1 | promotor34 | 73 | 83 | + | 16.6847 | 2.16e-07 | 0.0401 | GTTGTTCGTCT |
| GTTBWWCGTSW | MEME-1 | promotor305 | 87 | 97 | + | 15.4234 | 1.84e-06 | 0.171 | GTTTTTCGTGT |
| GTTBWWCGTSW | MEME-1 | promotor253 | 8 | 18 | + | 14.3333 | 7.36e-06 | 0.456 | GTTTAACGTCT |
| GTTBWWCGTSW | MEME-1 | promotor308 | 40 | 50 | + | 13.4955 | 1.46e-05 | 0.677 | GTTGAACGTCA |
| GTTBWWCGTSW | MEME-1 | promotor1269 | 56 | 66 | + | 12.3784 | 3.56e-05 | 1 | GTCCATCGTCT |
| GTTBWWCGTSW | MEME-1 | promotor310 | 128 | 138 | + | 12.1712 | 4.03e-05 | 1 | GTTCTTCGAGA |
| GTTBWWCGTSW | MEME-1 | promotor361 | 63 | 73 | + | 12.1081 | 4.23e-05 | 1 | TTTCTTCGTGT |
| GTTBWWCGTSW | MEME-1 | promotor473 | 52 | 62 | + | 12.0811 | 4.49e-05 | 1 | GTTCTACTTCT |
| GTTBWWCGTSW | MEME-1 | promotor661 | 16 | 26 | + | 11.2883 | 7.42e-05 | 1 | ATTTTTCGTGA |
| GTTBWWCGTSW | MEME-1 | promotor552 | 89 | 99 | + | 10.8198 | 9.54e-05 | 1 | GTCGATCGTGA |
| motif_id | motif_alt_id | sequence_name | start | stop | strand | score | p-value | q-value | matched_sequence | |
| GTTBWWCGTSW | MEME-1 | promotor214 | 137 | 147 | + | 15.0811 | 3.52e-06 | 0.655 | GTTCTTCGTCA | |
| GTTBWWCGTSW | MEME-1 | promotor642 | 113 | 123 | + | 13.4955 | 1.46e-05 | 0.946 | GTTGAACGTCA | |
| GTTBWWCGTSW | MEME-1 | promotor770 | 47 | 57 | + | 12.9189 | 2.16e-05 | 0.946 | GTCGTACGTCT | |
| GTTBWWCGTSW | MEME-1 | promotor1083 | 44 | 54 | + | 12.6216 | 2.81e-05 | 0.946 | GTTGATCTTCT | |
| GTTBWWCGTSW | MEME-1 | promotor322 | 128 | 138 | + | 12.6216 | 2.81e-05 | 0.946 | GTTGATCTTCT | |
| GTTBWWCGTSW | MEME-1 | promotor907 | 93 | 103 | + | 12.4865 | 3.12e-05 | 0.946 | GTTTTTCGAGA | |
| GTTBWWCGTSW | MEME-1 | promotor609 | 32 | 42 | + | 12.3784 | 3.56e-05 | 0.946 | GTCCATCGTCT | |
| GTTBWWCGTSW | MEME-1 | promotor717 | 56 | 66 | + | 12.1081 | 4.23e-05 | 0.985 | GTTCTTCTTGT |
Мне кажется, такие плохие результаты получились потому, что МЕМЕ изначально построила плохой мотив. Видимо, это связано с тем, что я выбрала неподходящие опероны для обучения. Перед этими оперонами могут быть промоторы, которые узнают разные сигма-факторы, а не сигма-фактор домашнего хозяйства. Наверное, это особенность моей бактерии. Ну либо я что-то напутала в коде(