Цель данного занятия ознакомится с возможностями гомологичного моделирования комплекса белка с лигандом. В этом занятии был использован пакет Modeller. Работа была проведена с белком лизоцимом LYS_HYACE, используя известную структуру лизоцима форели (PDB:1lmp) как образец.
Загрузили модуль
import sys
sys.path.append('/usr/lib/modeller9v7/modlib/')
sys.path.append('/usr/lib/modeller9v7/lib/x86_64-intel8/python2.5/')
import modeller
import _modeller
import modeller.automodel
Задали некоторые параметры
env=modeller.environ()
env.io.hetatm=True
Скачали белок-образец и последовательность
## известная структура лизоцима форели
! wget http://www.pdb.org/pdb/files/1lmp.pdb
## выбранная последовательность
! wget http://www.uniprot.org/uniprot/P05105.fasta
Создали объект выравнивание, добавили последовательность и структуру
alignm=modeller.alignment(env)
alignm.append(file='P05105.fasta', align_codes='all',alignment_format='FASTA')
## создадим модель
mdl = modeller.model(env, file='1lmp.pdb', model_segment=('FIRST:'+'A', 'LAST:'+'A'))
## и добавим в выравнивание
alignm.append_model(mdl, atom_files='1lmp.pdb', align_codes='1lmp')
## есть смысл поправить идентификаторы
alignm[0].code = 'LYS_HYACE'
alignm.salign()
alignm.write(file='all_in_one.ali', alignment_format='PIR')
## Выбираем объект для моделирования
s = alignm[0]
pdb = alignm[1]
print s.code, pdb.code
## Создаем объект automodel
a = modeller.automodel.automodel(env, alnfile='all_in_one.ali', knowns= pdb.code , sequence = s.code )
a.name='mod'+s.code
a.starting_model = 1
a.ending_model = 2
a.make()
Оптимизированная структура выбранного белка находится в этом файле, неоптимизированная - в этом.
# визуализация результата
import nglview
import ipywidgets
w1 = nglview.show_structure_file('LYS_HYACE.B99990001.pdb')
w1
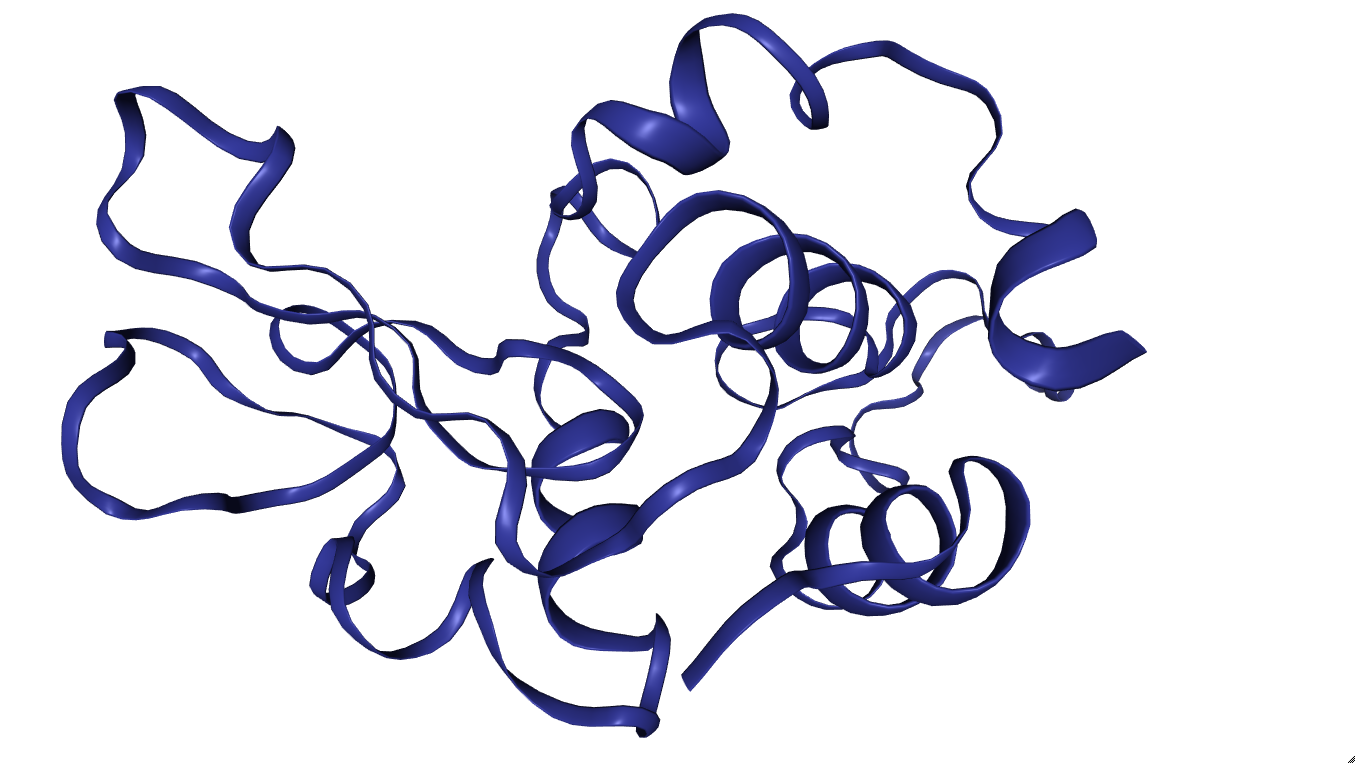
Лиганд был полностью проигнорирован. Чтобы это исправить нужно к последовательности нашего лизоцима добавить "...", что позволит считать три дополнительных остатка лиганда, которые есть в структуре лизоцима форели.
string=""
for i in alignm[0].residues:
string=string+i.code
string=string+"..."
out=open("With.fasta","w")
out.write(">WithLigand\n"+string)
out.close()
alignm.append(file='With.fasta', align_codes='all',alignment_format='FASTA')
alignm.salign()
alignm.write(file='all_in_one(2).ali', alignment_format='PIR')
## Выбираем объект для моделирования
s = alignm[2]
pdb = alignm[1]
print s.code, pdb.code
## Создаем объект automodel
a = modeller.automodel.automodel(env, alnfile='all_in_one(2).ali', knowns= pdb.code , sequence = s.code )
a.name='mod'+s.code
a.starting_model = 1
a.ending_model = 2
a.make()
import nglview
import ipywidgets
w1 = nglview.show_structure_file('WithLigand.B99990002.pdb')
w1
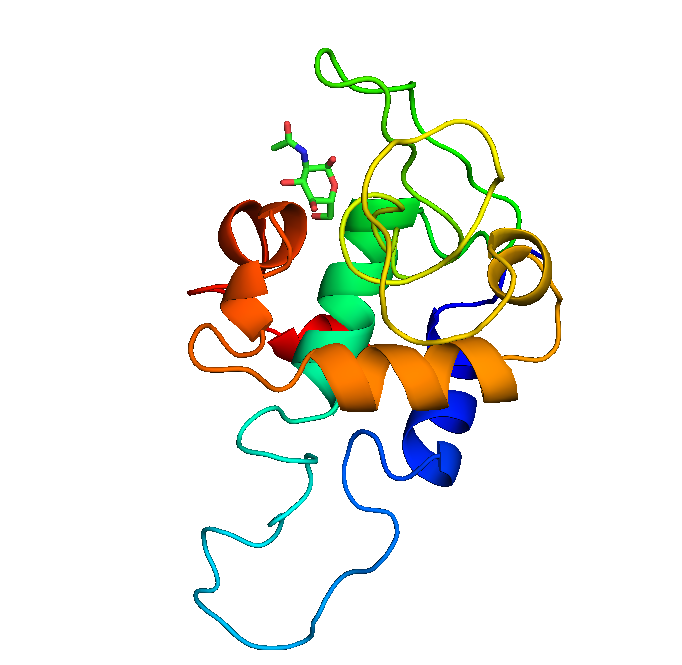
В итоге получили структуру, в которой отчётливо виден лиганд.