Докинг низкомолекулярных лигандов в структуру белка
Цель данного занятия ознакомится с возможностями докинга низкомолекулярного лиганда в структуру белка. В этом занятии был использован пакет Autodock Vina и oddt. Надо определить место докинга, удалить лигнад, запустить докинг и провести анализ.
Подключение модулей
import numpy as np
import copy
# Отображение структур
import IPython.display
import ipywidgets
from IPython.display import display,display_svg,SVG,Image
# Open Drug Discovery Toolkit
import oddt
import oddt.docking
import oddt.interactions
# Органика
from rdkit.Chem import Draw
from rdkit.Chem.Draw import IPythonConsole
import pmx # Модуль для манипулирования pdb
Подготовка белка
pdb=pmx.Model('WithLigand.B99990001.pdb')
for r in pdb.residues[135:]:
print r #посмотрим остатки
# создание объектов белок и лиганда
newpdb = pdb.copy()
lig = pdb.copy()
del newpdb.residues[139:]
del lig.residues[:139]
def average_in_list(lst):
return reduce(lambda x, y: x+y, lst)/len(lst)
x=[]
y=[]
z=[]
for a in lig.atoms:
x.append(a.x[0])
y.append(a.x[1])
z.append(a.x[2])
cen_x=average_in_list(x)
cen_y=average_in_list(y)
cen_z=average_in_list(z)
# найдите геометрический центр лиганда
newpdb.writePDB("hyace.pdb")
Подготовка белка для докинга
prot = oddt.toolkit.readfile('pdb','hyace.pdb').next()
prot.OBMol.AddPolarHydrogens()
prot.OBMol.AutomaticPartialCharge()
print 'is it the first mol in 1lmp is protein?',prot.protein,':) and MW of this mol is:', prot.molwt
smiles = ['CC(=O)N[C@H]1[C@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O', 'OC(=O)N[C@H]1[C@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O',
'[NH3+]C(=O)N[C@H]1[C@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O','C(=O)N[C@H]1[C@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O',
'C1=CC=CC=C1C(=O)N[C@H]1[C@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O',
'[O-]C(=O)C(=O)N[C@H]1[C@H](O)O[C@H](CO)[C@@H](O)[C@@H]1O']
mols= []
images =[]
for s in smiles:
m = oddt.toolkit.readstring('smi', s)
if not m.OBMol.Has3D():
m.make3D(forcefield='mmff94', steps=150)
m.removeh()
m.OBMol.AddPolarHydrogens()
mols.append(m)
###with print m.OBMol.Has3D() was found that:
### deep copy needed to keep 3D , write svg make mols flat
images.append((SVG(copy.deepcopy(m).write('svg'))))
display_svg(*images)
Докинг
#create docking object
dock_obj= oddt.docking.AutodockVina.autodock_vina(
protein=prot,size=(20,20,20),center=(cen_x, cen_y, cen_z),
executable='/usr/bin/vina',autocleanup=True, num_modes=20)
print dock_obj.tmp_dir
print " ".join(dock_obj.params) # Опишите выдачу
/tmp/autodock_vina_q6iT_Q --center_x 40.1232095238 --center_y 35.7878415584 --center_z 21.0768008658 --size_x 20 --size_y 20 --size_z 20 --cpu 1 --exhaustiveness 8 --num_modes 20 --energy_range 3
res = dock_obj.dock(mols,prot)
for i,r in enumerate(res):
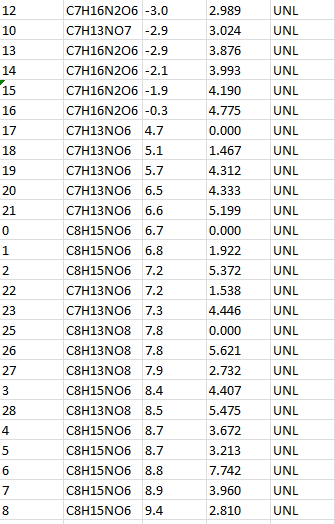
print "%4d%10s%8s%8s%8s" %(i,r.formula, r.data['vina_affinity'], r.data['vina_rmsd_ub'], r.residues[0].name)
0 C6H6O -3.0 0.000 UNL 1 C6H6O -3.0 1.829 UNL 2 C6H6O -3.0 4.389 UNL 3 C6H6O -2.8 3.005 UNL 4 C6H6O -2.7 3.747 UNL 5 C6H6O -2.7 2.933 UNL 6 C6H6O -2.7 2.755 UNL 7 C6H6O -2.6 3.665 UNL 8 C6H6O -2.6 4.117 UNL 9 C6H6O2 -3.0 0.000 UNL 10 C6H6O2 -3.0 3.656 UNL 11 C6H6O2 -3.0 3.231 UNL 12 C6H6O2 -3.0 4.154 UNL 13 C6H6O2 -2.9 4.191 UNL 14 C6H6O2 -2.9 4.397 UNL 15 C6H6O2 -2.7 2.282 UNL 16 C6H6O2 -2.7 3.675 UNL 17 C6H6O2 -2.1 2.743 UNL 18 C12H10O2 -0.1 0.000 UNL 19 C12H10O2 -0.0 2.188 UNL 20 C12H10O2 2.5 5.750 UNL 21 C12H10O2 2.8 5.403 UNL
for i,r in enumerate(res):
hbs = oddt.interactions.hbonds(prot,r)
stack= oddt.interactions.pi_stacking(prot,r)
phob = oddt.interactions.hydrophobic_contacts(prot,r)
print "%8s%8s%8s" % (len(hbs[1]), len(stack[1]), len(phob[1]))
1 0 8 1 0 8 2 0 6 1 0 9 2 1 9 0 0 7 0 0 7 1 1 6 0 0 5 1 0 6 1 0 6 1 0 6 2 0 5 2 0 3 2 0 4 2 0 2 2 1 8 0 0 4 2 4 38 2 4 37 3 2 37 2 2 36
Отсортировали от лучшего заместителя к худшему, результат представлен в таблице.
Из таблицы видно, что лучшим заместителем оказался C7H16N2O6, то есть тот лиганд, где метильная группа была заменена на NH3+. Также хочется отметить, что в соединении в любым типом лигандов вообще отсутствует стэкинг и очень мало гидрофобных взаимодействий.