Макромолекулярный докинг
In [7]:
%%bash
export LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:/home/preps/golovin/progs/lib
export PATH=${PATH}:/home/preps/golovin/progs/bin
In [ ]:
!echo 10 | pdb2gmx -f amylase.pdb -o amylase_h.pdb -p -water none -ignh
In [ ]:
!echo 10 | pdb2gmx -f camelid.pdb -o camelid_h.pdb -p -water none
In [ ]:
%%bash
export LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:/home/preps/golovin/progs/lib
export PATH=${PATH}:/home/preps/golovin/progs/bin
mark_sur amylase_h.pdb amylase_h_m.pdb
mark_sur помечает поверхностные основания в pdb файле.
In [ ]:
%%bash
export LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:/home/preps/golovin/progs/lib
export PATH=${PATH}:/home/preps/golovin/progs/bin
mark_sur camelid_h.pdb camelid_h_m.pdb
In [13]:
%%bash
export LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:/home/preps/golovin/progs/lib
export PATH=${PATH}:/home/preps/golovin/progs/bin
zdock
In [17]:
##докинг
%%bash
export LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:/home/preps/golovin/progs/lib
export PATH=${PATH}:/home/preps/golovin/progs/bin
zdock -R amylase_h_m.pdb -L camelid_h_m.pdb
In [6]:
%%bash
export LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:/home/preps/golovin/progs/lib
export PATH=${PATH}:/home/preps/golovin/progs/bin
zrank zdock.out.cp 1 2000
sort -n -k2 zdock.out.cp.zr.out | head
In [ ]:
%%bash
wget http://files.rcsb.org/download/1KXT.pdb
In [12]:
##получили файлы
%%bash
export LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:/home/preps/golovin/progs/lib
export PATH=${PATH}:/home/preps/golovin/progs/bin
ln -s /home/preps/golovin/progs/bin/create_lig
create.pl zdock.out
In [10]:
##считаем rmsd
import subprocess
for i in [1589, 1252, 610, 1436, 1952, 982, 1284, 851, 679, 1666]:
cmd1 = 'echo 1 1 | g_rms -s 1kxt_mol.pdb -f complex.%s.pdb -o %s.xvg' %(i,i)
subprocess.call(cmd1,shell=True)
cmd2 = 'sed -n -e 14p %s.xvg >> rmsd.txt' %(i)
subprocess.call(cmd2,shell=True)
In [11]:
!awk ' {print $2} ' rmsd.txt
Так как все значения RMSD оказались для всех моделей одинаковыми, то сравним в PyMol модель 610 и 1kxt.
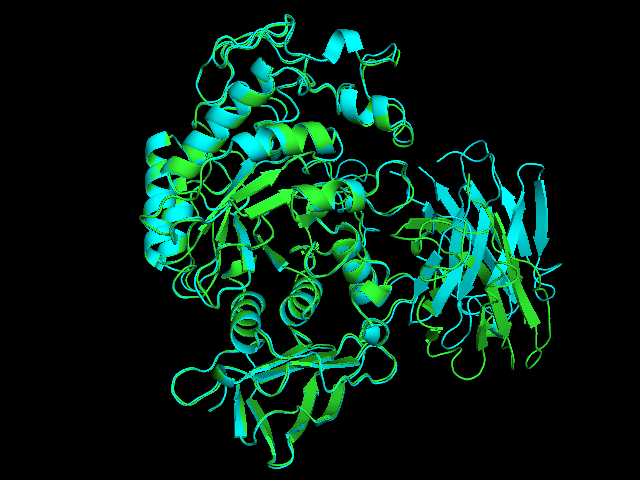
Видно, что сами амилазные части хорошо совпали, а лиганды совпали довольно плохо. Но можно заметить, что совпало место прикрепления лиганда к белку.