Пункт 1. Модуль pybel
In [3]:
import pybel
In [4]:
mol=pybel.readstring('smi','c1cc2cc3ccc(cc4ccc(cc5ccc(cc1n2)[nH]5)n4)[nH]3')
mol.addh()
mol.make3D(steps=10000)
In [6]:
mol.write(format='pdb',filename='pr5_1.pdb',overwrite=True)
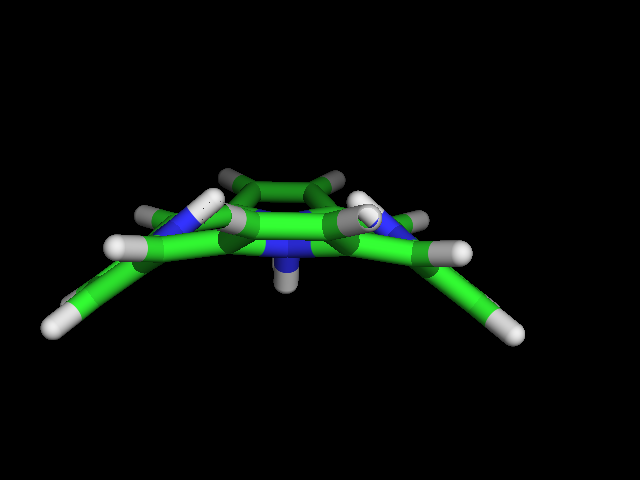
Порфирин получается неправильный: 4 водорода у центральных атомов кислорода и изогнутый. Правильный: плоский и 2 атома водорода. Попробовали другим способом.
Пункт 2. MOPAC способ PM6/АМ1
In [11]:
mop=mol.write(format='mopin',opt={'k':'PM6 CHARGE=%d' % mol.charge})
In [12]:
mop=mol.write(format='mopin',filename='my.mop',opt={'k':'PM6 CHARGE=%d' % mol.charge},overwrite=True)
В my.mop заряд молекулы равен нулю, поэтому перейдем в способу АМ1
In [14]:
mop_am1=mol.write(format='mopin',opt={'k':'AM1 CHARGE=%d' % mol.charge})
mop_am1=mol.write(format='mopin',opt={'k':'AM1 CHARGE=%d' % mol.charge},overwrite=True)
print mop_am1
In [15]:
%%bash
export MOPAC_LICENSE='/home/preps/golovin/progs/mopac/'
/home/preps/golovin/progs/mopac/MOPAC2016.exe my.mop
In [16]:
opt=pybel.readfile('mopout','my.out')
for i in opt:
print i
i.write(format='pdb',filename='my.pdb')
In [20]:
import subprocess
def mopac_pr5(smiles, method, prefix):
#стоило как-то иначе назвать функцию, но не хочется все переписывать
mol=pybel.readstring('smi',smiles)
mol.addh()
mol.make3D(steps=10000)
mop=mol.write(format='mopin',opt={'k': method+' CHARGE=%d' % mol.charge})
mop=mol.write(format='mopin',filename = prefix+'.mop',opt={'k': method+' CHARGE=%d' % mol.charge},overwrite=True)
mol.write(format='pdb',filename=prefix+'_before.pdb',overwrite=True)
cmd1 = " export MOPAC_LICENSE='/home/preps/golovin/progs/mopac/' "
cmd2 = '/home/preps/golovin/progs/mopac/MOPAC2016.exe '+prefix+'.mop'
subprocess.call(cmd1, shell=True)
subprocess.call(cmd2, shell=True)
opt=pybel.readfile('mopout',prefix+'.out')
for i in opt:
print i
i.write(format='pdb',filename=prefix+'.pdb',overwrite=True)
In [21]:
mopac_pr5('CC(=O)C','PM6', 'acetone')
mopac_pr5('C1=CC2=CC3=CC=C(N3)C=C4C=CC(=N4)C=C5C=CC(=N5)C=C1N2','PM6', 'por_pm6')
mopac_pr5('C1=CC2=CC3=CC=C(N3)C=C4C=CC(=N4)C=C5C=CC(=N5)C=C1N2','AM1', 'por_am1')
Получившиеся структуры:
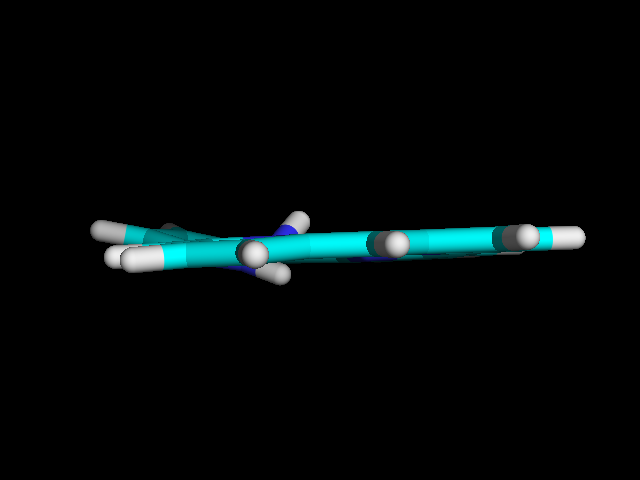

Из рисунок можно заметить, что правильная структура получилась в способе РМ6.
Пункт 3. Возбужденные состояния порфирина и спектр поглощения молекулы
In [22]:
%%bash
cp my.mop my_spectr.mop
echo "" >> my_spectr.mop
echo "cis c.i.=4 meci oldgeo" >> my_spectr.mop
echo "some description" >> my_spectr.mop
In [1]:
%%bash
export MOPAC_LICENSE='/home/preps/golovin/progs/mopac/'
/home/preps/golovin/progs/mopac/MOPAC2016.exe my_spectr.mop
In [4]:
opt=pybel.readfile('mopout','my_spectr.out')
for i in opt:
print i
i.write(format='pdb',filename='my_spectr.pdb',overwrite=True)
На основании значений энергий для электронных переходов, приведенных в конце файла, рассчитали длину волн, при которых происходят эти переходы ( по формуле Е=h*v)
In [6]:
f = open('my_spectr.out', 'r')
lines = f.readlines()[-21:-13]
f.close()
energies = [float(line.split()[1]) for line in lines]
print energies, 'eV'
Получили из файла значения энергий, теперь рассчитаем соотвутствующие им длины волн и частоты
In [7]:
wavelengths = [1239.84193/e for e in energies]
frequencies = [e/4.135667516 for e in energies]
print 'frequncy,THz\t wavelength,nm\tenergy,eV'
for i in range(len(energies)):
print '%.4f \t %.4f \t %.4f' % (frequencies[i]*1000, wavelengths[i], energies[i])
In [ ]: