Для работы мне была выдана плазмида с индикатором CP015205, который соответствует плазмиде pR8C2 бактерии Rhodococcus sp. 008. Rhodococcus - род грамотрицательных аэробных, неподвижных и неспорообразующих бактерий. Некоторые представители этого рода патогенны, однако большинство безвредны.

Рис. 1. Rhodococcus sp.
Плазмида pR8C2 - кольцевая и состоит из 65393 пар оснований. По данным GenBank, на ней находится 6916 генов, из которых 6845 кодируют белки, а 71 ген соответствует некодирующим РНК. Кроме того, плазмида включает 92 псевдогена.
Сначала я скачала последовательность плазмиды в двух форматах (fasta и gff) при помощи команды seqret пакета EMBOSS. Синтаксис этого действия таков: seqret embl:CP015205 fasta::cp015205.fasta и seqret embl:CP015205 gff::cp015205.gff -feature соответственно. Особенности (features), которые понадобятся для дальнейшей работы, были записаны с помощью -features.
С помощью Prodigal были предсказаны гены в выданной плазмиде (использовалась команда prodigal.win.exe -i cp015205.fasta -o out.fasta. Получившийся файл.
Далее с помощью команд grep CDS cp015205.gff | cut -f 4,5,7 --output-delimiter='_'> genbank.out и grep '>' prodigal.fasta | cut -f 2,3,4 -d '_' > prodigal.out
в отдельные файлы были записаны координаты генов (начало, конец, ориентация, разделенные символом '_'). Получившиеся файлы: и genbank.out.Для дальнейшей обработки данных был написан скрипт на Python script.py. Скрип работает следующим образом:
На вход подаются три файла: полученные по указанному выше способу файлы GenBank и Prodigal, а также вспомогательный файл result.out, куда для удобства будут записаны все полученные результаты. После получения файлов создаются два списка genbank и prodigal, содержащих строки с координатами из исходных файлов (координаты разделены символом '_'). Далее для каждого из этих списков создаются два списка, содержащих координаты С- и N-концов соответственно. Для этого в цикле из каждой строки делается список (разделитель '_') и проверяется ориентация гена ('+' или '-' как последний элемент полученного из строки списка). В зависимости от этого в качестве координаты N-конца (С-конца) в случае '+' берется первая (вторая) координата, а в случае '-' — вторая (первая). Итак, мы имеем 4 списка с координатами C- и N-концов белков, предсказанных GenBank и Prodigal. После этого для каждой координаты N-конца в n_genbank ищется такая же координата в n_prodigal. Если таковая обнаруживается, то проверяется координата С-конца и подсчитывается число генов, у которых белки совпадают или совпадают только N-концы. Далее проводится аналогичный поиск по С-концам (для тех случаев, когда N-концы не совпадают), в зависимости от результатов поиска подсчитывается число генов, у белков которых совпадают только С-концы, а также генов, у белков которых различаются как N-, так и С-концы. Все результаты записываются в файл result.out.
Итак, результаты работы скрипта таковы:
1. GenBank аннотирует 73 гена, а Prodigal - 82.
2. Из этих генов идентичны 42 (51,22 %).
3. Аннотация только N-конца белка не совпадает с аннотацией Prodigal у белков 27 (32,93 %) генов.
4. Аннотация только С-конца белка не совпадает с аннотацией Prodigal у белков 2 (2,44 %) генов.
5. Prodigal не предсказывает 7 генов, аннотированных в GenBank, а в GenBank не аннотировано 11 генов, предсказанных Prodigal.
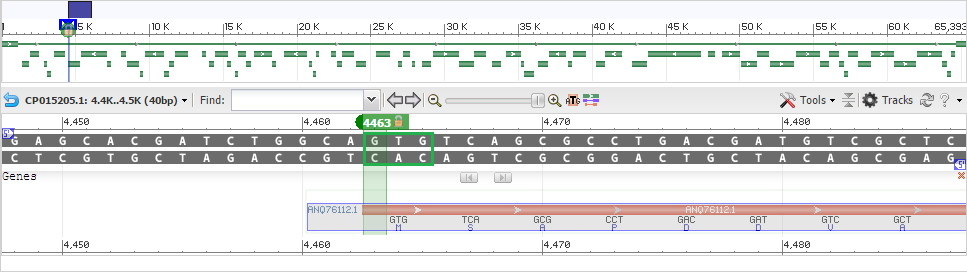
Пример гена, для которого по-разному был предсказан N-конец белка. Ниже приведён пример гена, для которого и Prodigal, и Ganbank определили С-конец одинаково (31957), но N-конец в GenBank начинается с позиции 31001, а Prodigal поместил его в 30947. (на рисунках ниже обведён триплет TAC, комплементарный старт-кодону AUG мРНК)
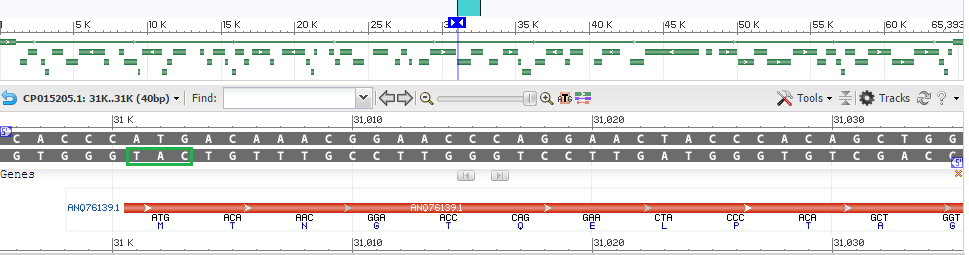
Рис 2. Начало гена по данным GenBank
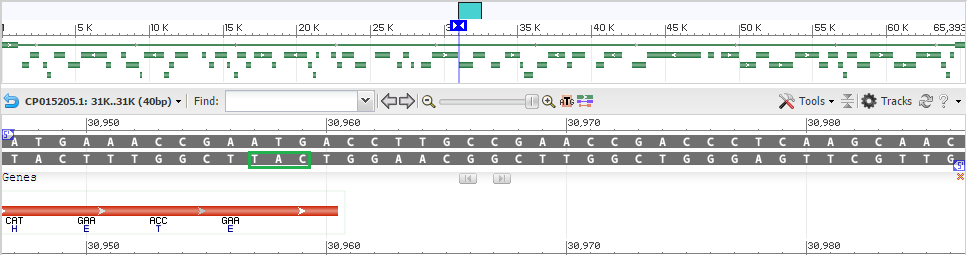
Рис 3. Начало гена по данным Prodigal
Как можно видеть, у GenBank и Prodigal для этого гена по-разному расположены рамки считывания, поэтому начало гена по Prodigal находится в конце гена, аннотированного в GenBank. По-видимому, в предыдущем гене предсказания GenBank и Prodigal также различаются, что может быть обусловлено разнообразными причинами: ошибкой программ, использованием нестандартных кодонов и т. п.
Ещё один пример гена, для которого по-разному был предсказан N-конец белка. И Genbank, и Prodigal определили положение С-конца как 5293, а вот

Рис 4. Начало гена по данным GenBank
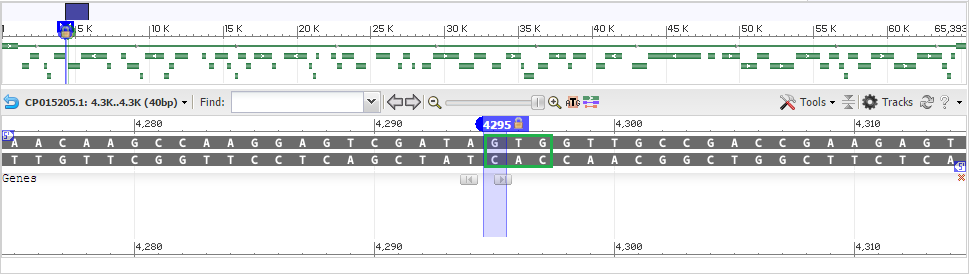
Рис 5. Начало гена по данным Prodigal
Как можно видеть, в обоих случаях обнаруживаются нестандартные старт-кодоны, и, возможно, путаница со старт-кодонами и лежит в основе противоречий данных Genbank и Prodigal.