Мне достался контиг мха фискомитрелла раскрытая (Physcomitrella patens). Таксономия организма (последовательно перечислены таксоны в порядке уменьшения ранга): Eukaryota; Viridiplantae; Streptophyta; Embryophyta; Bryophyta; Bryophytina; Bryopsida; Funariidae; Funariales; Funariaceae; Physcomitrella.

Рис. 1. Фискомитрелла раскрытая (Physcomitrella patens)
Предложенный контиг имеет идентификатор NW_001865543 (скачать контиг). Контиг имеет длину 564169 пар оснований и содержит 43 белок-кодирующие последовательности, при этом всего он содержит 61 ген.
К сожалению, примеров альтернативного сплайсинга мне найти не удалось. Вот один из генов (PHYPADRAFT_108581) и его геномное окружение.
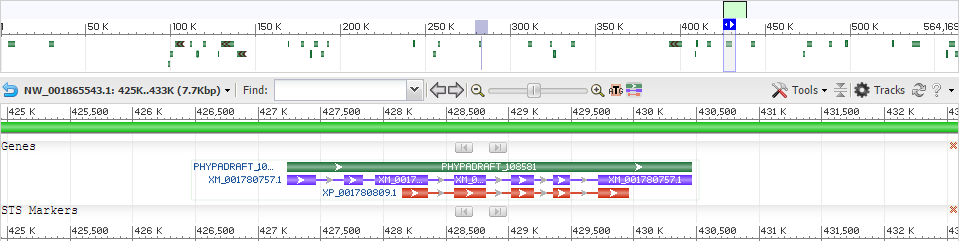
Рис. 2. Геномное окружение и мРНК гена PHYPADRAFT_108581
Как видно на рисунке, у этого гена имеется одна мРНК и один соответствующей ей белок.
Вот другой контиг, где есть альтернативный сплайсинг. Как видно, одному гену соответствует несколько мРНК и белков.
Предсказание предлагалось выполнить с помощью онлайн-ресурса AUGUSTUS. Для выполнения предсказания ресурс запрашивает не только нужную последовательность (контиг в моём случае), но и вид, который будет использоваться для построения модели. Вид должен быть как можно ближе к тому виду, последовательность которого исследуется. Поскольку в моём случае контиг принадлежит геному мха, я сразу отбросила все организмы, которые предлагает Augustus, кроме растений. У AUGUSTUS их всего 4 вида: арабидопсис, помидор, пшеница и кукуруза - только сосудистые растения, мхов нет. Поскольку считается, что однодольные (в том числе злаки) - эволюционно более молодая группа организмов, то злаки я сразу отбросила, и попробовала запустить предсказание с арабидопсисом, чтобы посмотреть, получится ли. Получилось :) Проблему можно было бы решить с помощью BLAST, в частности, blastx: найти виды на основании белков, считанных с данного в задании контига. Но у меня BLAST почему-то упорно отказывался запускаться: мучился полчаса и в итоге выдал ошибку, и так повторялось всё время. Поэтому я выбрала организм на основании чисто биологических соображений. Все остальные настройки я оставила по умолчанию.
Итак, выдача AUGUSTUS состояла в архиве predictions.tar.gz, который содержит следующие файлы:
1) augustus.aa с аминокислотными последовательностями белков (в формате fasta), соответствующих предсказанным генам;
2) augustus.cdsexons с нуклеотидными последовательностями предсказанных экзонов в формате fasta;
3) augustus.codingseq с кодирующими нуклеотидными последовательностями предсказанных генов целиком в формате fasta;
4) augustus.gbrowse с информацией (координаты в геноме, прямая или обратная цепь) о структурных элементах пре-мРНК (кодирующая последовательность, интрон, старт- и стоп-кодоны, инициаторная и терминирующая последовательности), предшествующей каждому из предсказанных генов (предсказание геномного браузера GBrowse);
5) augustus.gtf - предсказание генов в формате .gtf;
6) augustus.gff - файл со всей вышеперечисленной информацией вместе (формат .gff).
AUGUSTUS нашёл 115 белок-кодирующих генов против 43, предсказанных базой, так что о точном предсказании здесь говорить не приходится. Ген, изображение которого приведено выше, также опознан не был. Я полагаю, такая низкая точность поиска связана с тем, что выбор организмов-моделей в AUGUSTUS очень ограничен, и арабидопсис эволюционно слишком далёк от мха, чтобы служить моделью для предсказания генов мха. Возможно, контиг был не очень удачным и содержал в основном характерные для мхов последовательности, а не общие для всех растений гены (например, гены белков, участвующих в фотосинтезе и сопряжённых процессах).