
В данном практикуме предлагалось осуществить сборку de novo генома бактерии Buchnera aphidicola. Buchnera aphidicola - внутриклеточный эндосимбионт тлей (в частности, гороховой тли).

Сначала архив с чтениями, полученными по технологии Illumina, был скачан со страницы проекта по адресу http://www.ebi.ac.uk/ena/data/view/SRR4240383. Полученный архив был распакован программой gunzip: gzip -d SRR4240383.fastq.gz.
Далее была проведена подготовка скачанных чтений. Сначала требовалось удалить возможные остатки адаптеров, для чего был создан файл adapters.fasta. Далее очистка от адаптеров проводилась с помощью команды Trimmmatic, запущенной как показано на рисунке:
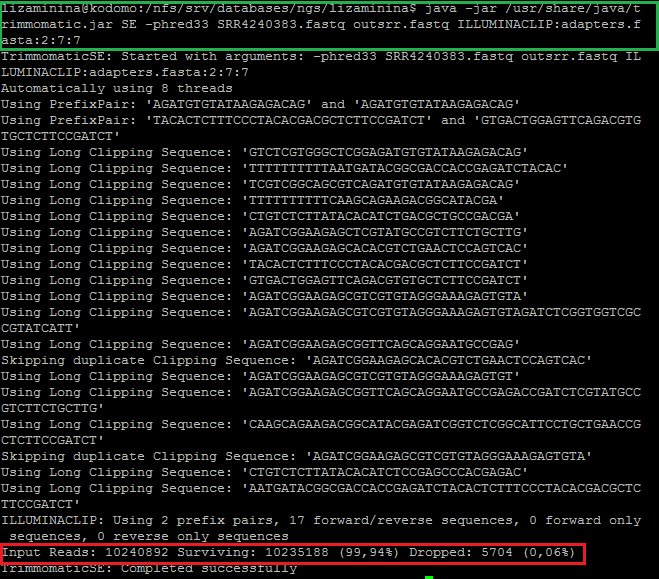
Зелёной рамкой выделен синтаксис программы, а красной - конечный отсев ридов. Можно видеть, что из 10240892 входных ридов 5704 чтения были отбракованы.
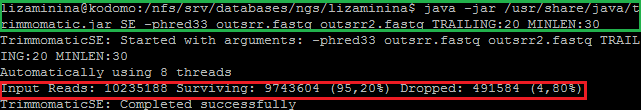
После этого были удалены плохие буквы с концов чтений, и были оставлены только чтения длиной не менее 30. Синтаксис команды выделен зелёной рамкой, а результат работы - красной рамкой. Видно, что из оставшихся в результате первой обработки Trimmomatic 10235188 чтений было отброшено 491584, а размер файла изменился с 1033 Мб до 973 Мб.
Для подготовки k-меров длины 29 была запущена программа velveth, для которой я указала директорию Assem, в которую она будет складывать результаты своей работы, длину k-мера (31), опции -short (короткие одноконцевые чтения) и -fastq (формат входного файла). Синтаксис программы обведён красной рамкой на рисунке ниже. Также красной рамкой обведено количество ридов, с которыми столкнулась программа в очищенным с помощью Trimmomatic файле.

Далее на основе k-меров была осуществлена сборка контигов с помощью программы velvetg. Я запускала её так: velvetg Assem, где Assem - директория, в которой находятся результаты работы velveth. Итоговая выдача velvetg приведена на картинке:

Как можно видеть, N50 = 302 (то есть контигами длиной не менее 302 п. н. можно покрыть 50% генома), а длина наибольшего контига равна 37582. Другие результаты работы программы:
* Три самых длинных контига имеют длину 37582 (номер 27), 27361 (номер 20) и 22184 (номер 122), им соответствует покрытие 40,5, 52,3 и 30,55.
* "Типичное" покрытие примерно составляет 30-50. Однако есть и сильно выбивающиеся контиги, причём контигов с очень малым покрытием больше, чем с очень большим покрытием. Самое большое покрытие у контига под номером 674 - 422, а одно из самых малых покрытий у контига 1380 - 3,4.
С последовательностями самых длинных контигов можно ознакомиться тут: 27, 20, 122.
Финальной частью работы было построение выравнивания трёх самых длинных контигов с хромосомой исследуемой бактерии (CP009253) с помощью megablast. Итак, для самого длинного контига megablast построил шесть выравниваний, самое длинное из которых соответствует координатам хромосомы бактерии: начало 236940 и конец 247596.
Для второго по величине контига (27361) было построено восемь выравниваний, самое длинное из которых соответствует координатам хромосомы бактерии 5935-1103. В целом, выравнивания для первых двух контигов довольно неплохие, в них нет протяжённых гэпов и совсем непохожих участков.
У третьего по величине контига (22184) и бактериальной хромосомы не было найдено никакого сходства, поэтому и выравнивания построено не было.
Наконец, требовалось построить аналогичные выравнивания для двух контигов с самым большим покрытием. В данном случае таковыми были контиги 104 (покрытие 328) и 674 (покрытие 422). Для контига 674 megablast ничего не смог построить, как и для контига 104. Меня очень смущают эти результаты, но я могу лишь предположить, что это связано с несовершенством сборщика, потому что megablast предназначен для выравнивания очень похожих последовательностей.