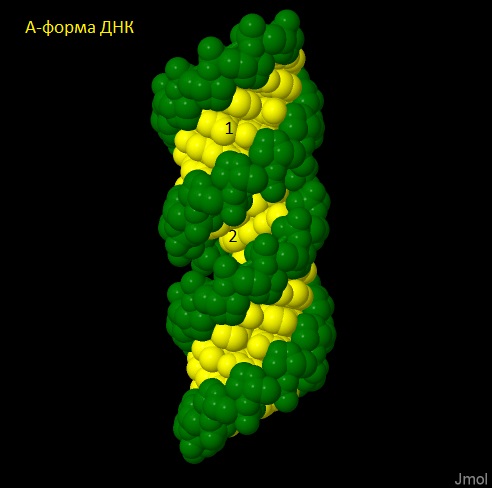
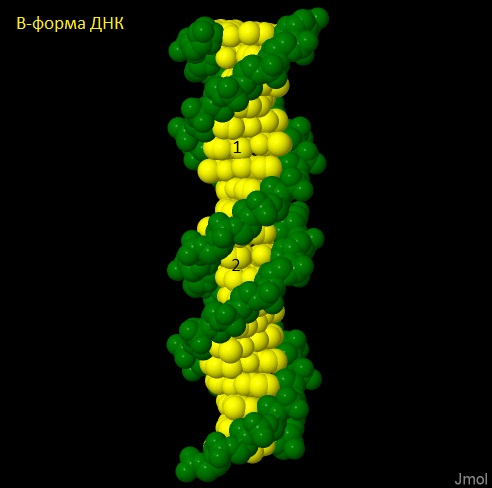
С помощью команды fiber пакета 3DNA были построены структуры ДНК с последовательностью, состояющую из 5 повторов gatc, в формах А и B.
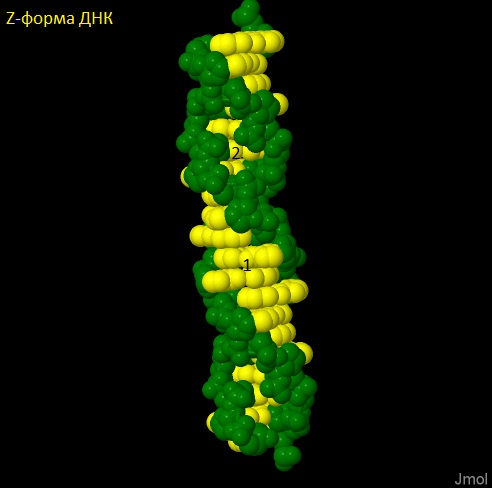
Z-форма ДНК может образовываться только в GC-повторах, и для построения структуры Z-ДНК было использовано 10 повторов GC. Полученные структуры
представлены ниже. Цифрой 1 отмечена большая бороздка, цифрой 2 - малая.
 |  |  |
Для выполнения задания 2 был использован 10-й аденин. Было изучено, какие атомы смотрят в сторону большой и малой бороздки в разных формах ДНК. На рисунке ниже представлен аденин, и красным цветом покрашены атомы, смотрящие в сторону большой бороздке в В-форме, а синим - в сторону малой, также в В-форме.
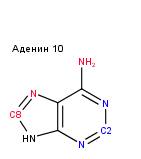
Ниже в таблице перечислено, какие атомы смотрят в сторону большой и малой бороздки в А-ДНК и В-ДНК. Z-ДНК состоит только из повторов GC и потому не содержит аденина. Используются имена атомов из соответствующих структур PDB.
| A-ДНК | B-ДНК | |
| Обращены в сторону большой бороздки | A10.N3, A10.C2, A10.N1, A10.C4 | A10.C8, A10.N7, A10.N6 |
| Обращены в сторону малой бороздки | A10.N6, A10.N7, A10.C8, A10.C5 | A10.N3, A10.C2, A10.N1 |
| Остальные атомы основания | A10.N9, A10.C6 | A10.N9, A10.C4, A10.C5, A10.C6 |
В таблице ниже сравниваются 3 формы ДНК (А-, В- и Z-ДНК). Два последних параметра были определены для атома фосфора C32.P.
| Параметр | A-ДНК | В-ДНК | Z-ДНК |
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28,03 | 33,75 | 43,5 |
| Количество оснований на виток | 11 | 10,5 | 12 |
| Ширина большой бороздки (Å) | 21,25 | 18,04 | 16,08 |
| Ширина малой бороздки (Å) | 7,98 | 11,69 | 10,67 |
С помощью Jmol измерялись торсионные углы выбранного нуклеотида (аденозин 10) и сравнивались с теоретическими значениями. Ниже представлено две таблицы: в первой измеренные торсионные углы, а во второй теоретические значения этих углов (в обоих случаях значения углов приведены в градусах).
| Форма ДНК | α | β | γ | δ | ε | ζ | χ |
| A | 64,1 | 174 | 41,7 | 79,1 | 163,3 | -75,1 | -157,2 |
| B | 85,9 | 136,3 | 31,2 | 143,3 | 105,8 | -44,7 | -98 |
| Форма ДНК | α | β | γ | δ | ε | ζ | χ |
| A | 62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| B | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
Как можно заметить, некоторые углы оказались очень близки к теоретическим, однако многие значения в той или иной мере расходятся с теоретическими. В целом, для большинства углов (кроме углов γ и ς) результат их сравнения между А и В формой (что где больше) оказался верным.
При помощи программ find_pair и analyze были изучены структуры тРНК (1u0b) и ДНК (1bdt). Были получены значения торсионных углов для каждого нуклеотида, с конкретными значениями можно ознакомиться в файлах 1u0b.out и 1bdt.out.
С помощью Excel были рассчитаны средние значения каждого из торсионных углов для тРНК и ДНК (в обоих случаев для расчётов бралась цепь I). Средние значения торсионных углов приведены в таблице:
| Молекула | α | β | γ | δ | ε | ζ | χ |
| тРНК | -54,26 | 38,75 | 65,29 | 84,31 | -143,84 | -77,49 | -136,50 |
| ДНК | -49,97 | 14,31 | 22,33 | 137,82 | -90,35 | -77,35 | -110,96 |
Самыми деформированными (то есть с наиболее выраженно отклоняющимися значениями торсионных углов) нуклеотидами в тРНК оказались U7, U8, G11, A15, A27, G30, а в ДНК G4, A6, C12, T13, T18.
С расчётами можно ознакомиться в файле.
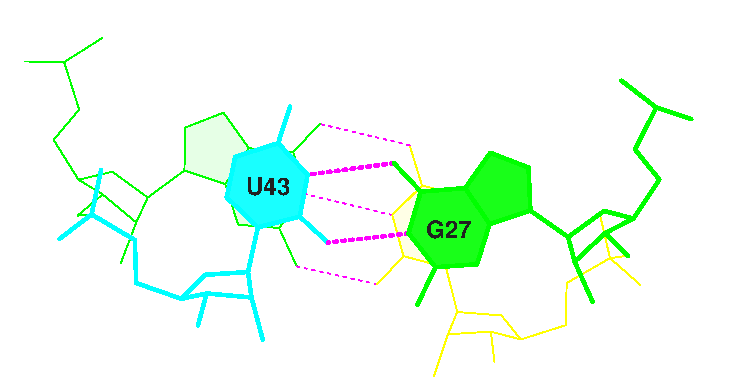
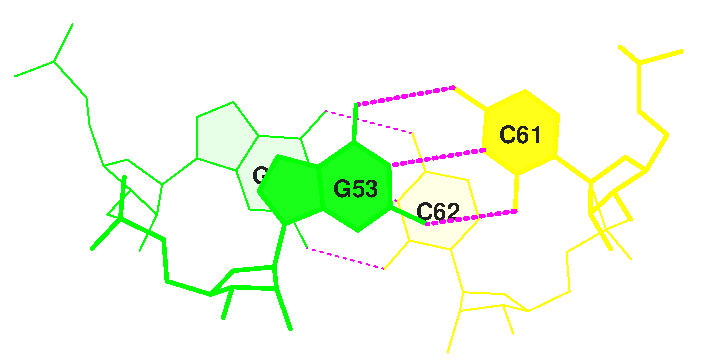
В полученном с помощью программы find_pair файле 1u0b.out содержится информация о водородных связях, поддерживающих структуру молекулы. В структуре молекулы имеется 4 стебля, как показано на рисунке ниже.
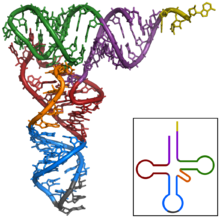
| Фиолетовый стебель | 1_:[..G]G-----C[..C]:..72 2_:[..G]G-----C[..C]:..71 3_:[..C]C-----G[..G]:..70 4_:[..G]G-----C[..C]:..69 5_:[..C]C-----G[..G]:..68 6_:[..G]G-----C[..C]:..67 7_:[..U]U-----A[..A]:..66 |
| Зелёный стебель | 49_:[..U]U-----A[..A]:..65 50_:[..C]C-----G[..G]:..64 51_:[..C]C-----G[..G]:..63 52_:[..G]G-----C[..C]:..62 53_:[..G]G-----C[..C]:..61 |
| Синий стебель | 38_:[..A]A-**--U[..U]:..32 39_:[..U]U-----A[..A]:..31 40_:[..C]C-----G[..G]:..30 41_:[..C]C-----G[..G]:..29 42_:[..G]G-----C[..C]:..28 43_:[..U]U-*---G[..G]:..27 44_:[..C]C-**--A[..A]:..26 |
| Красный стебель | 10_:[..A]A-----U[..U]:..25 11_:[..C]C-----G[..G]:..24 12_:[..A]A-----U[..U]:..23 |
Следующие водородные связи не принимают участия в образовании стеблей:
54_:[..U]U-**--A[..A]:..58
55_:[..U]U-**+-G[..G]:..18
36_:[..A]A-**--U[..U]:..33
13_:[..A]A-**--A[..A]:...9
14_:[..A]A-**--U[..U]:...8
15_:[..G]G-**+-G[..G]:..48
16_:[..C]C-**+-C[..C]:..59
19_:[..G]G-*---C[..C]:..56
73_:[..U]U-**+-C[..C]:..74
Как показала программа find_pair, в данной структуре тРНК имеется 12 не Уотсон-Криковских пар оснований, а именно пары (в скобках дан номер пары): U-A (13), U-G (14), A-U (15), A-U (16), U-G (21), C-A (22), A-A (26), A-U (27), G-G (28), C-C (29), G-C (30), U-C (31).