Мой белок - YP_526380.1 бактерии Saccharophagus degradans. Поиск по его последовательности в базе CDD дал следующие результаты:
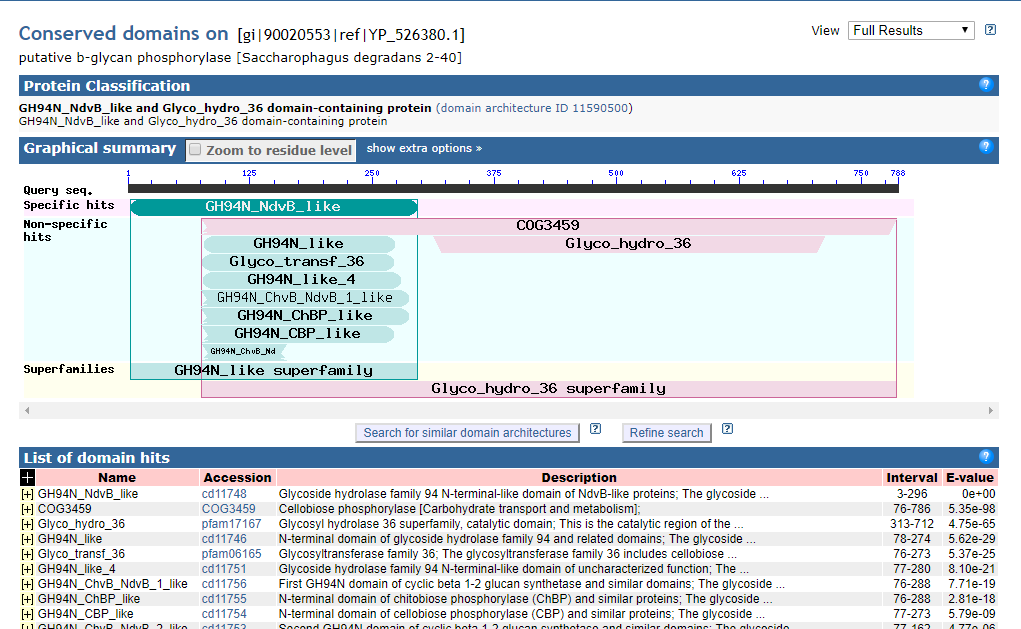
Как можно видеть, мой белок с высокой достоверностью (E-value 5.35e-98) относится к COG3459. В этот КОГ попадают остатки с 76 по 786 (при общей длине белка 788 аминокислотных остатка). COG3459 - Cellobiose phosphorylase [Carbohydrate transport and metabolism] (фосфорилаза целлобиозы; целлобиоза - это дисахарид). Функциональная группа - метаболизм и транспорт углеводов.
Далее с помощью программы COGNAT (параметры по умолчанию: neighborhood size = 9, occurrence threshold = 20 %) было визуализировано геномное окружение для данного COG'a, которое представлено ниже:

Цифрами обозначены:
1 - Hypothetical protein (Q21L46)
2 - Regulatory protein, TetR (Q21L47)
3 - Hypothetical protein (Q21L48)
4 - Cellobiose phosphorylase (Q21L49) - наш рабочий белок, поэтому он и выделен синим цветом.
далее идут гипотетические белки и самым последним идёт Regulatory protein, TetR (Q21L53.)
Геномное окружение не является консервативным, как видно на представленном ниже рисунке:

С помощью BLAST в базе GO был выполнен поиск белка, похожего на мой. Лучшие результаты представлены ниже:
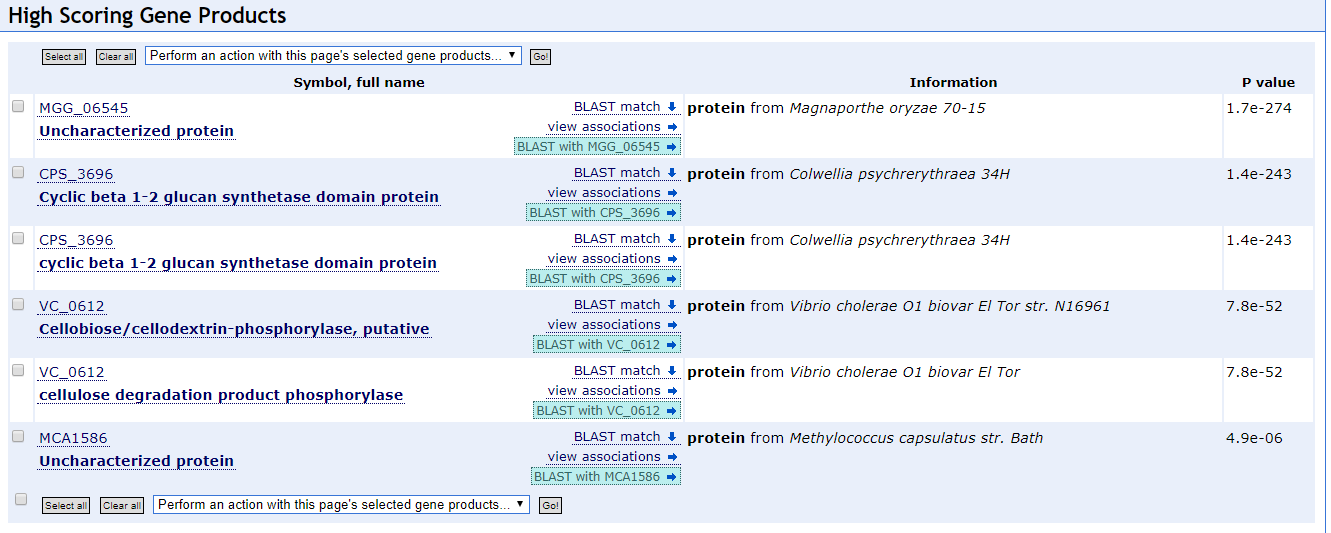
Как ни странно, находка с лучшим значением p-value относится к растениям. Естественно, это другой белок. Мне кажется, что целесообразней было бы рассматривать находку VC_0612: это тоже бактериальный белок, и функция у него близкая к моему белку.
Таблица 1. Термины GO, отнесённые к белку с идентификатором YP_526380.1
| Аспект | Идентификатор GO | Перевод названия термина | Код типа достоверности |
| carbohydrate catabolic | GO:0016052 | Катаболизм углеводов | ISS |
| phosphorylase activity | GO:0004645 | Фосфорилазная активность | ISS |
Таблица 2. Описание кодов достоверности, использованных в Таблице 1.
| Код типа достоверности | Расшифровка кода типа достоверности | Объяснение |
| ISS | Inferred from Sequence or structural Similarity | Используется при каком-либо анализе, включающем последовательности. |
| ISO | Inferred from Sequence Orthology | Используется, когда две последовательности предположительно ортологичны, т.е. имеют общего предка |
| ISA | Inferred from Sequence Alignment | Используется, когда основой анализа является выравнивание. |
| ISM | Inferred from Sequence Model | Используется, когда анализ включает работу с математической моделью. |
| IGC | Inferred from Genomic Context | Используется, когда известен геномный контекст гена. |
| IBA | Inferred from Biological aspect of Ancestor | Используется, когда вывод о свойствах потомка делается на основе свойств предполагаемого предка. |
| IBD | Inferred from Biological aspect of Descendant | Используется, когда вывод о свойствах предка делается на основании свойств потомка. |
| IKR | Inferred from Key Residues | Выводы делаются на основании отсутствия ключевых остатков. |
| IRD | Inferred from Rapid Divergence | Используется при быстрой дивергенции предковой последовательности. |
| RCA | Inferred from Reviewed Computational Analysis | Используется при каком-либо анализе, включающем последовательности. |
Сначала мы нашли кластер ортологичных групп, в состав которого входит мой белок. Далее мы исследовали геномное окружение гена, кодирующего наш белок, то есть выяснили, какие гены соседствуют с ним. Затем в базе GO (посвещённой генной онтологии) с использованием алгоритма BLAST было найдено несколько похожих белков, но сам белок найден не был. Код достоверности был при этом ISS, то есть был основан чисто на последовательностях. Таким образом, мы нашли в базе GO несколько близких белков, но САМ белок не нашли.