В данном задании нужно было найти гомологов белка CLPX E. coli у бактерий из первого практикума и построить филогенетическое дерево этих белков. Вначале я осуществила поиск гомологичных белков среди выбранных бактерий при помощи BLAST (pblast, база nr, настройки по умолчанию). У всех этих бактерий нашлись белки CLPX. Кроме того, оказалось, что у этих бактерий есть белки, близкие к CLPX: HSLU. Дерево, реконструированное в программе MEGA (метод Neighbor-Joining) , выглядит следующим образом:
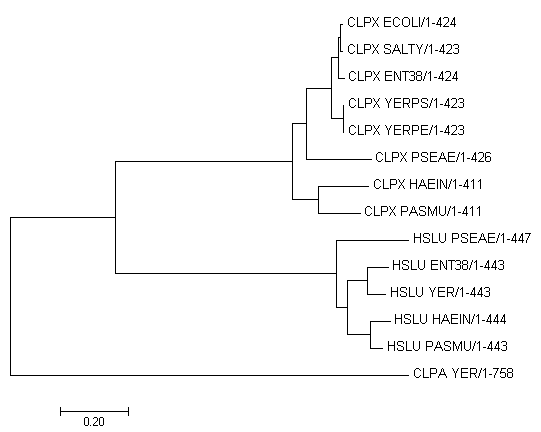
Мнемоника YER относится к всему роду ''Yersinia'' (BLAST нашёл эти белки в базе nr, озаглавленные MULTISPECIES, то есть относящиеся не к одному, а к нескольким видам). У бактерий рода ''Yersinia'' нашёлся ещё один гомолог - CLPA.
На данном дереве можно увидеть два события дупликаций гена: по-видимому, первая дупликация соответствует расхождению CLPA и общего предка CLPX и HSLU, а вторая дупликация привела к появлению CLPX и HSLU. В кладах CLPX и HSLU можно видеть несколько событий расхождения белков у разных видов. Все три белка обнаруживаются у ''Yersinia'', и CLPX, HSLU, CLPA являются паралогами у этих бактерий. Белки CLPX разных бактерий могут служить примером ортологов.
Примеры: CLPX_ECOLI, CLPX_PASMU и остальные белки CLPX - группа попарно ортологичных белков. HSLU_PASMU, HSLU_YER и остальные елки HSLU - вторая группа попарно ортологичных белков. CLPX_YERPS и CLPA_YER, CLPX_YERPS и HSLU_YER - пары паралогов.