Для выполнения данного практикума я выбрала домен Nucleoplasmin (PF03066) (страница домена в Pfam). Белки, содержащие этот домен, объединяются в нуклеоплазминовое семейство. В состав семейства входят три преимущественно ядрышковых белка: NPM1 (также известен как В23 и нуклеофозмин), NPM2 и NPM3. Белки семейства способны к олигомеризации: NPM1 и NPM2 формируют пентамеры, а NPM3 - димеры. Кроме того, возможно образование гетероолигомеров белков NPM1 и NPM3. Нуклеоплазминовый N-концевой домен необходим именно для олигомеризации. NPM1 задействован в самых разных клеточных процессах: биогенезе рибосом, апоптозе, сборке веретена деления, а его мутации выявляются в более чем половине случаев острого миелоидного лейкоза. NPM2 экспрессируется только в ооцитах, а о NPM3 почти ничего не известно.
Известно 16 доменных архитектур (512 последовательностей), включающих данный домен, они представлены на рисунке ниже:
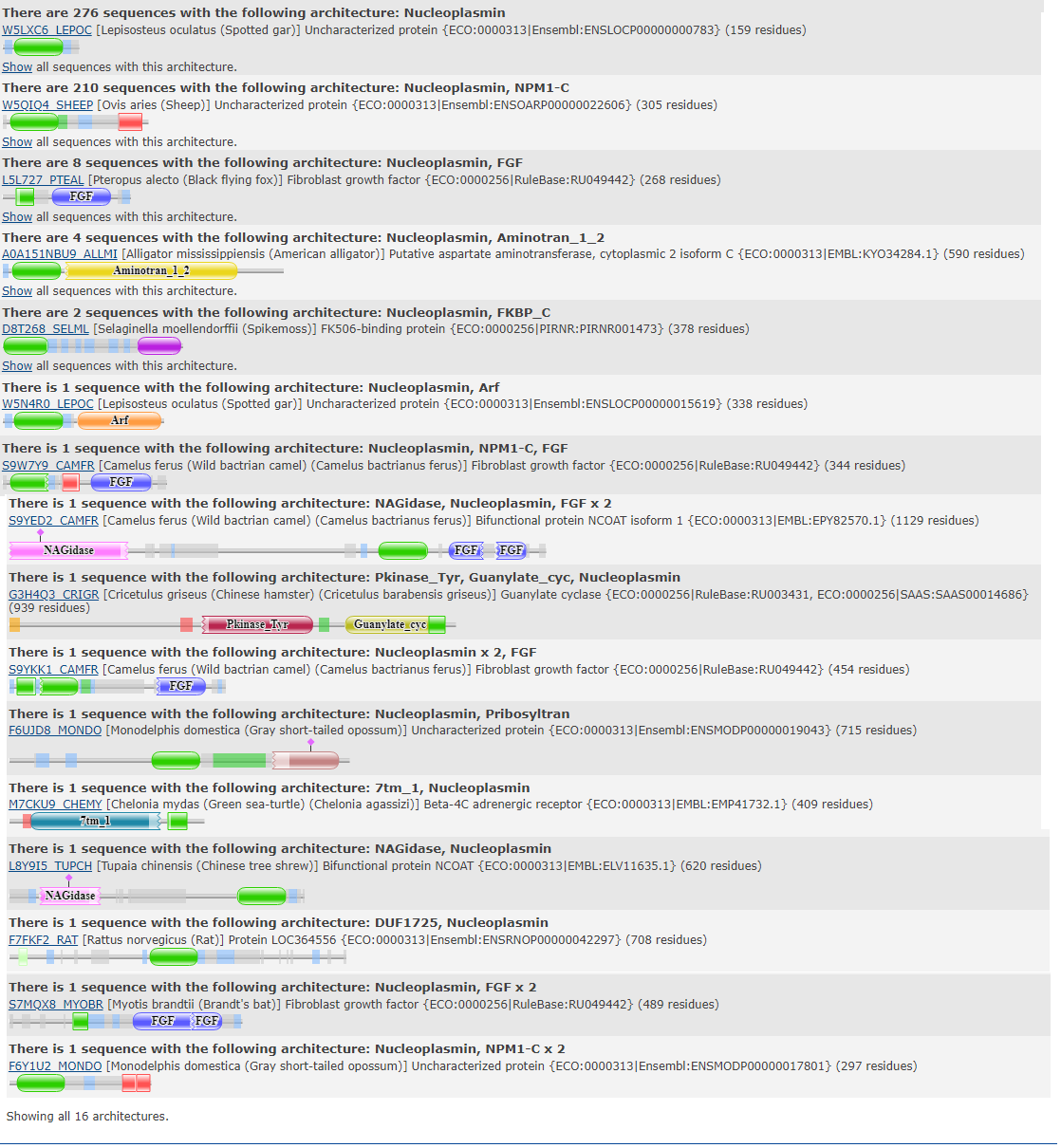
Для дальнейшей работы я выбрала первые две архитектуры.
Выравнивание N-концевых доменов было скачано с Pfam (full, в fasta-формате). Далее для человеческого NPM1 была загружена 3D структура из PDB. Скачать проект, скачать выравнивание в fasta-формате.
С помощью скрипта swisspfam_to_xls.py были отобраны последовательности с нуклеоплазминовым доменом и представлены ввиде таблицы Excel.
Команда:
python swisspfam_to_xls.py -z -i /srv/databases/pfam/swisspfam.gz -p PF03066 -o PF03066.xls
Таблица со всеми белками, содержащими нуклеоплазмновый домен.
Далее была создана таблица, где строками являются AC всех последовательностей, а по столбцам различные домены Pfam с указанием их встречаемости
в последовательностях. После этого для каждого AC в базе данных Uniprot была скачана их таксономия посредством скрипта uniprot_to_taxonomy.py.
Команда:
python uniprot-to-taxonomy.py -i uniprot2.txt -o taxonomy.xls
Получилась следующая таблица.
Я выбрала около 35 последовательностей с двумя доменными архитектурами: есть только N-концевой домен или есть и N-концевой, и С-концевой домен. Выбранные ID я собрала в отдельный файл, и с помощью скрипта вытащила соответствующие последовательности из выравнивания:
python filter-alignment.py -i ndomain.fasta -m selected.txt -o selected.fasta.
Полученное выравнивание было обработано: были удалены гэповые колонки, фрагменты и очевидные ошибки. Проект нового выравнивания с разбинением по группам можно скачать здесь (сверху - доменная архитектура из двух доменов, снизу - из одного).
Поскольку белки с нуклеоплазминовым доменом встречаются только у животных, в качестве таксона был выбран таксон животные (Animalia), а подтаксона - позвоночные (V) и беспозвоночные (I). Число 1 соответствует доменной архитектуре с N-концевым и С-концевыми доменами, в число 2 - доменной архитектуре с одним N-концевым доменом.
На основе полученного выравнивания с помощью метода Neighbor-joining было построено филогенетическое дерево домена:
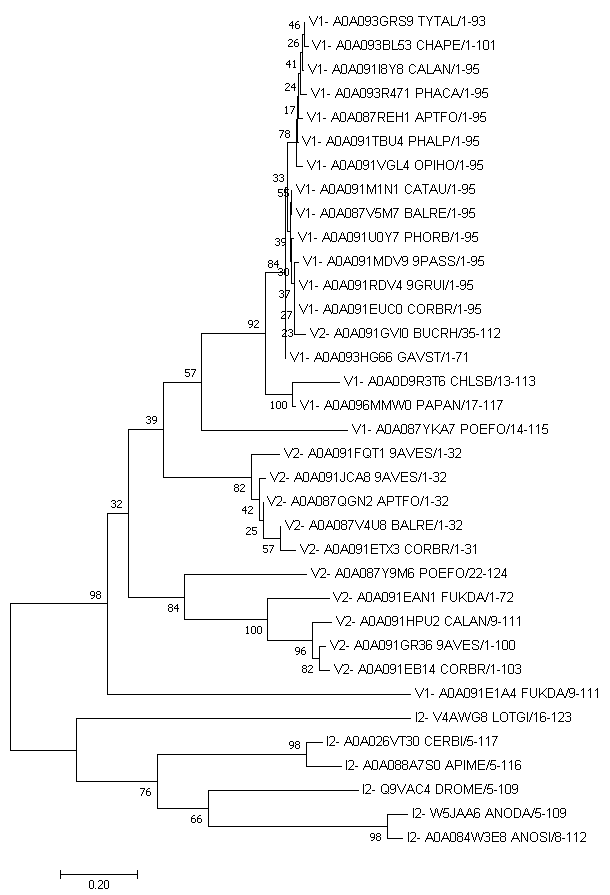
Можно видеть, что белки беспозвоночных со второй доменной архитектурой формируют отдельную кладу. Это подтверждается надёжной бутстрэп-поддержкой.
Файл со скобочной формулой дерева