1. Составление репрезентативной выборки гомологов белка YPWA_BACSU с помощью BLAST
Целью данного практикума было составить репрезентативную выборку гомологов моего белка (то есть выборка должна включать белки из разных таксономических групп). Затем выборка анализировалась с помощью множественного выравнивания. Для поиска гомологов в разных группах использовались следующие параметры работы BLAST:| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
| По прокариотам | Blastp (protein-protein BLAST) | RefSeq | Исключая Firmicutes и Eukaryota | 3 (989 находок; е последней 2,9) | 5 000 |
| По эукариотам | Blastp (protein-protein BLAST) | RefSeq | Eukaryota | 3 (35 находок; е последней 3e-43) | 5 000 |
| Домен | Филум/Царство | Название организма | Количество белков |
| Archaea | Euryarchaeota | Haladaptatus paucihalophilus DX253 | 1 |
| Archaea | Euryarchaeota | Haloferax mediterranei ATCC 33500 | 1 |
| Bacteria | Actinobacteria | Atopobium rimae ATCC 49626 | 1 |
| Bacteria | Actinobacteria | Atopobium vaginae DSM 15829 | 1 |
| Bacteria | Actinobacteria | Atopobium vaginae PB189-T1-4 | 1 |
| Bacteria | Bacteroidetes/Chlorobi group | Salinibacter ruber M8 | 1 |
| Bacteria | Bacteroidetes/Chlorobi group | Salinibacter ruber DSM 13855 | 1 |
| Bacteria | Bacteroidetes/Chlorobi group | Niastella koreensis GR20-10 | 1 |
| Bacteria | Bacteroidetes/Chlorobi group | Candidatus Amoebophilus asiaticus 5a2 | 1 |
| Bacteria | Cyanobacteria | Synechococcus sp. WH 8102 | 1 |
| Bacteria | Cyanobacteria | Synechococcus sp. CC9902 | 1 |
| Bacteria | Cyanobacteria | Synechococcus sp. WH 5701 | 1 |
| Bacteria | Cyanobacteria | Synechococcus sp. RCC307 | 1 |
| Bacteria | Cyanobacteria | Synechococcus sp. CB0101 | 1 |
| Bacteria | Cyanobacteria | Oscillatoria sp. PCC 6506 | 1 |
| Bacteria | Cyanobacteria | Oscillatoria nigro-viridis PCC 7112 | 1 |
| Bacteria | Cyanobacteria | Lyngbya sp. PCC 8106 | 1 |
| Bacteria | Proteobacteria | Francisella philomiragia subsp. philomiragia ATCC 25015 | 1 |
| Bacteria | Proteobacteria | Francisella philomiragia subsp. philomiragia ATCC 25017 | 1 |
| Bacteria | Proteobacteria | Francisella tularensis subsp. holarctica FSC022 | 1 |
| Bacteria | Proteobacteria | Francisella novicida FTE | 1 |
| Bacteria | Proteobacteria | Francisella novicida U112 | 1 |
| Bacteria | Proteobacteria | Francisella cf. novicida 3523 | 1 |
| Bacteria | Proteobacteria | Francisella tularensis subsp. holarctica LVS | 1 |
| Bacteria | Deinococcus-Thermus | Deinococcus peraridilitoris DSM 19664 | 1 |
| Bacteria | Deinococcus-Thermus | Deinococcus maricopensis DSM 21211 | 1 |
| Bacteria | Deinococcus-Thermus | Leptospira interrogans str. 2002000626 | 1 |
| Bacteria | Firmicutes | Bacillus subtilis str.168 | 1 |
| Bacteria | Nitrospinae | Nitrospina gracilis 3/11 | 1 |
| Eukaryota | Viridiplantae | Chlamydomonas reinhardtii | 1 |
| Eukaryota | Plantae | Cucumis sativus | 1 |
| Eukaryota | Plantae | Ostreococcus lucimarinus CCE9901 | 1 |
| Eukaryota | Plantae | Physcomitrella patens subsp. patens | 1 |
| Eukaryota | Plantae | Selaginella moellendorffii | 2 |
| Eukaryota | Chromalveolata | Phaeodactylum tricornutum CCAP 1055/1 | 1 |
| Eukaryota | Chromalveolata | Thalassiosira pseudonana CCMP1335 | 1 |
| Eukaryota | Viridiplantae | Micromonas pusilla CCMP1545 | 1 |
| Eukaryota | Viridiplantae | Micromonas sp. RCC299 | 1 |
| Eukaryota | Euglenozoa | Leishmania donovani | 1 |
| Eukaryota | Euglenozoa | Leishmania major strain Friedlin | 2 |
| Eukaryota | Euglenozoa | Leishmania braziliensis MHOM/BR/75/M2904 | 1 |
| Eukaryota | Euglenozoa | Leishmania infantum JPCM5 | 1 |
| Eukaryota | Euglenozoa | Leishmania mexicana MHOM/GT/2001/U1103 | 1 |
| Eukaryota | Euglenozoa | Trypanosoma brucei brucei strain 927/4 GUTat10.1 | 1 |
| Eukaryota | Euglenozoa | Trypanosoma cruzi strain CL Brener | 2 |
| Eukaryota | Percolozoa | Naegleria gruberi | |
3. Построение и анализ множественного выравнивания с помощью Muscle и JalView (соответственно)
Последовательности были выровнены с помощью дополнительных опций в программе JalView, через Web Service --> Alignment--> Muscle of Defaults. Файлы с сохраненными выравниваниями находятся здесь:В результате были получены картинки выравниваний для гомологов среди эукариот и прокариот:
1.
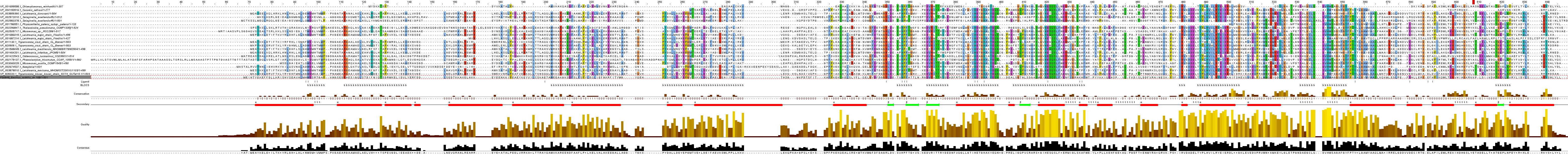
2.
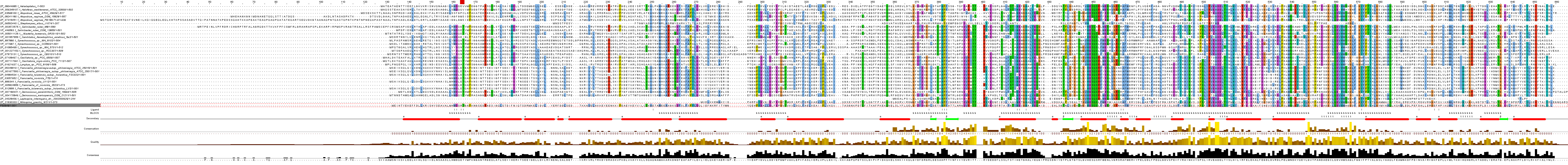
На данных выравниваниях использовалась покраска CLustalX с cut-off 60%, таким образом выделяются цветом только достаточно консервативные участки белка. Строка SECONDARY отражает вторичную структуру (t означает поворот цепи, т.е. неструктурированные участки). Строка LIGAND отмечает аминокислоты, вовлеченные в координацию многочисленных лигандов.
Выравнивание в целом можно отметить как достаточно хорошее и для прокариотических гомологов, и для эукариотических гомологов. Замечается следующий факт: участки наибольшего сходства организованные в блоки часто совпадают или как минимум частично перекрываются с элементами вторичной структуры. Внутри элементов вторичной структуры колонок-гепов немного меньше, что связано непосредственно со сложностями сохранения единства вторичной структуры, возникающими при изменении последовательности. Области блоков также в абсолютном своем большинстве содержат и аминокислоты, координирующие лиганды. Это вполне объяснимо именно функциональной важностью и значимостью этих участков, которые видимо сложно изменять без потери функции белка. Кроме того, иногда блоки в выравнивании у эукариот и прокариот совпадают, что говорит о их чрезвычайной консервативности. Самое плохое выравнивание наблюдается обычно на концах последовательности и в неструктурированных участках цепи, часто располагающихся на поверхности белка. Блоки искались как участки хорошего множественного выравнивания без гэпов (в большинстве случаев). Однако этот факт иногда не учитывался, если из хорошего выравнивания выбивалась лишь 1-2 последовательности.
Как уже было замечено выше, лиганд-связывающие сайты почти всегда находились внутри достаточно консервативных блоков, из чего можно заключить, что гомологи исследуемого белка способны связывать лиганды тоже, и, вероятно, имеют ту же функцию в клетке.
На рисунке 1 представлены остатки белка, координирующие все лиганды (расстояние от лиганда меньше 5 ангстремов). Некоторые из них подписаны в силу обилия лигандов подписи сделаны не для всех во избежание путаницы и загромождения рисунка.
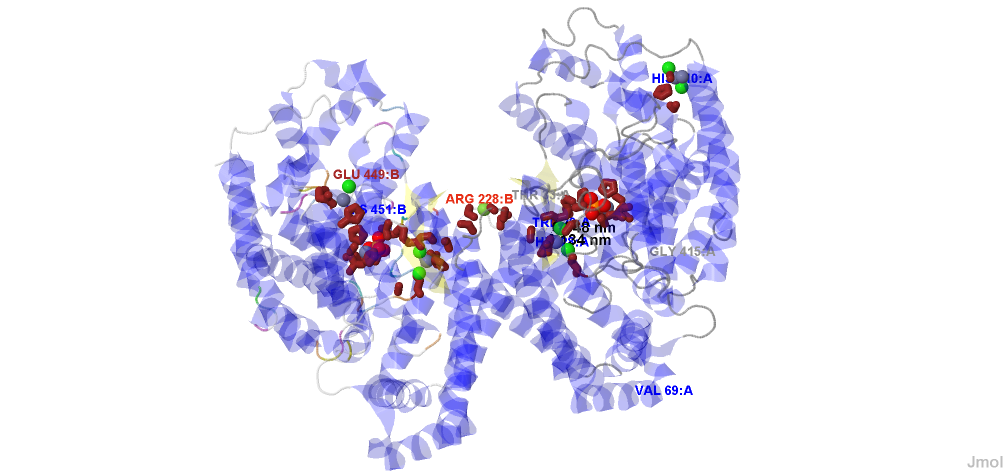 |
| Рис. 1. Лиганды белка и координирующие остатки. Цвет белковой последовательности в соответствии со вторичной структурой (синие альфа-спирали, желтые бета-листы). Координирующие лиганды аминокислотные остатки выделены wireframe и коричневым цветом. |