A-, B- и Z-формы ДНК
 |
| Рис.1. А-форма ДНК |
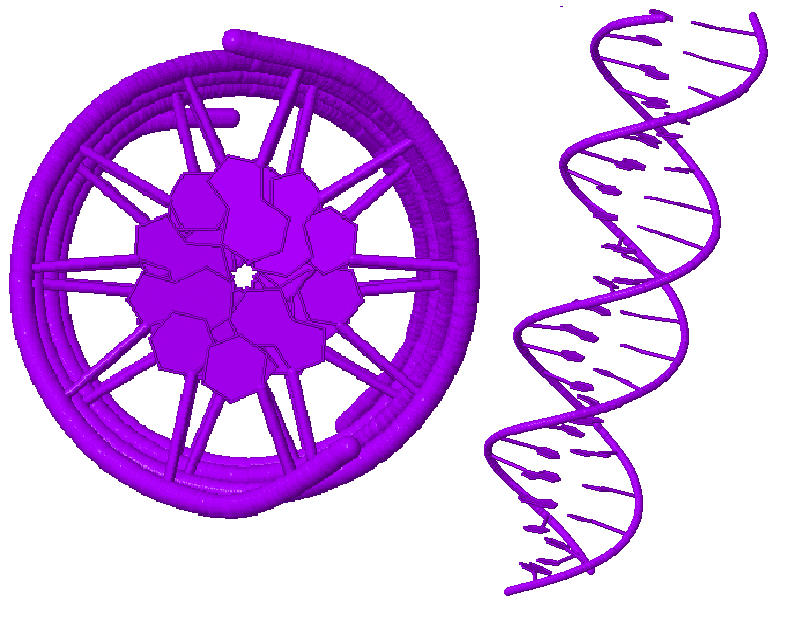 |
| Рис.2. В-форма ДНК |
 |
| Рис.3. Z-форма ДНК |
С помощью инструментов пакета 3DNA были построены модели структур A, B и Z форм ДНК. Для этого после подключения к серверу Kodomo и перехода в рабочую директорию были введены следующие команды для указания пути к 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
Командой fiber строились различные формы ДНК последовательности GATC, повторяющейся 5 раз; синтаксис команды:
fiber -a gatc-a.pdb (Файл gatc-a.pdb)
fiber -b gatc-b.pdb (Файл gatc-b.pdb)
fiber -z gatc-z.pdb (Файл gatc-z.pdb)
На рисунках 1, 2 и 3 представлены формы ДНК с торца и сбоку.Для тренировки работы с ДНК в JMol были выделены и покрашены различные химические группировки A-формы ДНК, полученой в предыдущем задании (см. рис. 2):
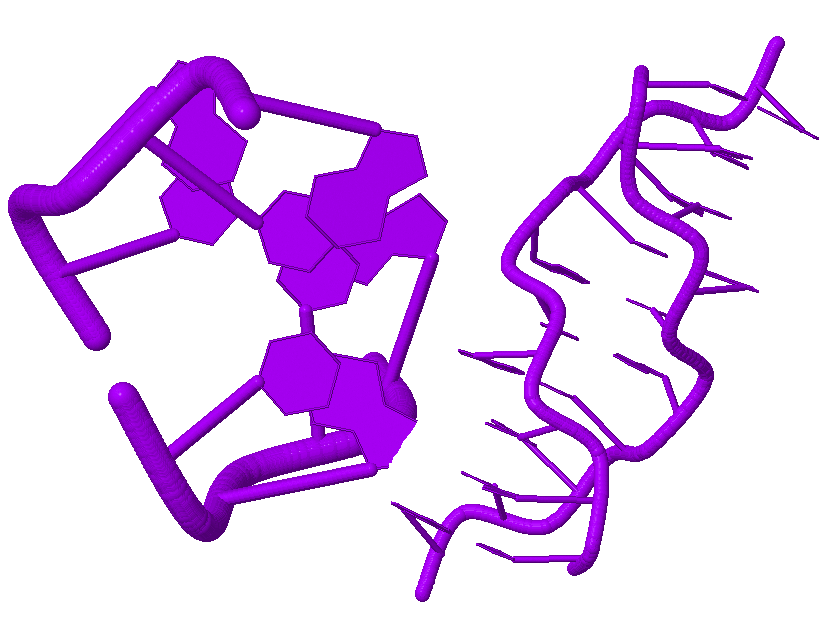
 |
| Рис. 2. Выделение химических группировок ДНК. A - покрашены все нуклеотиды; B - выделены все аденины; С - покрашены атомы N7 во всех гуанинах; D - выделен сахарофосфатный остов ДНК. |
Для дальнейшей работы с РНК, ДНК и комплексами из БД PDB были скачаны pdb-файлы, которые затем редактировались (удалялись лишние цепи, посторонние молекулы). Результатом работы стали следующие файлы:
- 1N77.pdb - тРНК в комплексе с белком, а также 1N77_onlyRNA.pdb - без белков
- 1A02.pdb - ДНК-белковый комплекс, а также 1A02_onlyDNA.pdb - без белков
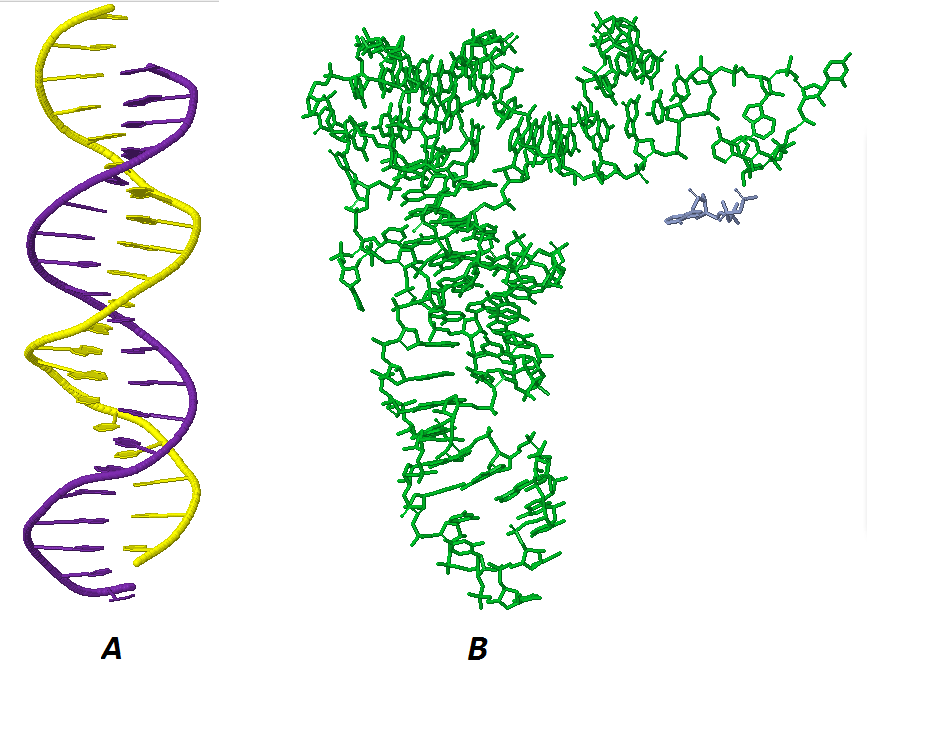 |
| Рис. 4. Структуры ДНК (А) и РНК (B) из комплексов с белками. Цвета отличают нуклеотиды, принадлежащие разным цепям; АТФ из B формально принадлежит цепи белковой молекулы. |
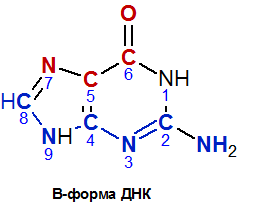 |
| Рис. 5. Экспонирование атомов гуанина между большой и малой бороздками |
В-форма ДНК отличается следующим набором атомов, обращенных в cторону большой и малой бороздок:
- В сторону большой бороздки обращены атомы C6, O6, C5, N7, C8
- В сторону малой бороздки обращены атомы C2, N2, N3, C4, N9
- Остальные атомы основания обращены в сторону соседней цепи ДНК
Сравнение параметров форм ДНК
Табл. 1. Сравнение параметров различных форм ДНК
| 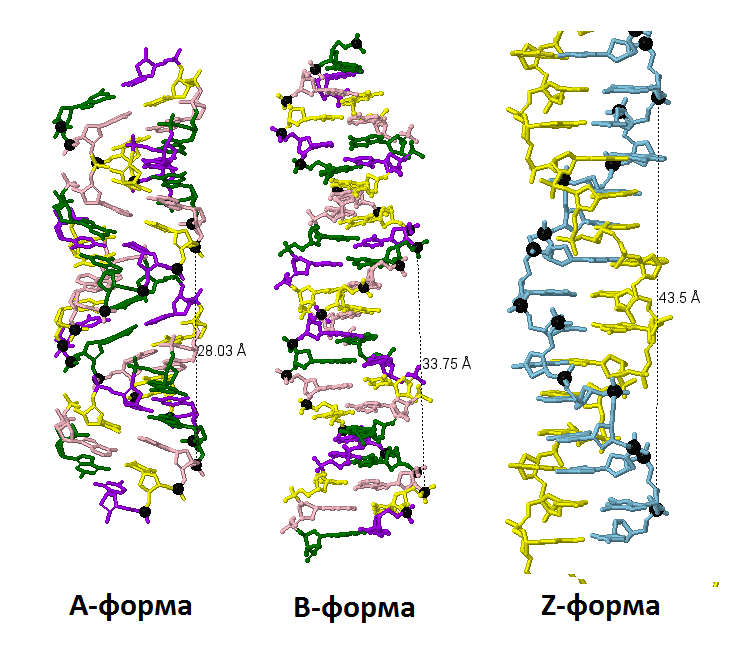 | ||||||||||||||||||||||||
| Рис. 6. Иллюстрация к таблице 1. Черным цветом отмечены атомы фосфора; расстояние между ними считалось за шаг спирали; для формы А и В разными цветами покрашены разные типы нуклеотидов. |
Следует отметить, что необычная организация А-формы ДНК приводит к тому, что малая бороздка становится шире большой бороздки.
| Табл. 2. Сравнение торсионных углов | ||||||||||||||||||||||||||||||||||||||||
|
Анализ последовательностей с помощью программ find_pair и analyze.
Для трех форм ДНК и последовательности ДНК, полученной ранее, были определены средние значения торсионных углов. Для этого результаты работы команды find_pair подавались на вход команде analyze. Предварительно pdb-файлы переводились в старый формат, с которым работает команда find_pair.
Например,
remediator --old 1A02_onlyDNA.pdb > 1A02_onlyDNA__old.pdb
find_pair -t 1A02_onlyDNA_old.pdb stdout | analyze
| Табл. 3. Значения торсионных углов | ||||||||||||||||||||||||||||||||||||||||
|
Среди последовательности из файла 1A02_onlyDNA_old.pdb у части нуклеотидов наблюдалось очень большое отклонение значений торсионных углов от среднего значения (например, 13-15 нуклеотиды в цепи А). Прямой зависимости я не наблюдаю, возможно лишь, на мой взгляд, это следствие большого сосредоточения на одной цепи пуринов, а на другой - пиримидинов.
Вторичная структура тРНК
Вторичная структура тРНК из файла 1N77_onlyRNA_old.pdb имеет следующие особенности:- Нуклеотиды нумеруются с 501 по 576, причем участок [501-519] образует водородные связи с участком [556-572], формируя спираль, внутри которой отличают:
- [501-506] & [567-572] - акцепторный стебель
- [549-552] & [562-565] - дигидроуридиновый стебель
- [539-543] & [527-531] - антикодоновый стебель
- [510-512] & [523-526] - Т-стебель
- В последовательности есть неканонические, т.н. минорые нуклеотиды, водородные связи с которыми не являются Уотсон-Криковскими. Кроме того, мажорные нуклеотиды также могут взаимодействовать не по Уотсон-Криковскиму типу. Здесь приведена картинка, иллюстрирующая информацию об этих необычных водородных связях.
- Стабилизирующие структуру водородные связи образуются между следующими нуклетидами: {507}&{566}, [553-538]&{532, 558, 561}, {544}&{526}, [513-519]&{556, 548, 508, 522}; ({x,y} - отдельные нуклеотиды х, у; [x,y] - все нуклеотиды от х до у.)
Третичная структура тРНК
Был произведе поиск соседних комплементарных пар, то есть находящихся друг под другом, с наибольшей площадью перекрывания азотистых оснований - в проекции на торец молекулы. Таких пар оказалось 2:2 (0.005) C:.502_:[..G]G-*---U[..U]:.571_:C (0.008) | 17 (0.006) C:.540_:[..G]G-----C[..C]:.530_:C (0.004) | 3 (0.004) C:.503_:[..C]C-----G[..G]:.570_:C (0.004) | 18 (0.003) C:.541_:[..C]C-----G[..G]:.529_:C (0.008) | step i1-i2 i1-j2 j1-i2 j1-j2 sum [*1] GC/GU 6.97( 4.16) 0.00( 0.00) 0.00( 0.00) 6.44( 3.84) 13.40( 8.00) [*2] GC/GC 6.06( 3.06) 0.00( 0.00) 0.00( 0.00) 7.09( 4.17) 13.15( 7.23)- области перекрывания; где in, jn - n-ые нуклеотиды антинаправленных участков цепи. В файле stacking.pdb были найдены структуры, моделирующие эти взаимодествия (ориентация на нумерацию атомов) и зафиксированы их номера. Командой
ex_str -№ stacking.pdb step№.pdb,
где № - номер структуры, были вырезаны эти структуры в отдельные файлы step№.pdb. Затем командойstack2img -cdolt step№.pdb step№.ps
были построены изображения, присутствующие на рис. 7.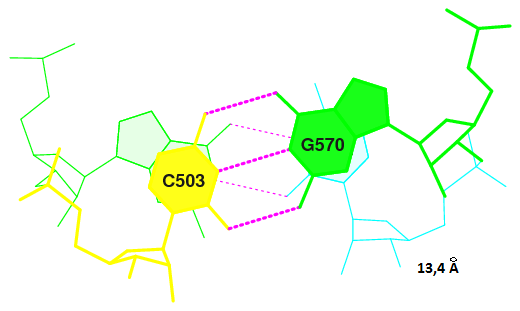 | 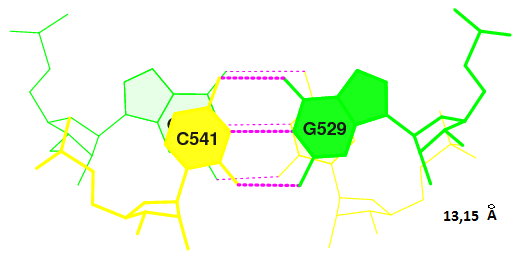 |
| Рис. 7. Стекинг-взаимодействия между азотистыми основаниями: [*1] и [*2]. Цвета отличают нуклеотиды, принадлежащие разным цепям; АТФ из B формально принадлежит цепи белковой молекулы. |
|