Комплексы ДНК - белок
| |
| Контакты атомов белка с |
Полярные |
Неполярные |
Всего |
| остатками 2'-дезоксирибозы |
4 |
10 |
35 |
| остатками фосфорной кислоты |
16 |
0 |
98 |
| остатками азотистых оснований со стороны большой бороздки |
4 |
2 |
23 |
| остатками азотистых оснований со стороны малой бороздки |
0 |
1 |
6 |
|
Командой
nucplot, работающей со старым форматом pdb, было получено изображение контактов между ДНК и белком.
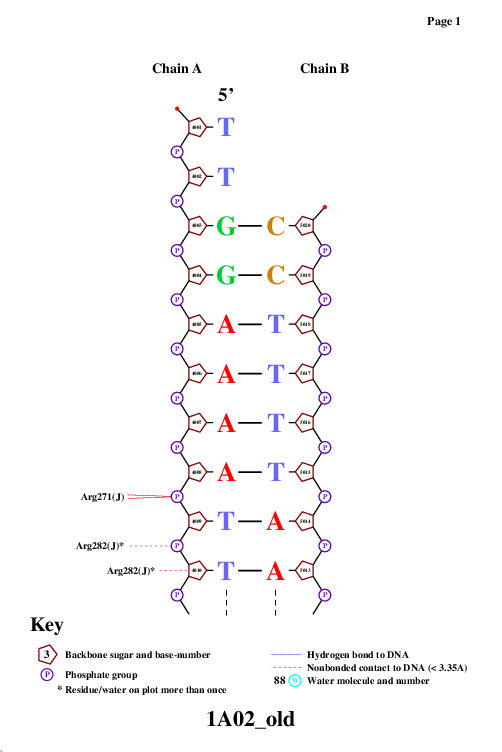 | 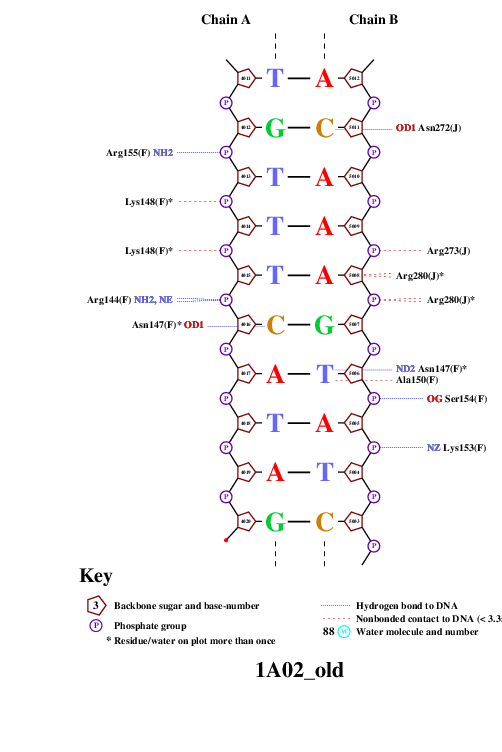 |  |
| Рис. 1. Схема ДНК-белковых контактов |
На схеме можно отметить несколько аминокислот, имеющих больше контактов с ДНК, нежеди другие аминокислотные остатки.
Это Arg271, Arg280, Lys148, Asn147. Но максимальное количество водородных связей образует Arg280 (4); вероятно, он играет большую роль
при возникновении взаимодействий между белковой молекулой и ДНК. Это взаимодействие также наверняка облегчается разноименной заряженностью
ДНК и аргинина. Кроме того, важность этого участка белка подтверждается и повышенной концентрацией здесь других водородных связей.
Другое дело, аргинин не взимодействует непостредственно с азотистыми основаниями, следовательно, не может играть участие в распознавании
последовательности ДНК белком. Возможно, эти связи важны для наилучшей координации белка относительно ДНК.
Связь с азотистыми основаниями наблюдается только у Asn147 (с цитозином и тимином) и Ala150 (с тем же тимином). Поэтому, безусловно, они
имеют большое значение в распознавании последовательности. Так как, собственно, распознается лишь пара нуклеотидов, то о распознавании специфической первичной
структуры говорить не приходится. Возможно, связывание определяется другими факторами.
На рисунке 2 иллюстрированы взаимодействия двух выбранных аминокислот с ДНК.
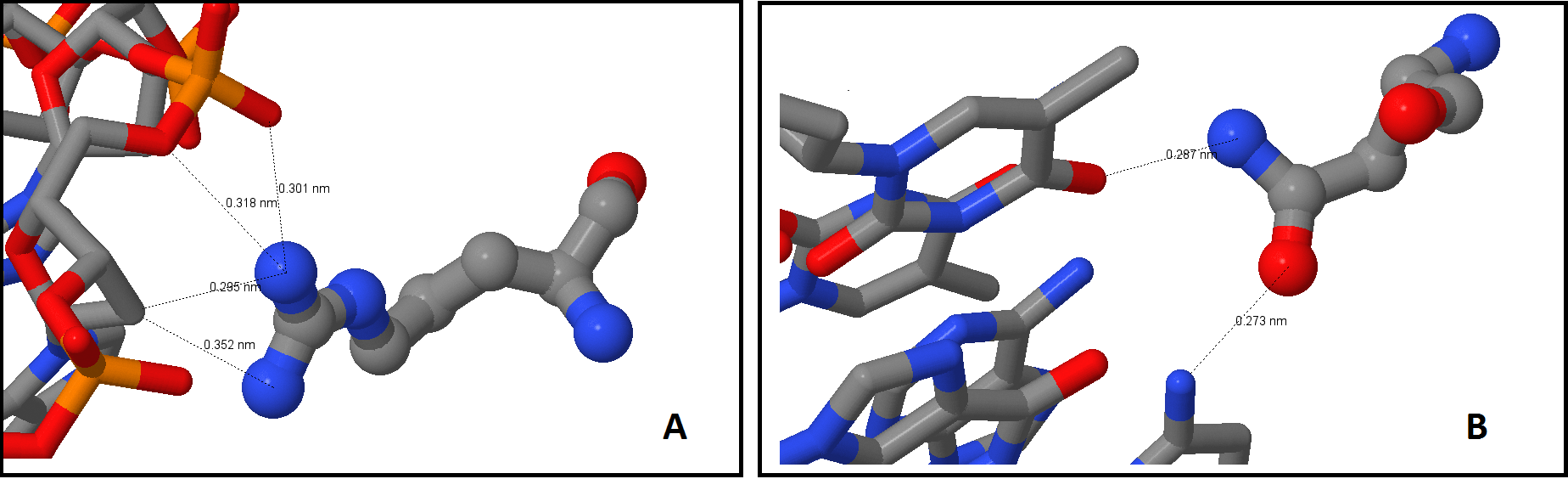 |
| Рис. 2. Контакты между ДНК и выбранными аминокислотами. A - Arg280 и ДНК; B - Asn147 и ДНК. Пунктиром
отмечены образующиеся связи с минимальной длиной, которая пописана. |
Предсказание вторичной структуры заданной тРНК
С помощью команды
einverted были определены комплементарные друг другу участки в последовательности
ДНК. Однако наилучшие подобранные параметры (шестая попытка, последующие не дали лучшего результата) не дали полного сходства с
определенными с помощью
find_pair стеблями. Совпал только 1 стебель - акцепторный. Алгоритм Зукера, однако,
лучше подошел для этих же целей. С помощью него с первой попытки не только нашлись все известные стебли, но и произошло удлинение одного из них.
Визуализации этих предсказаний предствлены на рисунке 3.
 |
| Рис. 3. Различные варианты предсказаний алгоритмов. A - команда einverted; B - программа mfold. |
| Табл. 1. Реальная и предсказанная вторичная структура тРНК из файла 1N77.pdb |
| Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель |
5'-501-506-3'
5'-567-572-3'
Всего 6 пар |
Предсказаны все пары |
Предсказаны все пары |
| D-стебель |
5'-549-552-3'
5'-562-565-3' |
Не предсказан |
Предсказаны все пары |
| T-стебель |
5'-510-512-3'
5'-523-526-3' |
Не предсказан |
Предсказаны все пары |
| Антикодоновый стебель |
5'-539-543-3'
5'-527-531-3' |
Определены неверно |
Предсказаны все пары |
| Общее число канонических пар нуклеотидов |
18 |
13 |
22 |
|




