Для домена известна 31 архитектура.
- Lipase - 1376 последовательностей
- Lipase, PLAT - 441 последовательности
- Lipase x 2 - 50 последовательностей
- Lipase x 2, PLAT - 8 последовательностей
- PMC2NT, DNA_pol_A_exo1, HRDC, Lipase - 3 последовательности
- Ldl_recept_a, Lipase - 2 последовательности
- Lipase, zf-H2C2_2 x 2 - 1 последовательность
- Lipase x 4 - 1 последовательность
- BTB, BACK, Kelch_1, Lipase - 1 последовательность
- Lipase, SCO1-SenC - 1 последовательность
- Lipase, GMC_oxred_N, GMC_oxred_C - 1 последовательность
- HEAT, HEAT_2, Lipase - 1 последовательность
- PLAT, Lipase, HemolysinCabind x 4 - 1 последовательность
- Med23 x 2, Lipase - 1 последовательность
- Lipase, Acetyltransf_1 - 1 последовательность
- Lipase x 3 - 1 последовательность
- Lipase x 3, PLAT - 1 последовательность
- Lipase, HemolysinCabind x 5 - 1 последовательность
- Lipase x 6 - 1 последовательность
- PAX, Lipase - 1 последовательность
- Lipase, BolA - 1 последовательность
- Lipase, Pep_M12B_propep
- Lipase, ThiF, UBA_e1_thiolCys, UBACT, E2_bind - 1 последовательность
- Lipase, Glyco_hydro_1 - 1 последовательность
- Big_5, Cadherin x 2, Lipase - 1 последовательность
- Lipase, CDP-OH_P_transf - 1 последовательность
- PP1c_bdg, Lipase - 1 последовательность
- Lipase, Peptidase_C1 - 1 последовательность
- HCBP_related, Lipase - 1 последовательность
- Lectin_C, Lipase, DUF2464 x 2 - 1 последовательность
- Lipase, adh_short - 1 последовательность
C помощью скрипта swisspfam_to_xls.py из файла swisspfam.gz, содержащего информацию для всех последовтельностей Uniprot была составлена таблица, содержащая последовательности с указанным доменом. Затем в таблицу добавили столбцы с таксономической принадлежностью: по AC последовательностей получили flat text (Retrieve из Uniprot), а затем перенесли в сводную таблицу.
Для дальнейшей работы были выбраны две доменные архитектуры:
- Lipase: 1376 последовательностей;
- Lipase, PLAT: 441 последовательность.
PLAT/LH2 - домен, который часто встречается в белках, связанных с мембраной или липидами. Он опосредует связь с мембранами.
Выбор таксона и подтаксонов
В качество таксона я выбрала Euteleostomi, в качестве подтаксонов - Actinopterygii, Amphibia и Mammalia.Euteleostomi - успешная группа, вкулючающая 90% современных позвоночных. Относится к челюстноротым. Соответственно, подтаксон Actinopterygii - это класс лучеперых рыб, самый многочиленный класс позвоночных, подтаксон Amphibia - Земноводные, а Mammalia - Млекопитающие. На отдельном листе сводной таблицы записаны идентификаторы, доменная организация и таксономия выбранных мною последовательностей. В выравнивание добавлена последовательность этого домена с известной структурой (последняя строка). Кроме того, выравнивание отредактировано (удалены пустые колонки, плохо выравнивающиеся N- и C-концы, фрагменты вместо целых последовательностей). Вручную подправлено несколько гэпов. Последовательности разделены на 2 группы в соответствии с доменной архитектурой: первая - это архитектура Липаза+PLAT, вторая - Липаза; обе группы покрашены Clustalx и по порогу консервативности 10%.
Полученное выравнивание можно увидеть на рисунке 1 и в файле.
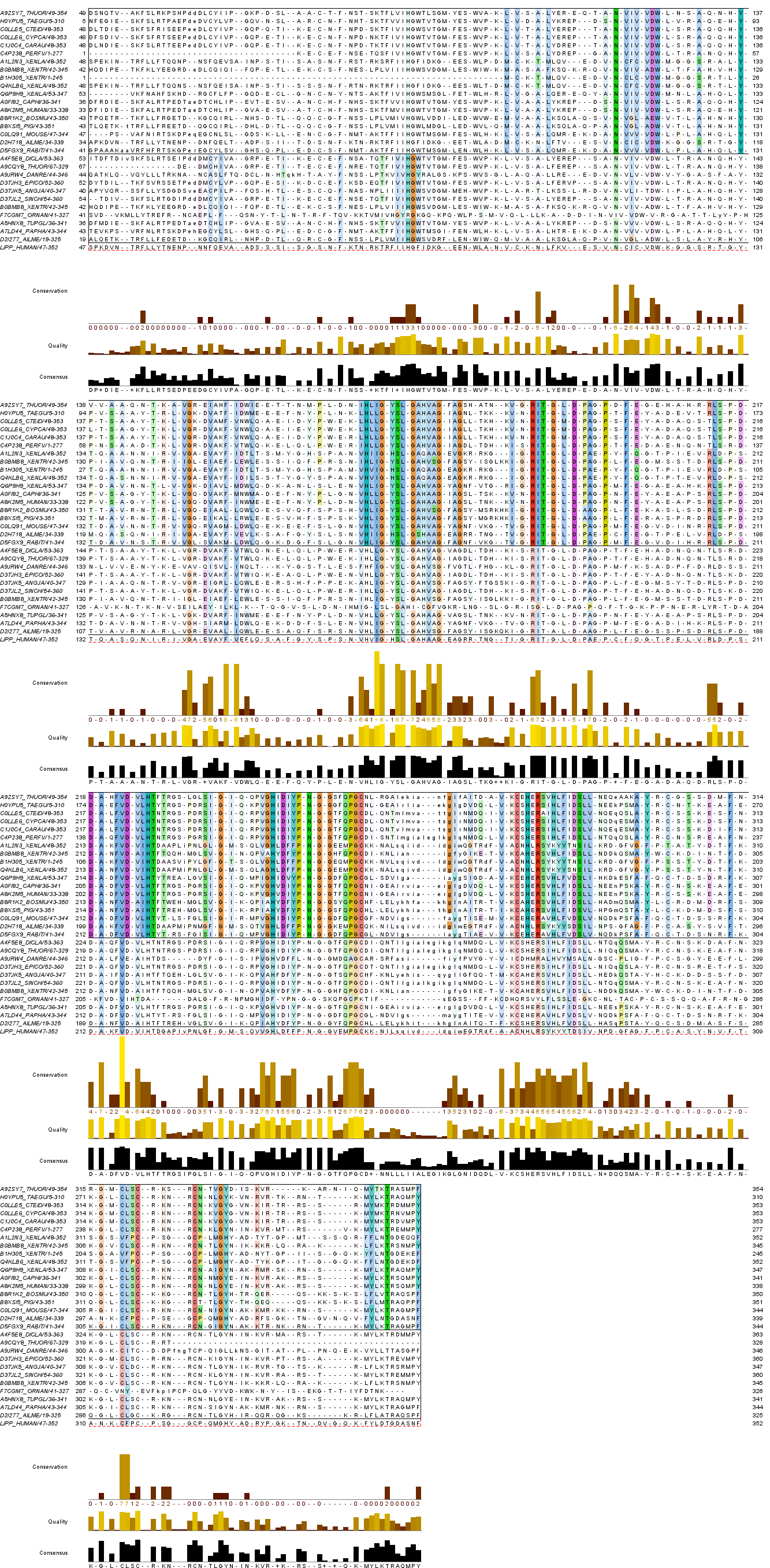 |
| Рис.1. Выравнивание доменов. |
Для удобства построения и визуализации филогенетичекого дерева были приняты следующие обозначения:
| 1 | Однодоменая архитектура Lipase |
| 2 | Двудоменная архитектура Lipase+PLAT |
| M | Mammalia |
| Am | Amphibia |
| Ac | Actinopterygii |
Например, 2_M_ID - это последовательность, относящаяся к архитектуре Lipase+PLAT из Млекопитающих.
Отформатированные последовательности использвали для построения филогенетического дерева в JalView методом NJ Using % Identity.
С использованием сервиса ITOL была создана следующая окраска листьев:
- 1_M - желтый
- 1_Am - темно-зеленый
- 1_Ac - синий
- 2_M - красный
- 2_Am - бирюзовый
- 2_Ac - малиновый
 |
| Рис.2. Филогенетическое дерево. |
Скобочная формула дерева выглядит следующим образом:
((1_Am_F7CGM7:20.548546,(((Structure:6.653141,2_M_D2H718:6.5491066):8.29822,
(2_Am_B1H305:0.0,(2_Am_Q4KLB6:4.7552147,2_Am_A1L2N3:4.7953453):5.6260815):5.536051):13.883747,
(((1_M_D2I277:9.817097,(2_M_B8XSI5:9.973434,2_M_B8R1K2:9.689484):0.997509):8.251818,
(1_Ac_D3TJK5:15.008562,(1_Am_B0BMB8:0.0,2_Am_B0BMB8:0.0):14.766722):2.8787994):4.8006334,
((((1_M_A7LD44:4.1741905,2_M_D5FGX9:4.8145714):0.7658901,2_M_C0LQ91:4.852087):6.477682,2_Am_Q6P9H9:10.938047):5.873478,
(((((1_Ac_D3TJH3:6.5953827,(1_Ac_A9CQY8:0.0,(1_Ac_D3TJL2:1.7169981,1_Ac_A4F5E8:2.2155886):2.7820292):0.7079849):1.0535545,
2_Ac_C4P238:1.7203236):2.718134,((2_Ac_C1J0C4:2.4020987,2_Ac_C0LLE6:1.8113871):0.8185539,2_Ac_C0LLE5:4.097172):8.21059):3.4634428,
(((1_M_A5HNX8:1.4959726,2_M_A8K2M5:1.5939169):0.8783026,2_M_A0FI82:2.773385):5.7223167,2_Ac_H0YPU5:7.9012794):4.6003284):4.4787464,
2_Ac_A9ZSY7:15.920986):2.582017):2.4637861):6.20018):0.055858612):13.46168,1_Ac_A9JRW4:13.46168);
Обе архитектуры присутствуют во всех выделенных подтаксонах. Это говорит о том, что архитектуры уже присутствовали у их предка. С другой стороны, архитектуры в основном не разделены между собой, то есть чаще на филогенетическом дереве теснее стоят таксоны, а не архитектуры. Скорее всего, это говорит о том, что архитектуры появлялись независимо в разных подтаксонах. Так, например, это демонстрируют Лучеперые рыбы, в которых, по-видимому, дупликация липазы с последующим присоединением к одному из генов домена PLAT произошла относительно недавно. Домены в обеих архитектурах очень похожи. Тем не менее, есть 4 "выброса" у Лучеперых рыб, таким образом данное суждение не охватывает всего многообразия их видов. То же касается и остальных подтаксонов. Из-за большого количества таких "выбросов" сложно с уверенностью говорить о том, что возникнование двудоменной архитетуры было независимым у разных классов. Таким образом, я склоняюсь к мысли, что обе архитектуры у предка присутствовали (получившись в результате дупликации), и их эволюция в классах независима. Но доменная архитектура довольно лабильна, и возможно, были повторные дупликации, приведшие к созданию довольно простой архитектуры Lipase+PLAT вторично в образовавшихся классах (Actinopterigii и Mammalia). Тем более, что PLAT, судя по литературным данным, очень часто встречается вместе с белками, функция которых сопряжена с липидами.
© Elizaveta Besedina, FBB 2012
lizaveta@kodomo.fbb.msu.ru