Множетсвенное выравнивание
Для этого задания я выбрала белки с ID BLA34_ECOLX, BLAC_BACLI, BLAC_STRBA, BLA1_STRCI. Я решила сравнивать выравнивание Jalview, Muscle и выравнивание Align на сайте Uniprot. Ссылка на проект с этими двумя выравниваниями (d_align.fasta - выравнивание с сайта Uniprot, нижнее окно - Jalview).
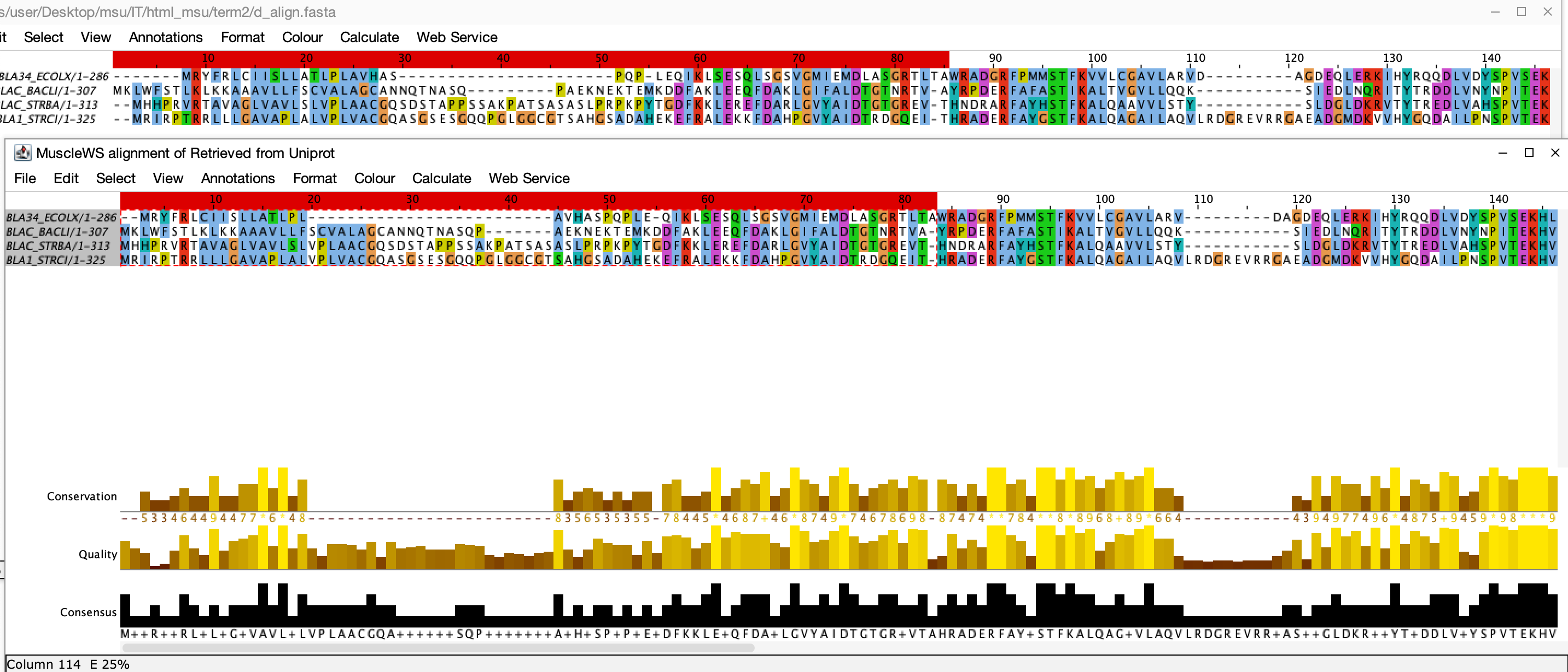
Совпадающие позиции выравнивания - 52-83 (координаты по верхнему выравниваню Uniprot), 52-83, 86-110, 121-175, 178-233, 239-335.
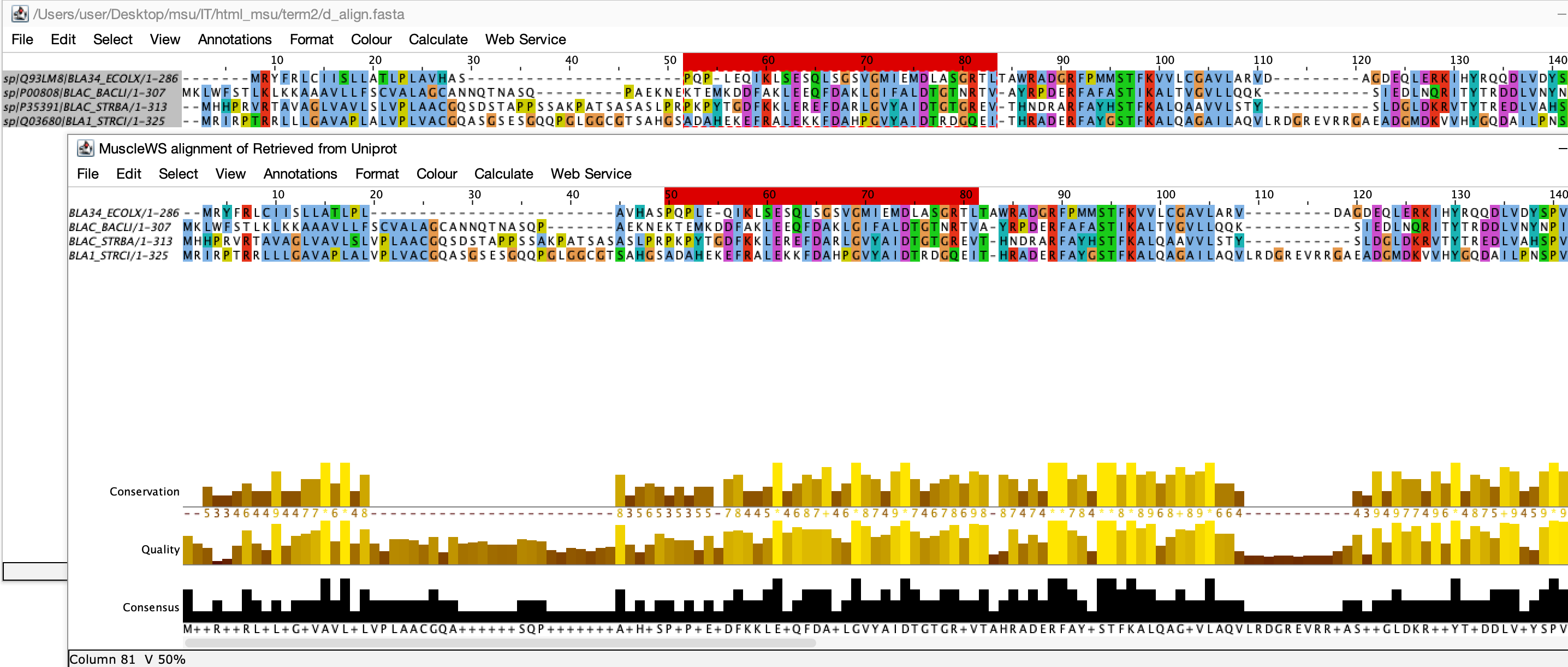
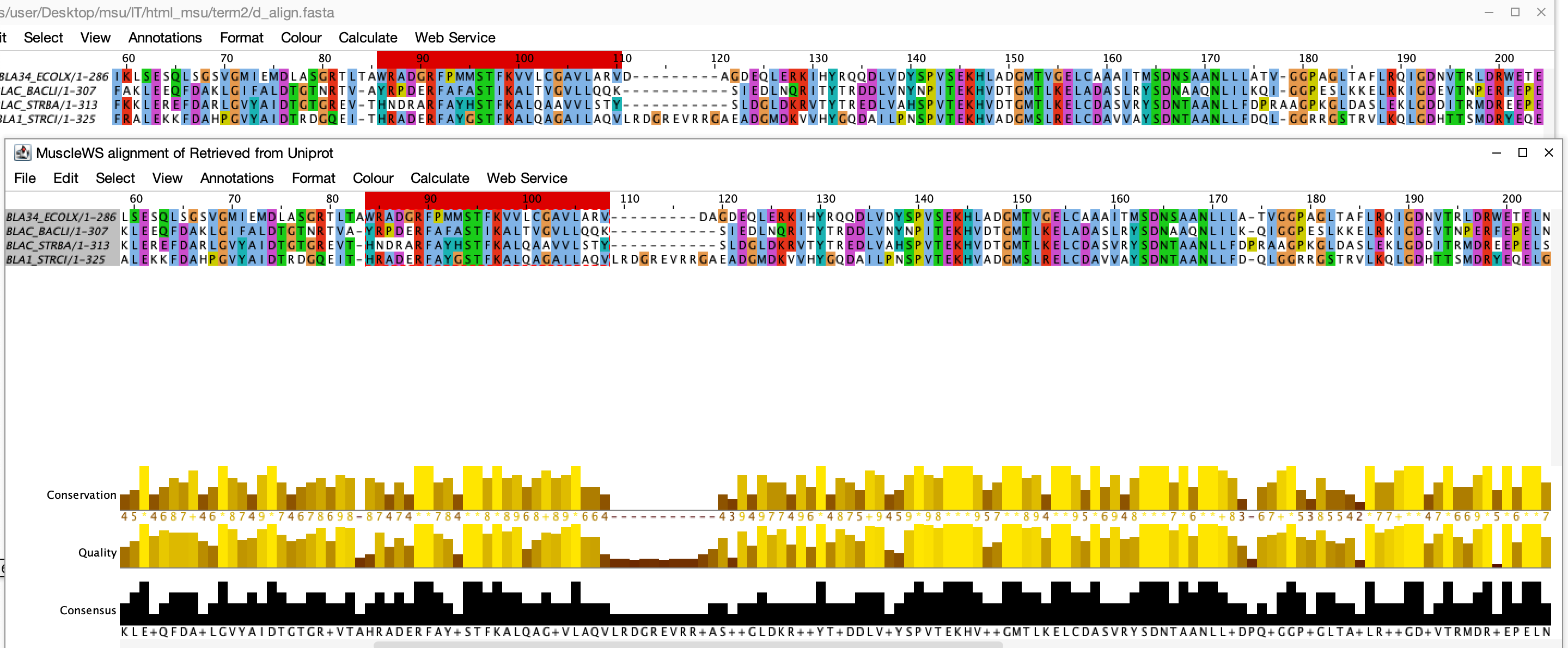
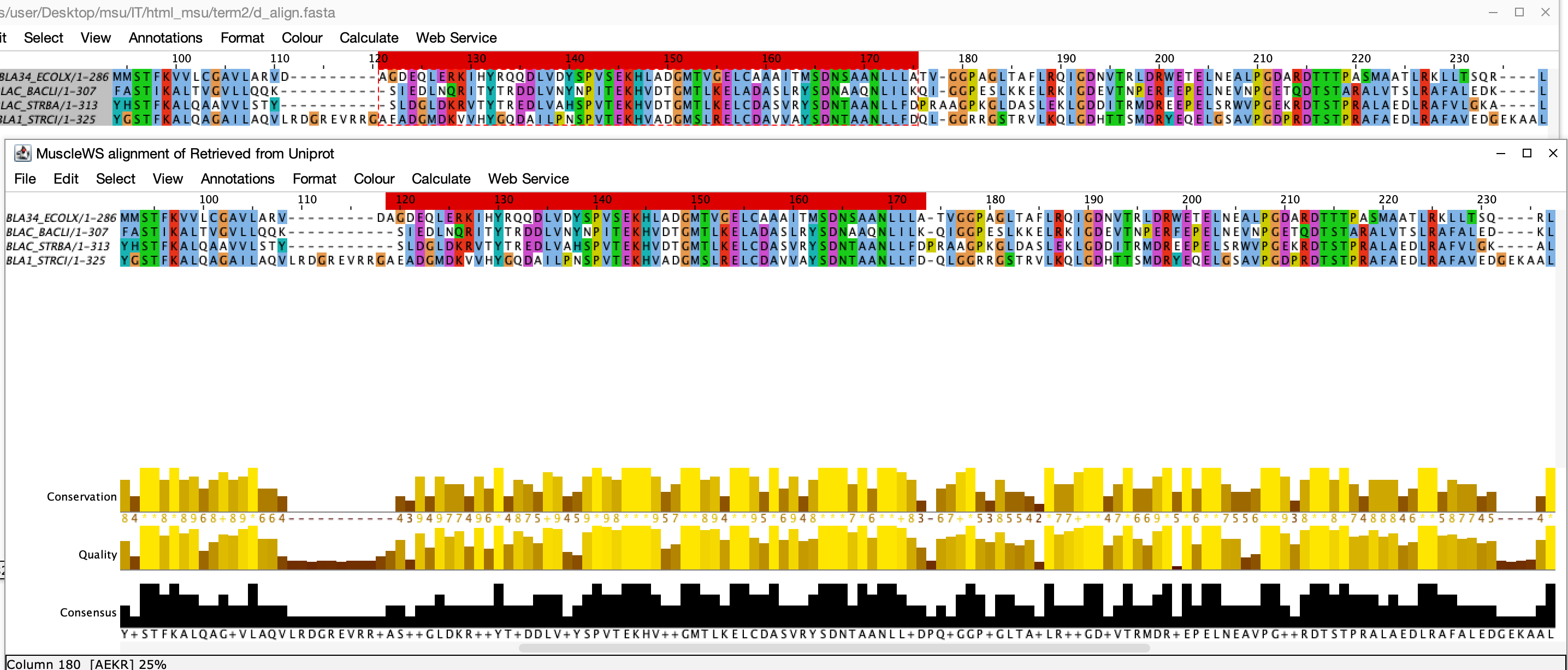
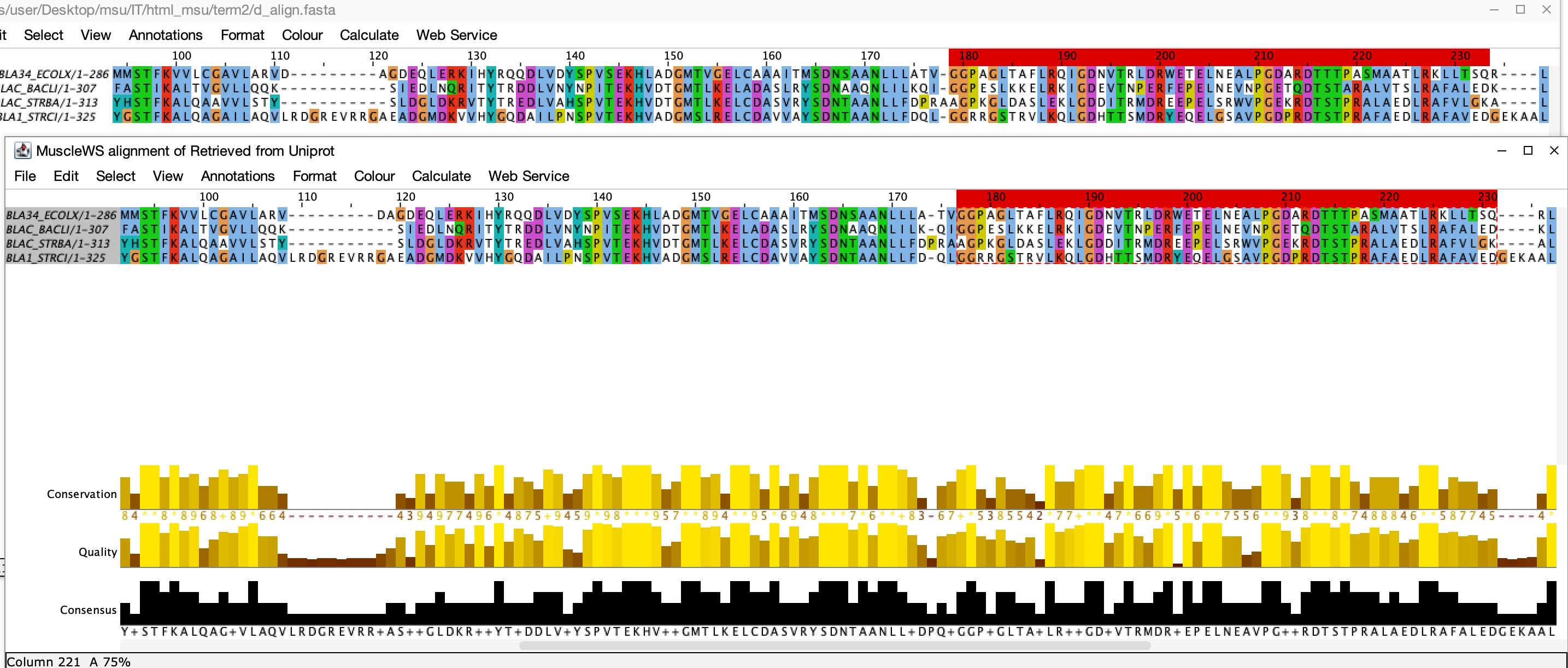
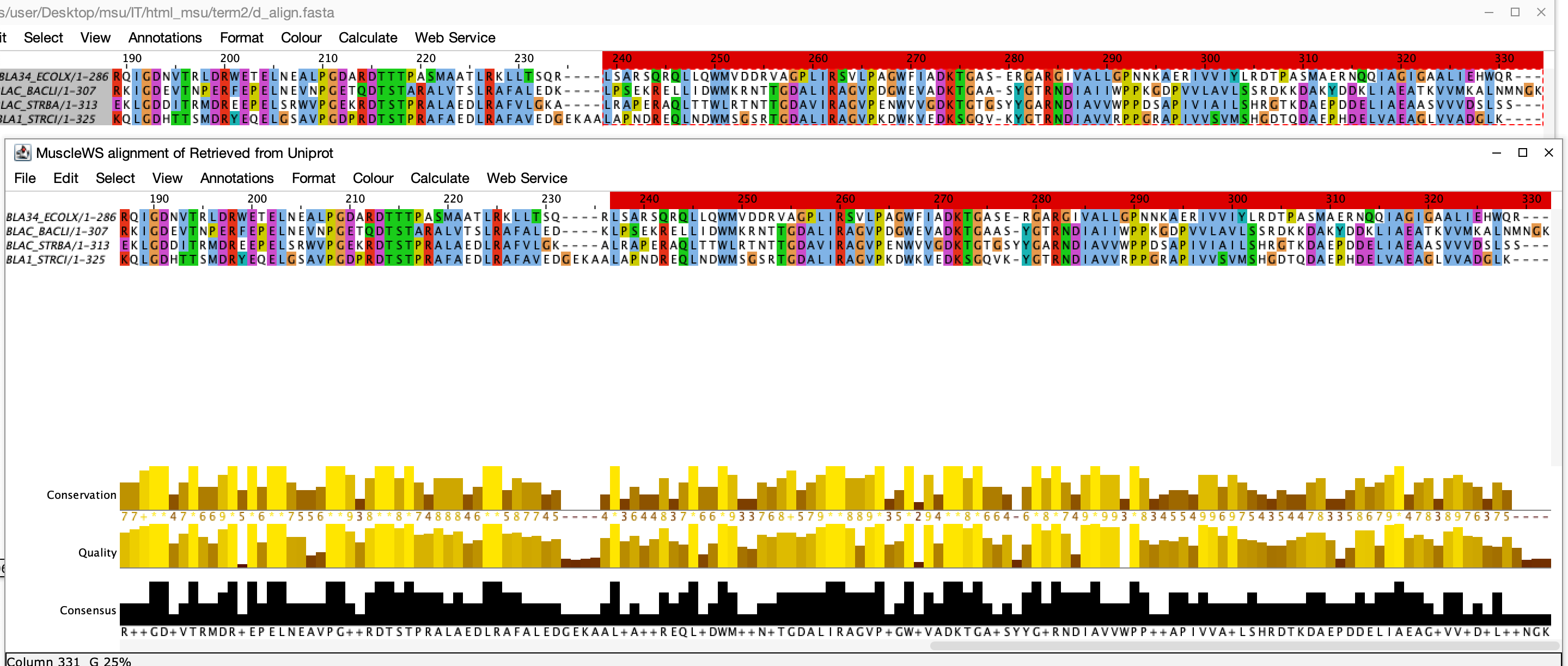
В целом все выравнивание совпадает, длина выравнивания с сайта Uniprot - 335, выравнивания программой Jalview - 332, отличия появляются из-за сдвига гэпов на 1-2 позиции, которые не должны сильноменять вес выравниваний. Единственным исключением являются первые 85 позиций верхнего и первые 84 позиции нижнего выравниваний, так как они очень сильно отличаются.
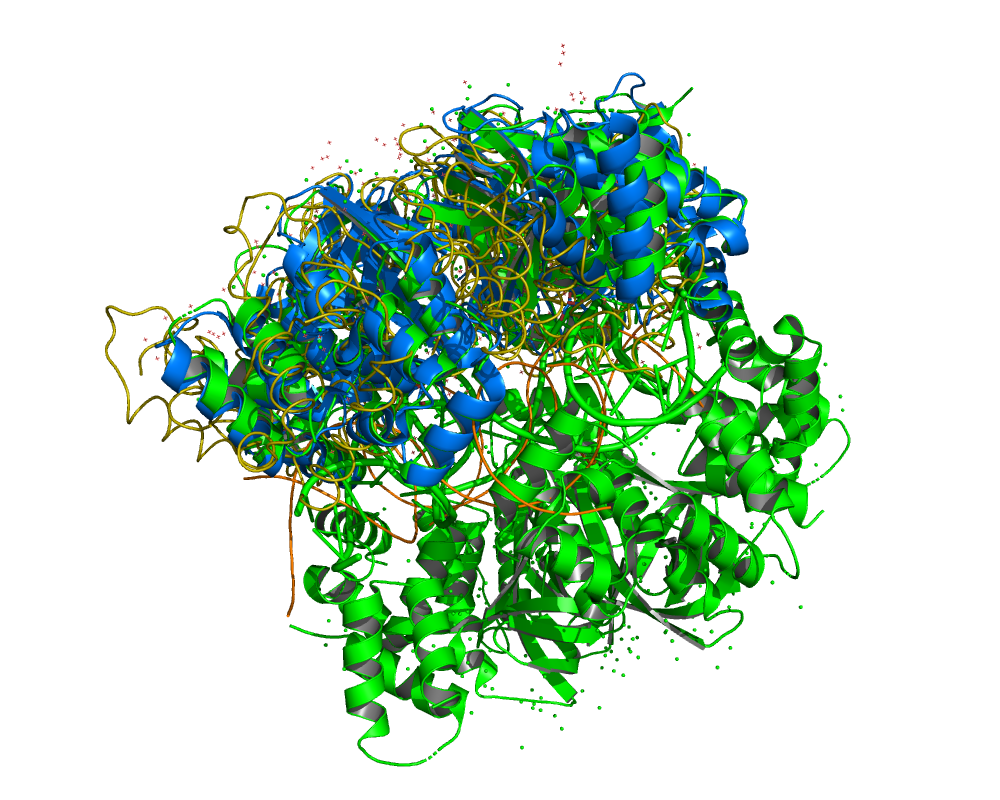
3D структуры
Я выбрала белки домена RuvA_N (ID:PF01330) с ID 1BDX, 7OA5 и 2H5X. Я выравнивала белки в pymol с помощью команды align. Ссылка на выравнивание (1BDX относится к RUVA_ECOLI, 7OA5 к RUVA_MYCLE, 2H5X к RUVA_MYCTU). На выравнивании видно чередование совпадающих и несовпадающих блоков, что заметно и на выравнивании в pymol.
Mufft
MAFFT - Multiple Alignment using Fast Fourier Transform (множественное выравнивание с использованием быстрого преобразования Фурье). Это программа для мноежственных выравниваний нуклеотидныйх и аминокислотных последовательностей, первую версию создал в 2002 Казутака Като. Алгорит состоит из 5 этапов:
- Попарное выравнивание - для каждой пары последовательностей определяются совпадающие участки.
- Матрица расстояний - на основе первого этапа рассчитываются "расстояния" между последовательностями и создается их матрица.
- Генеалогическое древо - на основе матрица расстояний создается древо, в котором отображается родство между организмами.
- Прогрессивное выравнивание - по методу прогрессивного выравнивания обрабатываюся все последовательности в древе по направлению от "корней" к "листьям".
- Итеративное выравнивание - процесс коректировки получившегося выравнивая для увеличения точности.
Список литературы:
- https://en.wikipedia.org/wiki/MAFFT
- https://en.wikipedia.org/wiki/Multiple_sequence_alignment#Progressive_alignment_construction
- Kazutaka Katoh, Kazuharu Misawa, Kei-ichi Kuma, Takashi Miyataa MAFFT: a novel method for rapid multiple sequence alignment based on fast Fourier transform Nucleic Acids Res. 2002 Jul 15; 30(14): 3059–3066. doi: 10.1093/nar/gkf436