Cборке de novo
Я скачала архив с помощью команды wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/000/SRR4240360/SRR4240360.fastq.gz. Далее я создала файле adapt.fa, в котором содержались все адаптеры из директории /mnt/scratch/NGS/adapters.
Чтобы убрать остатки адаптеров я воспользовалась командой TrimmomaticSE -phred33 SRR4240360.fastq.gz output.fq.gz ILLUMINACLIP:adapt.fa:2:7:7. Оказалось, что 41858 чтений (0,51%) отбор не прошли.
Затем с помощью команды TrimmomaticSE -phred33 output.fq.gz out_f.fq.gz TRAILING:20 MINLEN:32 я убрала чтения с длиной меньше 32 и качеством ниже 20. В итоге 297300 (3.62%) чтений было выкинуто, а файл стал весить 183,5 Мегабайт вместо 192,45.
Далее я использовала команду velveth ./velvet 31 -fastq.gz -short out_f.fq.gz, чтобы подготовить все к сборке. Я сохранила все созданные файлы в папке velvet. Затем я запустила сборку на основе k-меров с помощью команды velvetg ./velvet. В итоге N50 оказалось равно 43070, длина 3 самых длинных контигов (команда sort -k2,2n stats.txt) - 64155 (ID:4) с покрытием (прямое и обратное) 35.847323 и 35.847323,
83603 (ID:5) с покрытием 33.646065 и 33.646065 и 113474 (ID:1) с покрытием 33.525460 и 33.525460.
Что касается странных значений покрытий, то контиг с ID=173 имеет покрытия 134953.000000 и 134953.000000, при это есть 12 контигов с покрытием 1.
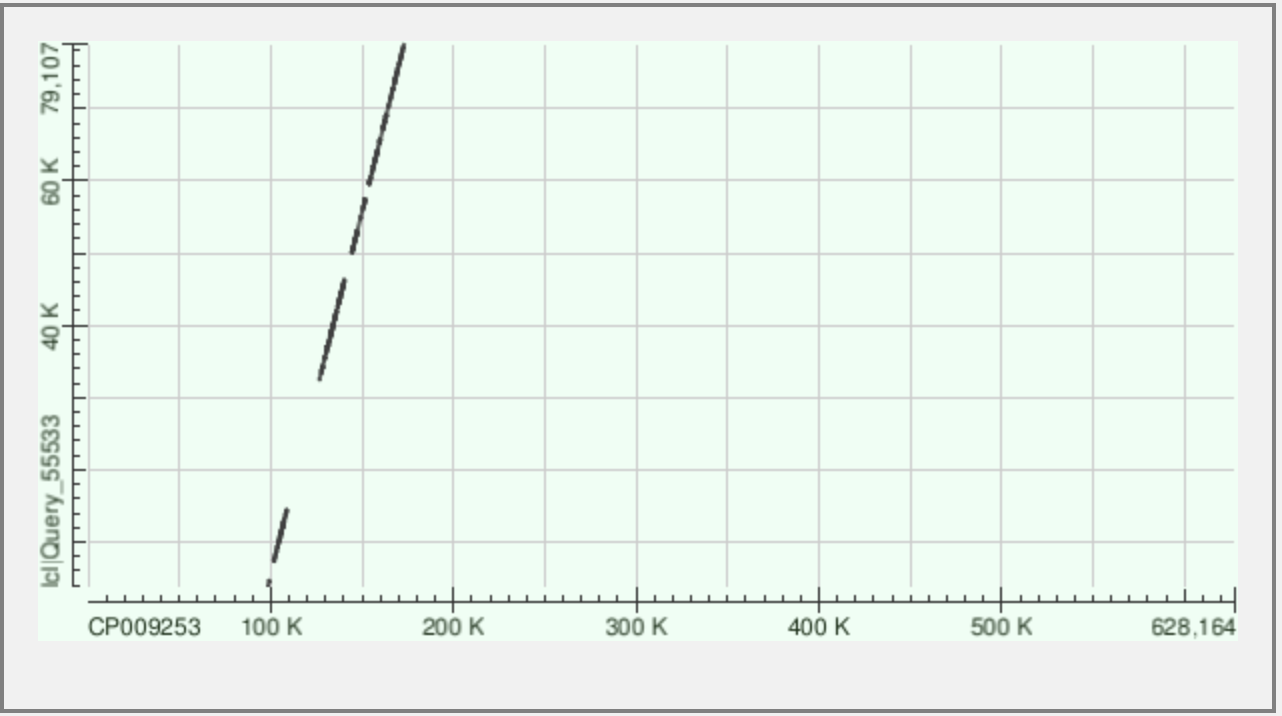
Я использовала алгоритм megablast, чтобы сравнить контиги 1, 4, 5 с Buchnera aphidicola (GenBank/EMBL AC — CP009253).
Для континга ID:1, участок континга 528794-550219 с 83427-104897 CP009253. Всего таких участков выравнялось 15.

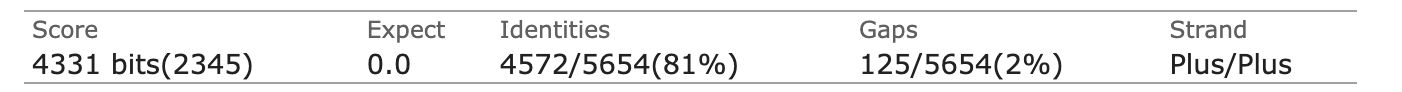
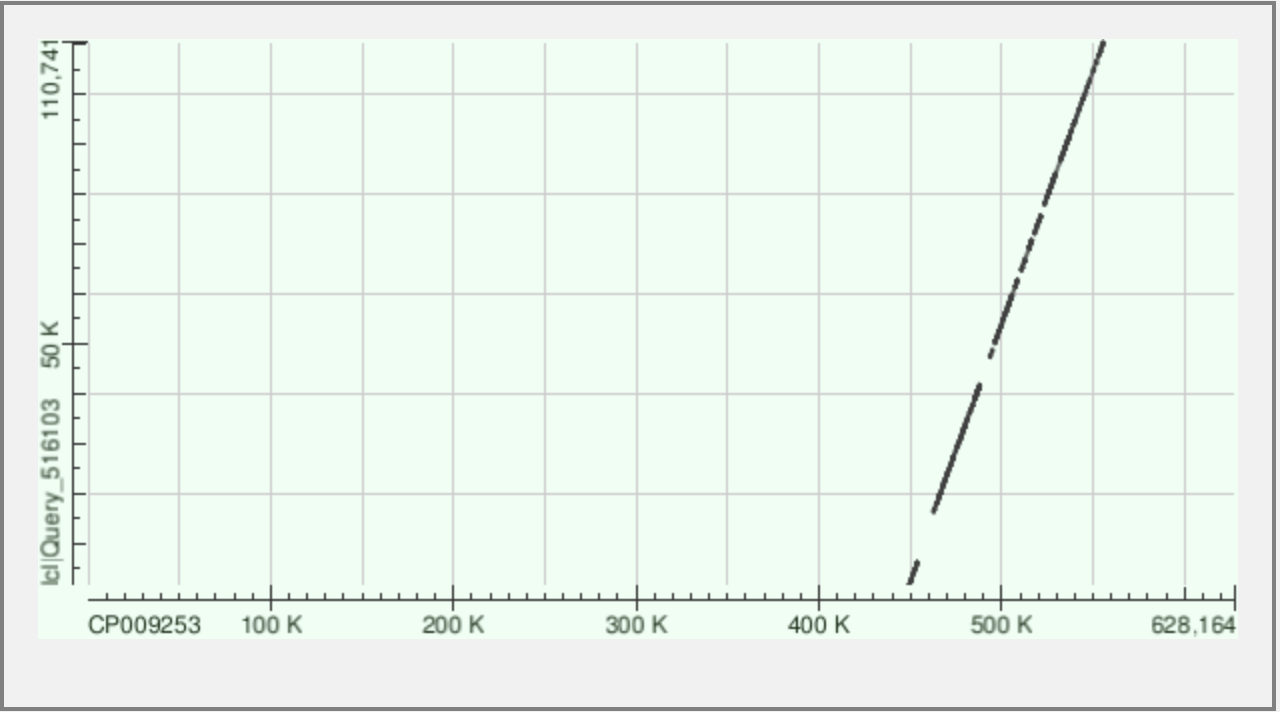
В результате для 4 контига получилось выравнивание 11 участков. Так фрагмент CP009253 2004-11103 выравнялся с фрагментом контига 31205-40294.


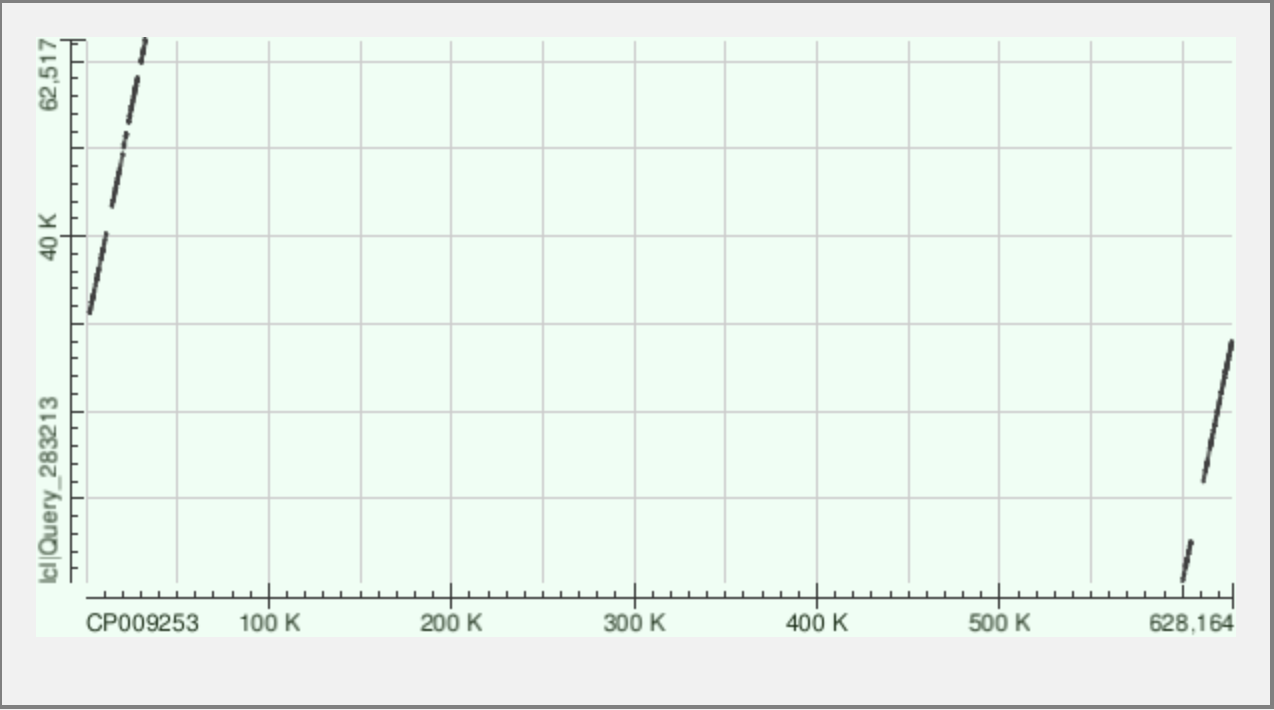
Для континга ID:5, участок континга 127825-140555 с 33725-46465 CP009253. Всего таких участков выравнялось 8.