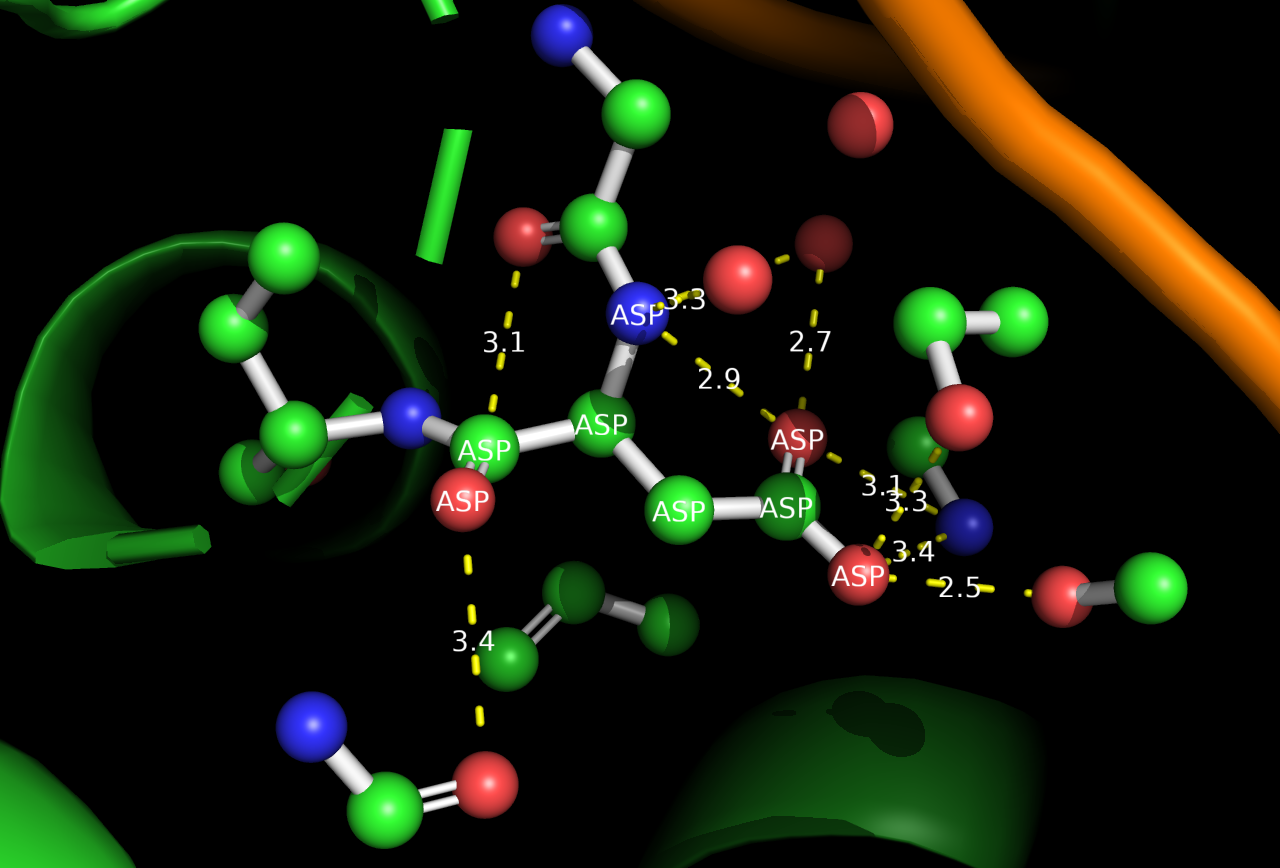
Задание 1
С помощью команды einverted -sequence 1F7V.seq -gap 12 -threshold 10 -match 3 -mismatch -3 -outfile outfile -outseq seqout я получила файл outseq, в котором содержалось:
: Score 15: 6/7 ( 85%) matches, 0 gaps
1 ttcctcg 7
|||| ||
72 aaggggc 66
В результате команды find_pair 1f7v.pdb fi.inp я получила:
1 72 0 # 1 | ....>B:.901_:[PSU]P-**--A[..A]:.972_:B<.... 0.84 0.30 15.36 8.88 2.21
2 71 0 # 2 | ....>B:.902_:[..U]U-----A[..A]:.971_:B<.... 0.28 0.16 9.22 8.76 -3.95
3 70 0 # 3 | ....>B:.903_:[..C]C-----G[..G]:.970_:B<.... 0.31 0.15 10.84 8.78 -3.85
4 69 0 # 4 | ....>B:.904_:[..C]C-----G[..G]:.969_:B<.... 0.47 0.25 5.66 8.85 -3.76
5 68 0 # 5 | ....>B:.905_:[..U]U-*---G[..G]:.968_:B<.... 2.35 0.09 12.19 8.81 0.14
6 67 0 # 6 | ....>B:.906_:[..C]C-----G[..G]:.967_:B<.... 0.39 0.17 16.03 8.68 -3.46
7 66 0 # 7 | ....>B:.907_:[..G]G-----C[..C]:.966_:B<.... 0.28 0.06 10.65 8.82 -4.06
49 65 0 # 8 | ....>B:.949_:[5MC]c-----G[..G]:.965_:B<.... 0.88 0.35 10.78 8.86 -2.88
50 64 0 # 9 | ....>B:.950_:[..C]C-----G[..G]:.964_:B<.... 0.20 0.11 7.54 8.87 -4.21
51 63 0 # 10 | ....>B:.951_:[..A]A-----U[..U]:.963_:B<.... 0.24 0.14 4.54 8.90 -4.25
52 62 0 # 11 | ....>B:.952_:[..G]G-----C[..C]:.962_:B<.... 0.39 0.14 7.57 8.80 -3.94
53 61 0 # 12 | ....>B:.953_:[..G]G-----C[..C]:.961_:B<.... 0.23 0.14 18.16 8.88 -3.58
54 58 0 # 13 | ....>B:.954_:[5MU]t-**--a[1MA]:.958_:B<.... 4.56 0.06 9.94 7.29 2.17
55 17 9 # 14 x ....>B:.955_:[PSU]P-**+-G[..G]:.917_:B<.... 5.37 0.84 27.67 8.17 8.44
39 31 0 # 15 | ....>B:.939_:[..C]C-----G[..G]:.931_:B<.... 0.41 0.05 7.53 8.75 -4.11
40 30 0 # 16 | ....>B:.940_:[..C]C-----G[..G]:.930_:B<.... 0.24 0.20 7.90 8.95 -3.96
41 29 0 # 17 | ....>B:.941_:[..A]A-----U[..U]:.929_:B<.... 0.15 0.14 2.91 8.80 -4.42
42 28 0 # 18 | ....>B:.942_:[..G]G-----C[..C]:.928_:B<.... 0.45 0.25 15.68 8.71 -3.27
43 27 0 # 19 | ....>B:.943_:[..A]A-**--P[PSU]:.927_:B<.... 0.40 0.02 28.12 8.65 1.85
44 26 0 # 20 | ....>B:.944_:[..A]A-*---g[M2G]:.926_:B<.... 2.83 0.01 32.83 10.46 4.50
10 25 0 # 21 | ....>B:.910_:[2MG]g-----C[..C]:.925_:B<.... 0.79 0.04 11.39 8.69 -3.56
11 24 0 # 22 | ....>B:.911_:[..C]C-----G[..G]:.924_:B<.... 0.56 0.33 3.09 8.83 -3.64
12 23 0 # 23 | ....>B:.912_:[..C]C-----G[..G]:.923_:B<.... 0.33 0.14 8.25 8.89 -3.98
13 22 0 # 24 | ....>B:.913_:[..C]C-**--C[..C]:.922_:B<.... 4.86 0.14 12.67 8.22 4.77
14 8 0 # 25 | ....>B:.914_:[..A]A-**--U[..U]:.908_:B<.... 4.81 0.51 11.97 7.11 3.43
15 48 9 # 26 x ....>B:.915_:[..A]A-**+-U[..U]:.948_:B<.... 1.36 0.06 16.07 8.93 -0.71
18 56 1 # 27 + ....>B:.918_:[..G]G-----C[..C]:.956_:B<.... 0.13 0.08 35.01 8.69 -2.96
Для алгоритма Зумера я использовала сайт. В результате я получила:
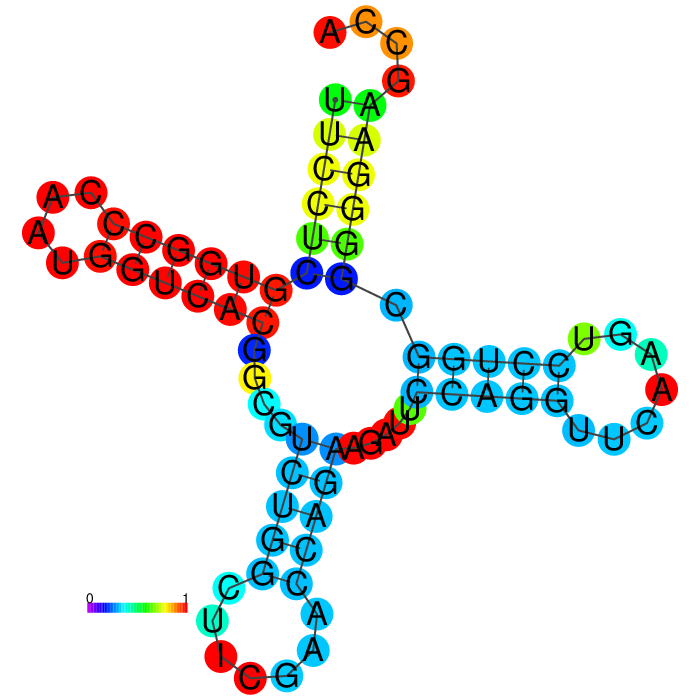
| Участок структуры | find_pair | einverted | А. Зукера |
|---|---|---|---|
| Акцепторный стебель | 6 пар | 6 пар | 6 пар |
| D-стебель | 4 пары | - | 6 пар |
| T-стебель | 4 пары | - | 5 пар |
| Антикодоновый стебель | 4 пары | - | 5 пар |
| Общее число канонических пар нуклеотидов | 18 пар | 6 пар | 22 пары |
Задание 2
Упражнение 1

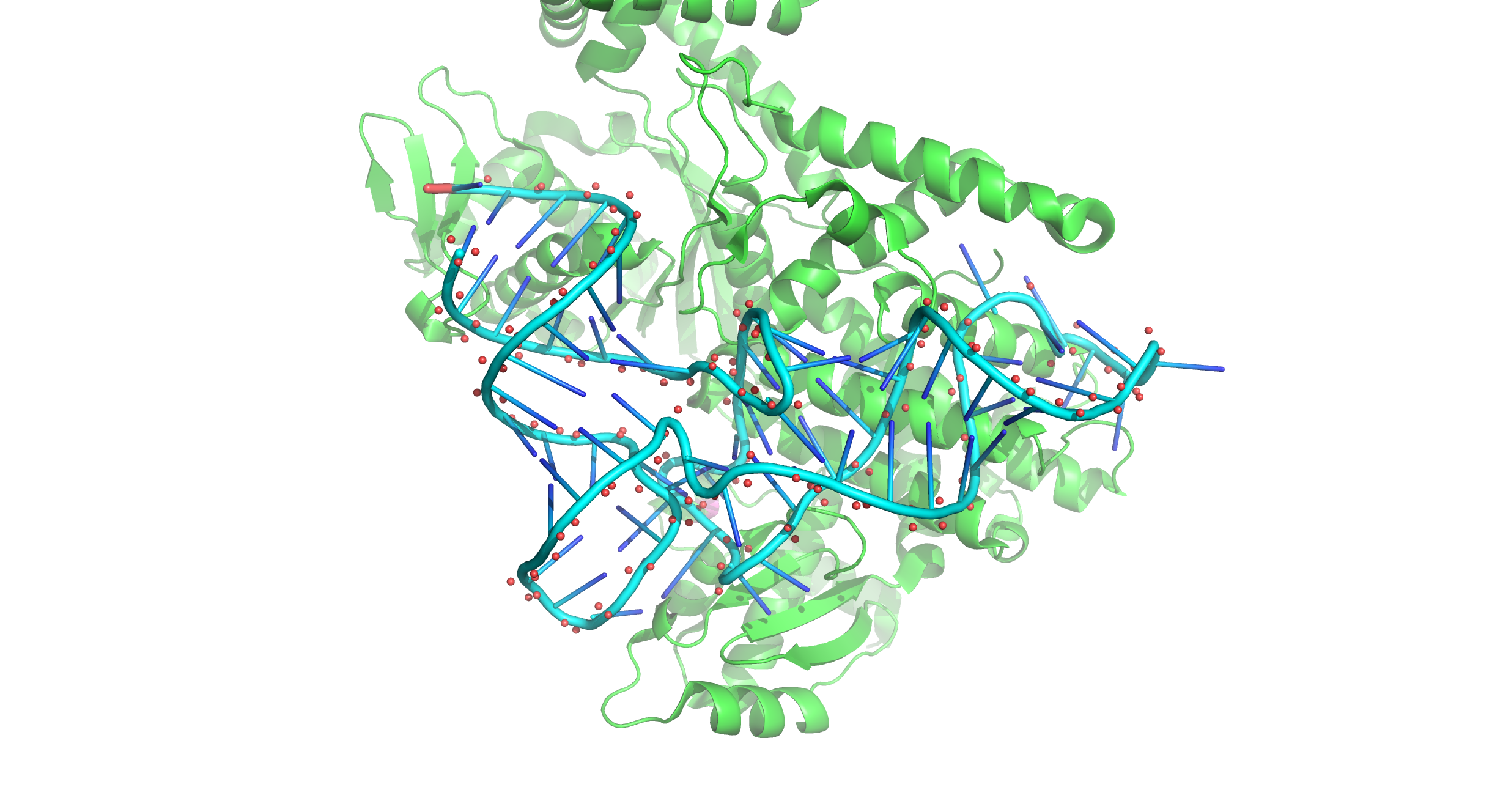
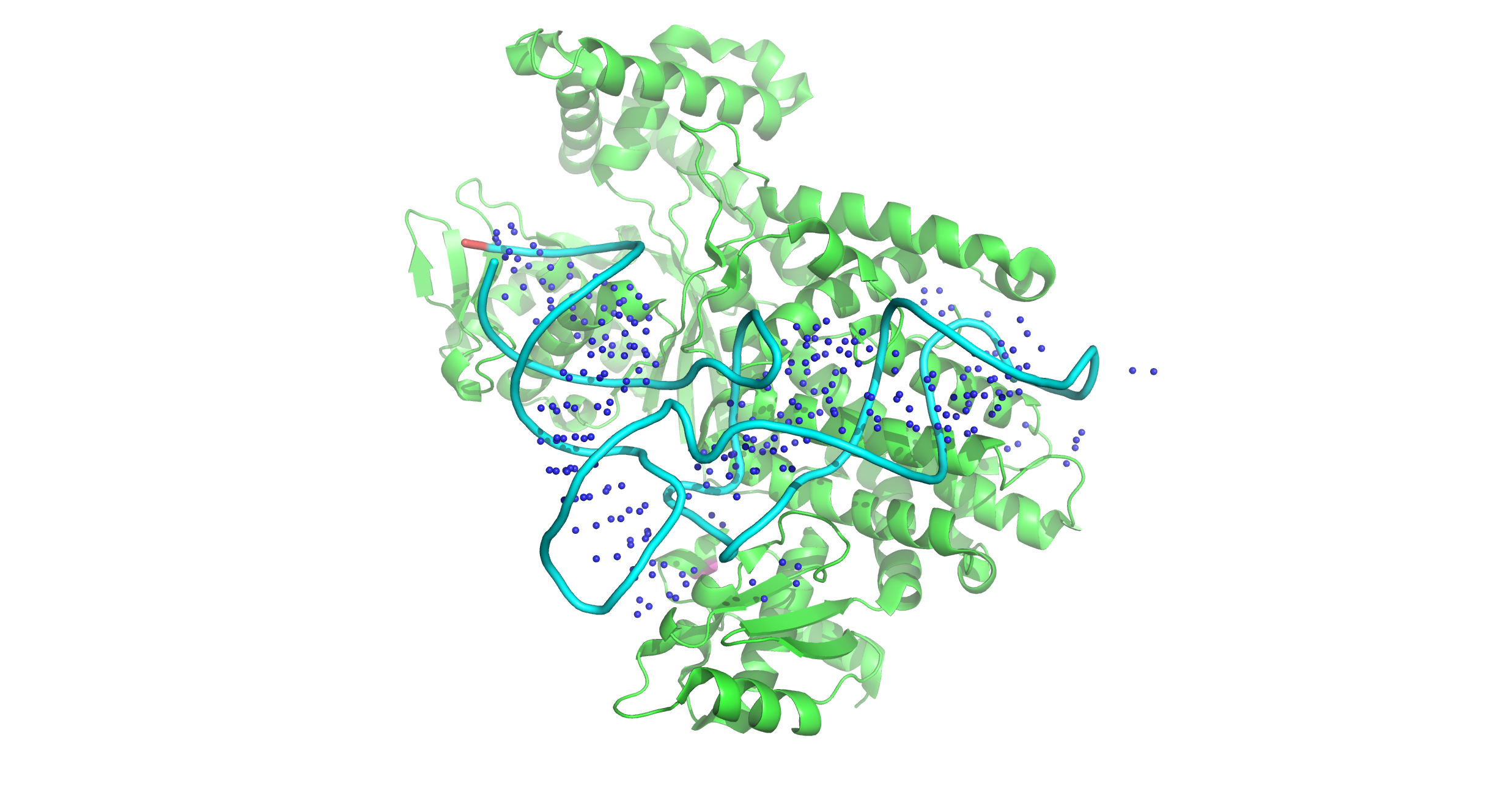
Скрипт-файл с определениями этих множеств.
Упражнение 2
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 9 | 51 | 60 |
| остатками фосфорной кислоты | 14 | 13 | 27 |
| остатками азотистых оснований со стороны большой бороздки | 5 | 0 | 5 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 5 | 5 |
Упражнение 3
Файл с белковыми контактами.
Аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК - Gly483, связанный с 2 основаниями и 1 фосфатом.
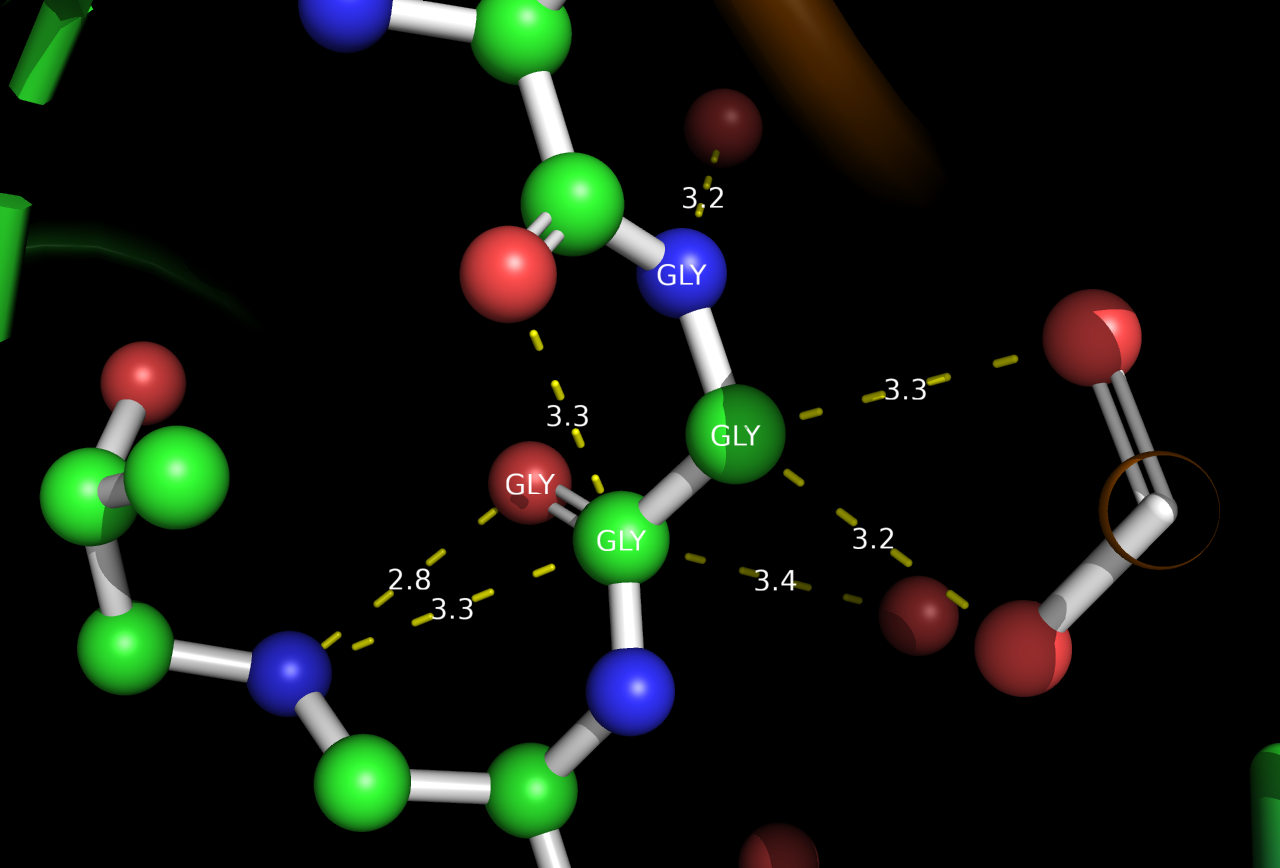
Аминокислотный остаток, по-вашему мнению, наиболее важный для распознавания последовательности ДНК можно назвать Asp484, так как он связан с 2 основаниями 4 nonbonded contacts.