Секвенирование по Сэнгеру:
1) Я работала с файлом 05_F.ab1 для второй группы в программе "Codon Code Aligner".
2) Длина моей хроматограммы составляет 905 оснований.
3) Прямая цепь: начальный трудночитаемый фрагмент - 31 основание, конечный - 183 основания. Обратная цепь: начальный трудночитаемый фрагмент - 194 основания, конечный - 33 основания.
4) Если исключить начальный и конечный трудночиттаемые фрагменты, то соотношение основных пиков и шума примерно 2 к 1 для обеих цепей.
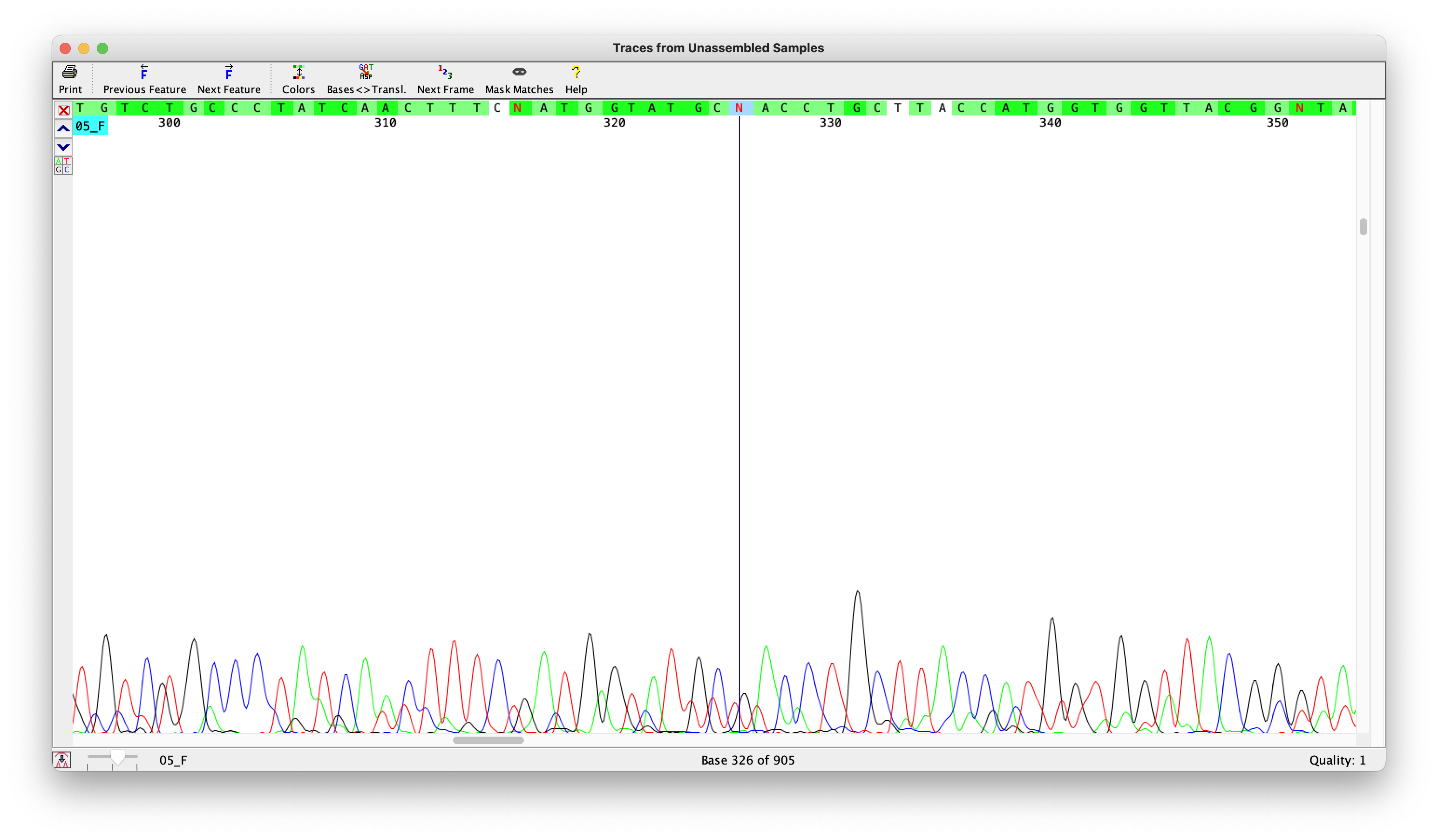
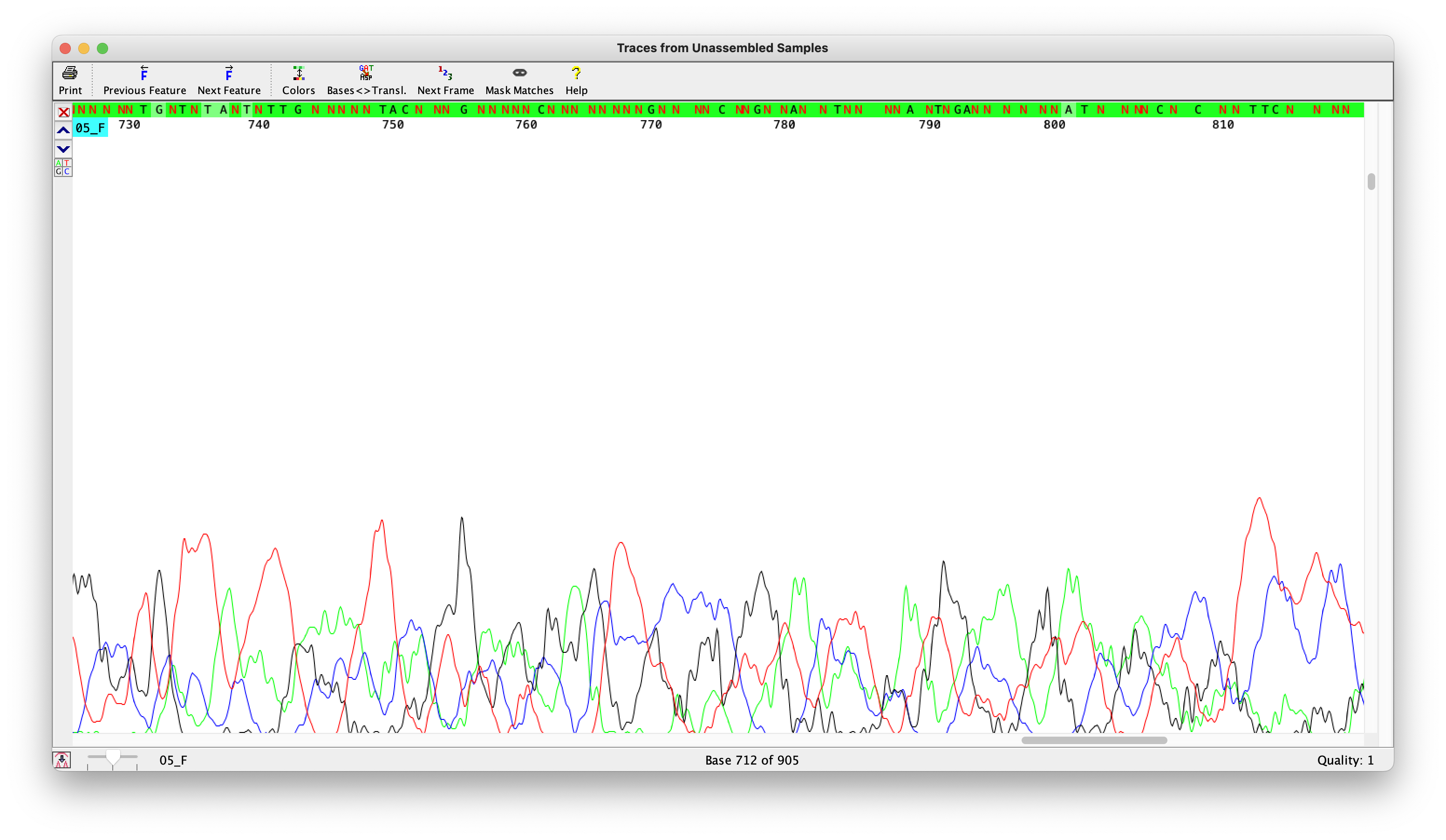
5) Шума почти нет:
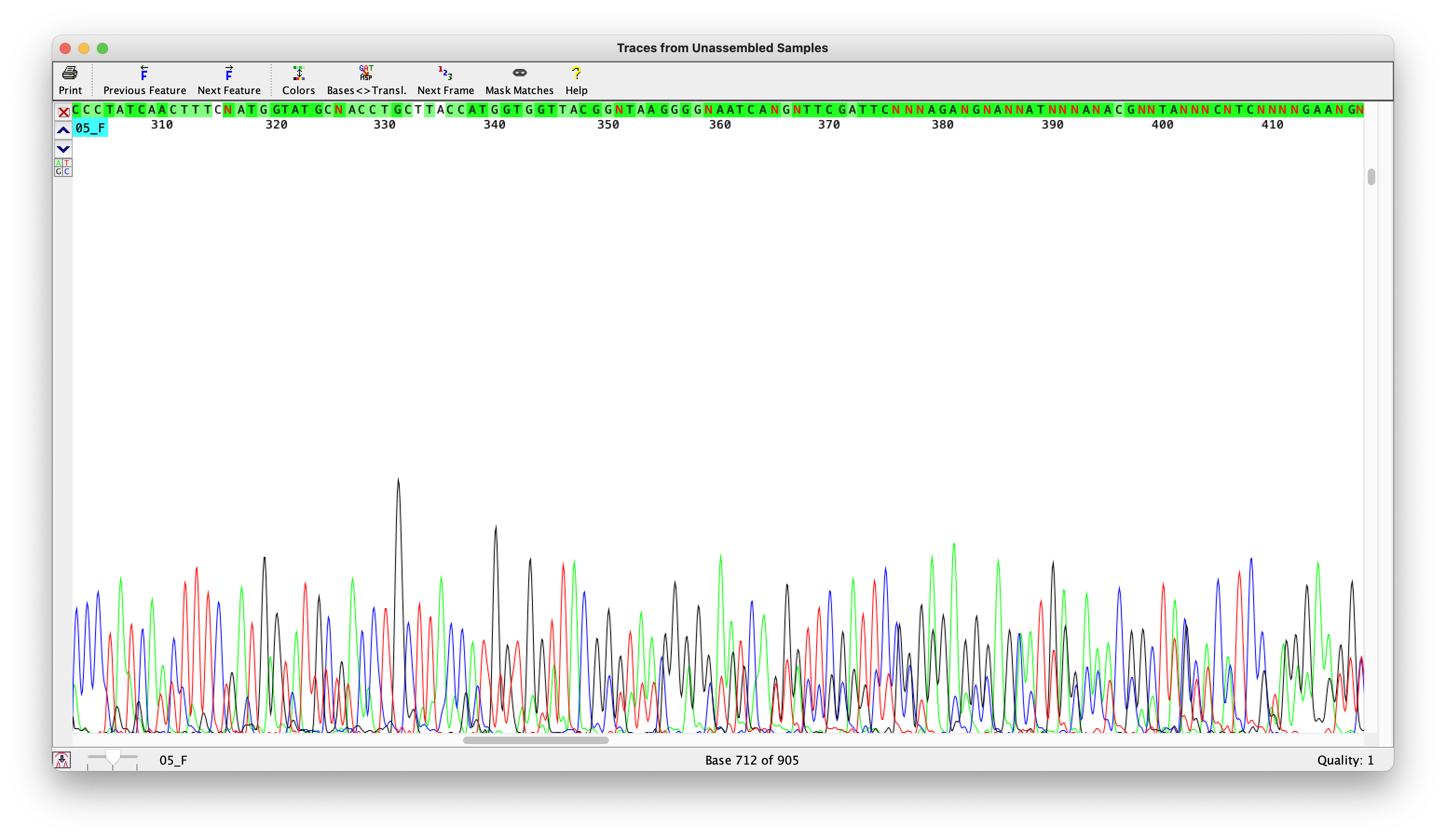
Шум мешает интерпретации сигнала
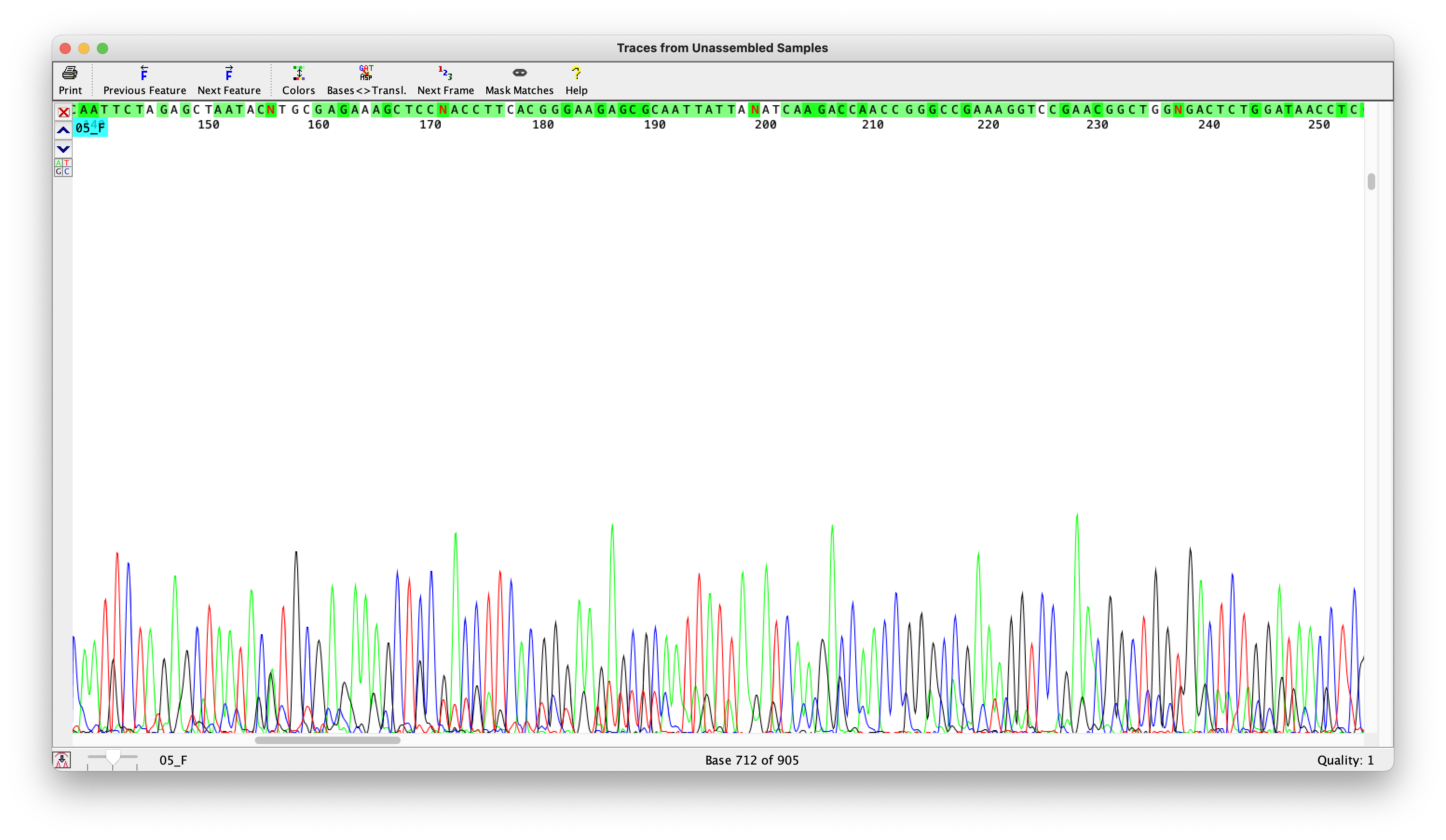
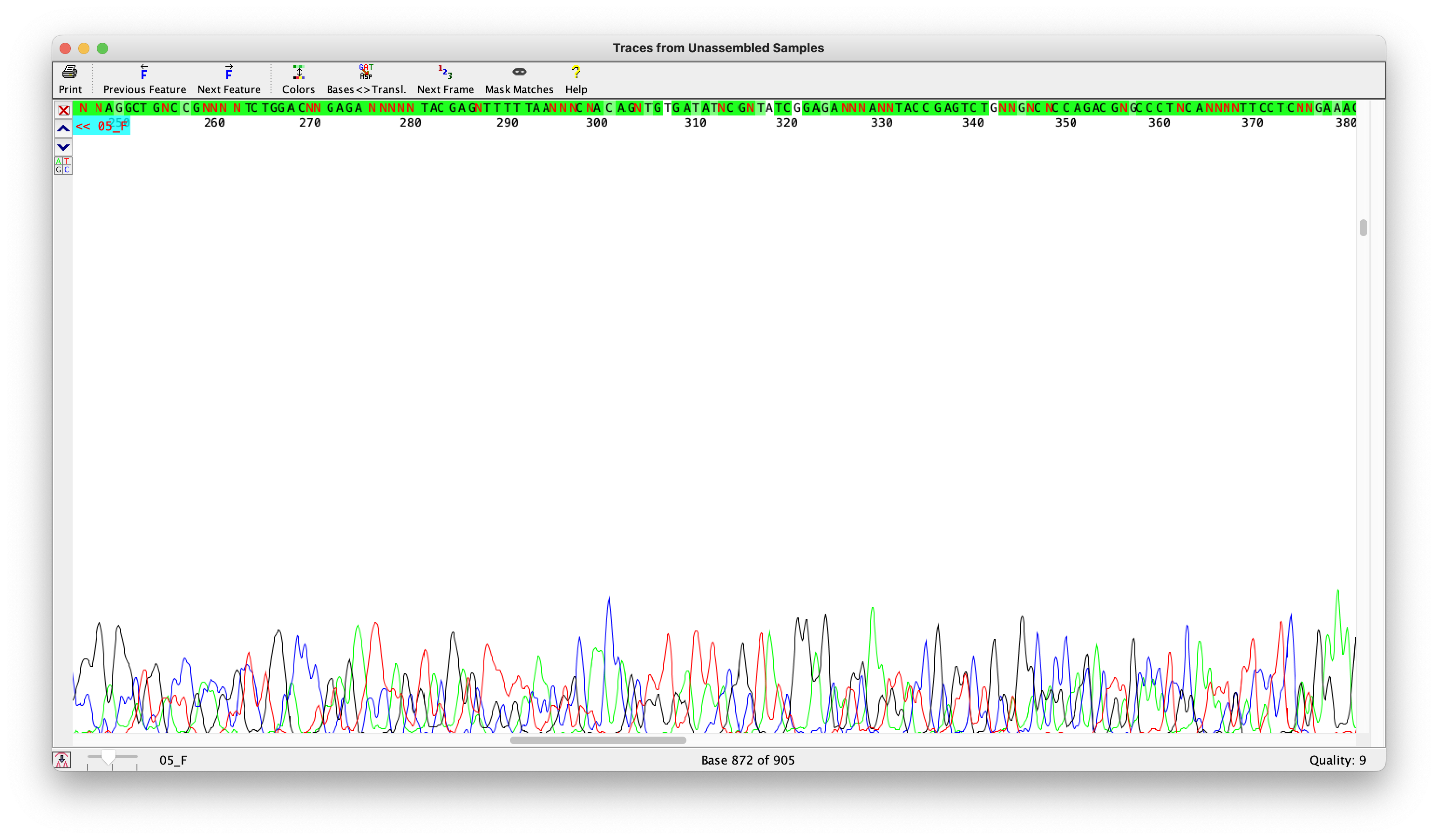
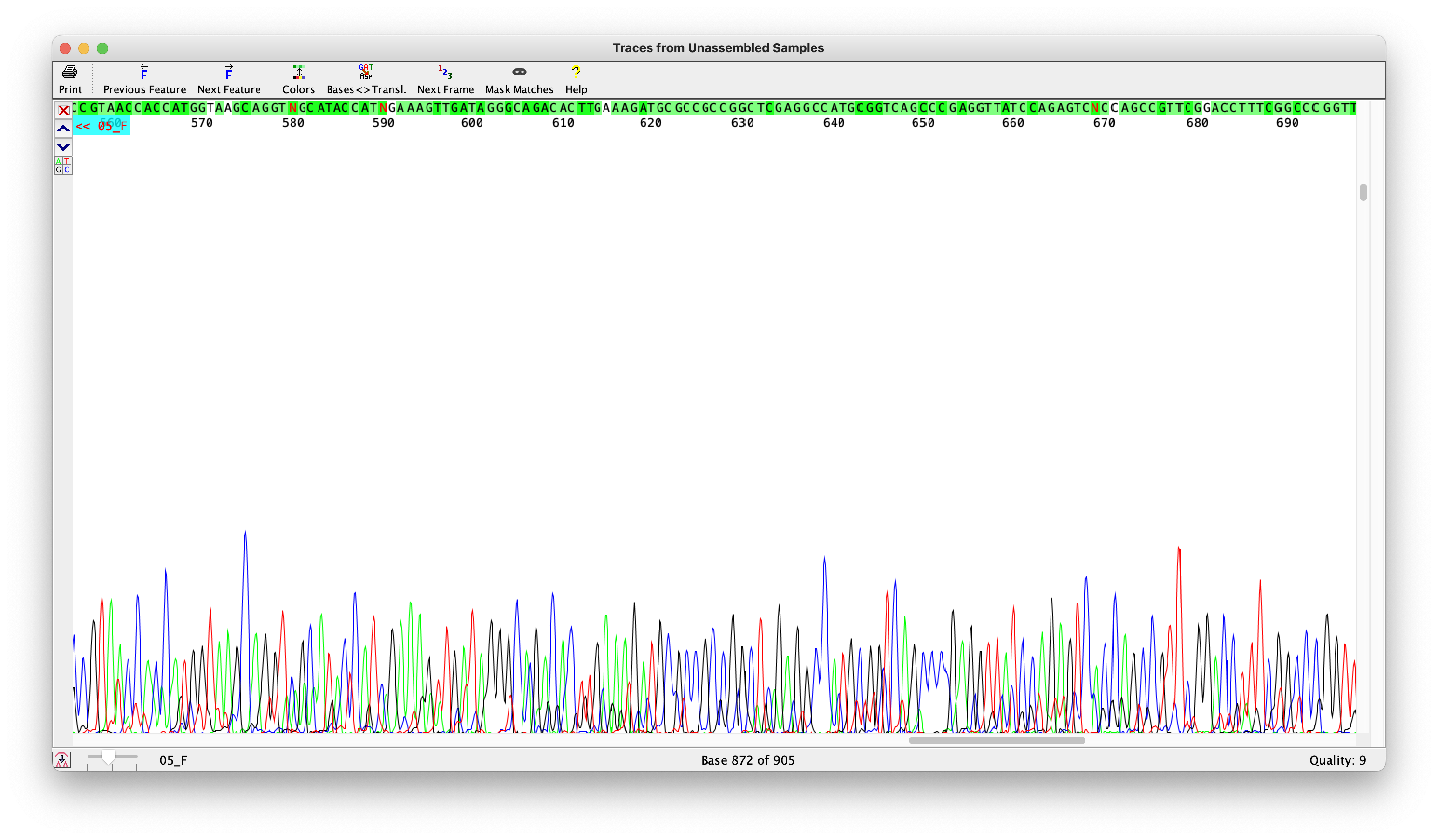
Шум есть, но не мешает интерпретации сигнала

6) Фотогорафии прямой цепи