Мотивы в белках
Консервативные мотивы в выравнивании
Для этого практикума я выбрала домен SH2, последовательностей между онкопротеинами Src и Fps. PF00017. Seed содержит 52 белков. Для исследования мотива я выбрала последовательность G.FL[VIAL]R.S, всего таких мотив оказалось 32. Паттерн в формате Prosite: G-x-F-L-[VIAL]-R-x-S.

Пропустив этот мотив через MyHits я нашла 335 совпадений, что означает, что такой паттерн не является характерной чертой выбранного домена.
Мотив, специфичный для одной клады филогенетического дерева
Я выбрала кладу из 15 последовательностей. В ней я нашла консервативный паттерн KHY.I, который был у 13 последовательностей. При этом при поиске в белках, не входящих в выбранную кладу совпадений не нашлось, что означает, что этот паттерн характерен только для этих 15 последовательностей.

PSI-BLAST
Выбрала индентификатор P39450, это S-(гидроксиметил)глутатиондегидрогеназа Pasteurella piscicida, которая катализирует синтез S-формилглутатиона.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 467 | Q9ZGI4.1 | 0,005 | D2S1F7.1 | 0,006 |
| 2 | 836 | Q5KWK4.1 | 0,005 | Q2RKY6.1 | 0,006 |
| 3 | 949 | P55100.2 | 0,005 | D1ZA70.1 | 0,006 |
| 4 | 1613 | B2AH07.1 | 0,005 | O01592.1 | 0,006 |
| 5 | 2897 | B5YEM1.1 | 0,005 | B5BL07.1 | 0,006 |
Поиск de novo мотивов в выборке поледовательностей с доменом из SwissProt
Я провела поиск своего домена в Swiss-Prot. Я решила использовать только белки человека, поэтому последовательностей осталось 100. После remove redundancy на 30% осталось 89. Далее я запустила MEME и FIMO с помощью команд:
meme def.fasta -o meme_results -mod anr -minw 4 -maxw 8 -nmotifs 4
fimo meme_results/meme.txt uniprotkb_PF00017_AND_organism_id_9606_2024_05_16.fasta
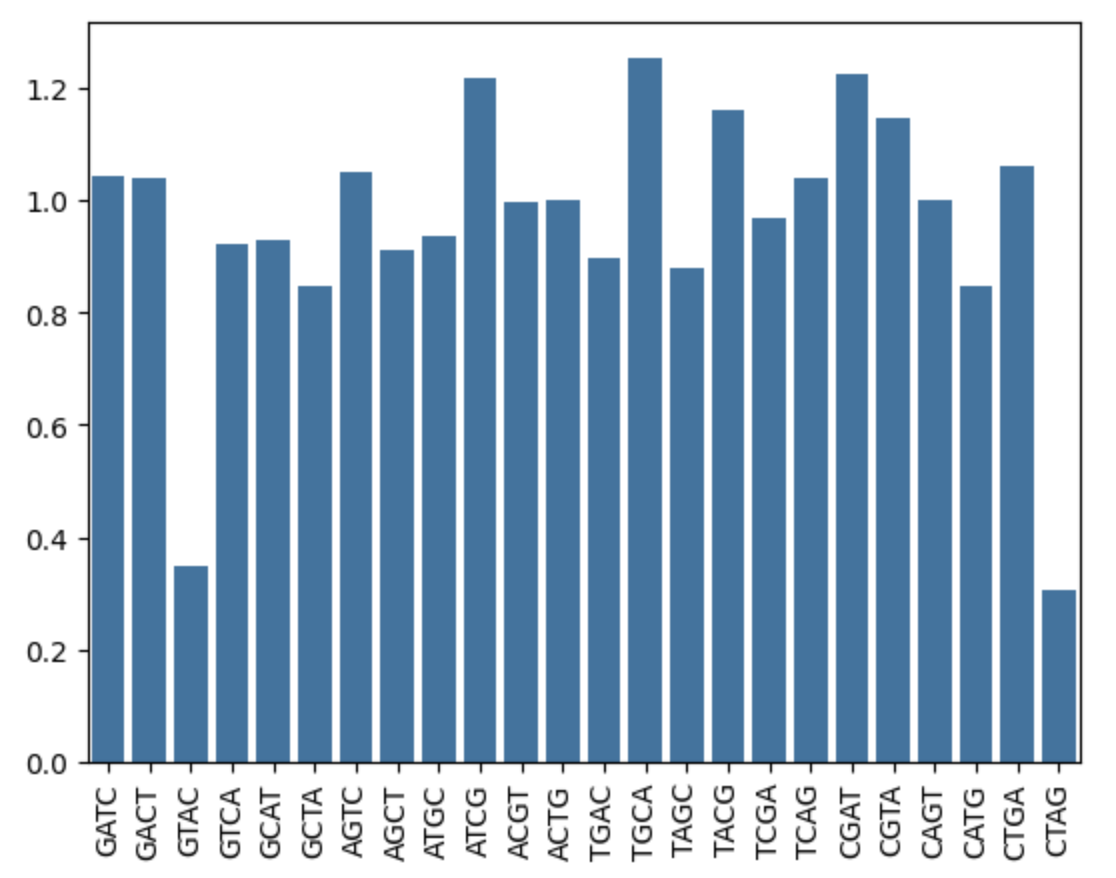
Представленность сайта GATC в геноме
Взяла в качетсве бактерии Escherichia coli. Далее с помощью скрипта Каримовой Каримы я подсчитала контрасты obs/exp по методу Карлина для сайта всех сайтов длиной 4 нуклеотида.