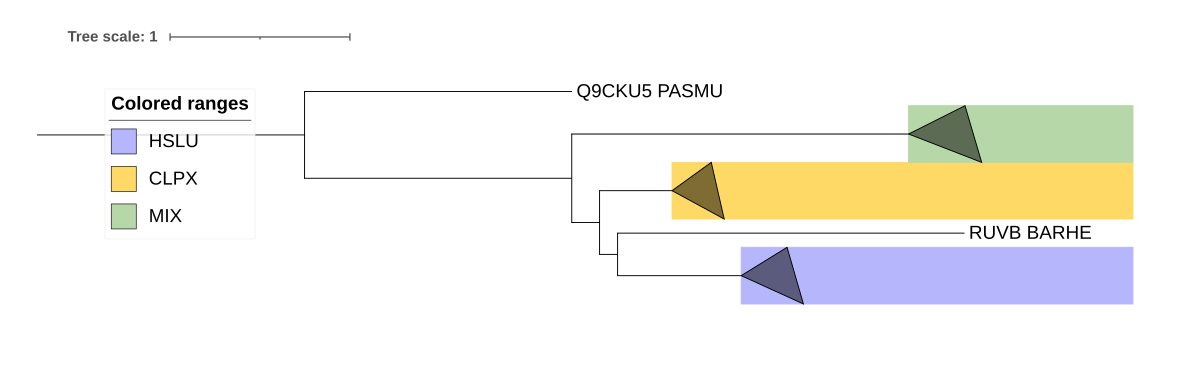Составление списка гомологичных белков, включающих паралоги
Для поиска достоверных гомологов белка CLPX_ECOLI с порогом E-value = 0,0005 я использовала скачанный протеомы из директории /P/y22/term4/Proteomes и следующий команды:
cat PASMU.fasta HAEIN.fasta SACD2.fasta BARHE.fasta BRUSU.fasta AGRFC.fasta > ~/term4/pr4/proteomes.fasta - копирование последовательностей в один файл.
makeblastdb -dbtype prot -in proteomes.fasta -out myprot_db - создание базы данных из последовательностей белков.
blastp -db myprot_db -query CLPX_ECOLI.fasta -out homologs -evalue 0.0005 - поиск гомологов белка CLPX_ECOLI в созданной базе данных.
В результате я получила 19 результатов:
Query= sp|P0A6H1|CLPX_ECOLI ATP-dependent Clp protease ATP-binding subunit
ClpX OS=Escherichia coli (strain K12) OX=83333 GN=clpX PE=1 SV=2
Length=424
Score E
Sequences producing significant alignments: (Bits) Value
sp|Q21KA8|CLPX_SACD2 ATP-dependent Clp protease ATP-binding subun... 645 0.0
sp|P57981|CLPX_PASMU ATP-dependent Clp protease ATP-binding subun... 612 0.0
sp|Q8UFY5|CLPX_AGRFC ATP-dependent Clp protease ATP-binding subun... 596 0.0
sp|Q6G3Z2|CLPX_BARHE ATP-dependent Clp protease ATP-binding subun... 588 0.0
sp|P44838|CLPX_HAEIN ATP-dependent Clp protease ATP-binding subun... 587 0.0
sp|Q8G0I5|CLPX_BRUSU ATP-dependent Clp protease ATP-binding subun... 586 0.0
sp|Q21H71|HSLU_SACD2 ATP-dependent protease ATPase subunit HslU O... 99.8 4e-23
sp|Q6G5G0|HSLU_BARHE ATP-dependent protease ATPase subunit HslU O... 97.1 3e-22
sp|P57968|HSLU_PASMU ATP-dependent protease ATPase subunit HslU O... 93.2 7e-21
sp|Q8UJ87|HSLU_AGRFC ATP-dependent protease ATPase subunit HslU O... 92.0 1e-20
sp|P43773|HSLU_HAEIN ATP-dependent protease ATPase subunit HslU O... 92.0 2e-20
sp|Q8FY12|HSLU_BRUSU ATP-dependent protease ATPase subunit HslU O... 90.9 3e-20
tr|A0A0H3GCZ6|A0A0H3GCZ6_BRUSU ATP-dependent zinc metalloprotease... 45.4 2e-05
tr|A0A0H3LXZ4|A0A0H3LXZ4_BARHE ATP-dependent zinc metalloprotease... 45.4 2e-05
tr|Q7CT50|Q7CT50_AGRFC ATP-dependent zinc metalloprotease FtsH OS... 45.4 3e-05
sp|Q6G5R1|RUVB_BARHE Holliday junction branch migration complex s... 43.5 7e-05
tr|Q9CNJ2|Q9CNJ2_PASMU ATP-dependent zinc metalloprotease FtsH OS... 43.1 1e-04
sp|P71377|FTSH_HAEIN ATP-dependent zinc metalloprotease FtsH OS=H... 42.7 1e-04
tr|Q9CKU5|Q9CKU5_PASMU ComM OS=Pasteurella multocida (strain Pm70... 41.6 4e-04
| Бактерия | Количество совпалений | Гомологичные белки |
|---|---|---|
| PASMU | 4 | CLPX_PASMU, HSLU_PASMU, Q9CNJ2_PASMU, Q9CKU5_PASMU |
| HAEIN | 3 | CLPX_HAEIN, HSLU_HAEIN, FTSH_HAEIN |
| SACD2 | 2 | CLPX_SACD2, HSLU_SACD2 |
| BARHE | 4 | CLPX_BARHE, HSLU_BARHE, A0A0H3LXZ4_BARHE, RUVB_BARHE |
| BRUSU | 3 | CLPX_BRUSU, HSLU_BRUSU, A0A0H3GCZ6_BRUSU |
| AGRFC | 3 | CLPX_AGRFC, HSLU_AGRFC, Q7CT50_AGRFC |
Реконструкция и визуализация
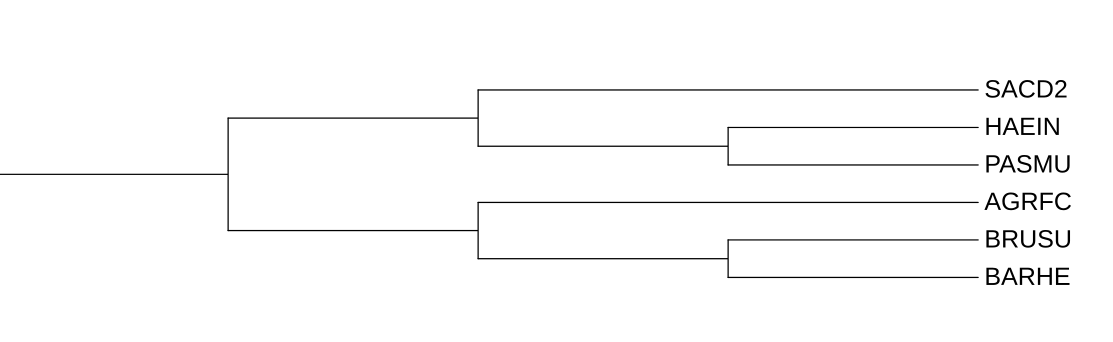
Я создала файл из последовательностей белков-гомологов и посторила по ним дерево. Дерево имело следующую формулу.

Ортологи и паралоги
Ортологи:HSLU_HAEIN и HSLU_SACD2, CLPX_AGRFC и CLPX_BRUSU, A0A0H3GCZ6_BRUSU и A0A0H3LXZ4_BARHE.
Паралоги:CLPX_HAEIN и FTSH_HAEIN, HSLU_BARHE и A0A0H3LXZ4_BARHE, Q7CT50_AGRFC и HSLU_AGRFC.
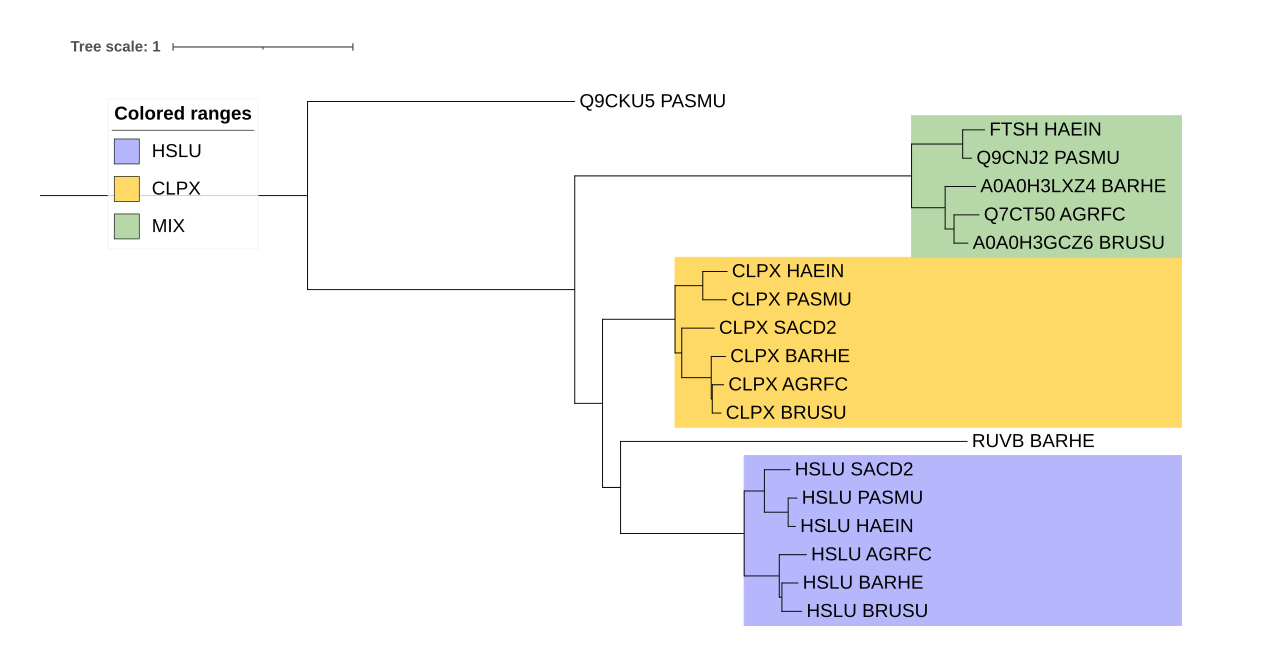
| Группа HSLU | Группа CLPX | Группа MIX |
|---|---|---|
| Филогенетическое дерево белков полностью совпадает с древом бактерий | AGRFC ближе к BRUSU, чем BARHE, SACD2 отделяется от HAEIN и PASMU раньше чем в филогении бактерий | SACD2 отсутствует в этой кладе, AGRFC ближе к BRUSU, чем BARHE |
Группа HSLU - группа АТФазных субъединиц АТФ-зависимых протеаз. Связывание АТФ и его последующий гидролиз HslU необходимы для разворачивания белковых субстратов, впоследствии гидролизуемых HslV. HslU распознает N-концевую часть своих белковых субстратов и разворачивает их, прежде чем они направляются к HslV для гидролиза.
Группа CLPX - группа АТФ-зависимых компонентов специфичности протеазы Clp. Используют циклы связывания и гидролиза АТФ для разворачивания белков и их переноса в протеазу ClpP.
Группа MIX - группам АТФ-зависимых цинк металлопротеаз. Играют роль в контроле качества интегральных мембранных белков. Кроме отсутствующей бактерии в этой кладе, есть 1 отличие от филогенетического древа бактерий: AGRFC ближе к BRUSU, чем BARHE