Сравнение предсказаний трансмембранных участков в бета-листовом белке
Я выбрала белок Serine protease EspP (Сериновая протеаза EspP). Это белок Escherichia coli, расщепляющий пепсин А и человеческий фактор свертывания крови, из-за чего происходит кровоизлияние в слизистые оболочки. Последовательность белка я взяла со страницы Uniprot.
Class: Beta-barrel transmembrane
Family: Autotransporters of N-terminal passenger domain
Localization: Наружная мембрана грамотрицательных бактерий
ID Uniprot: ESPP_ECO57
ID PDB: 3SLJ
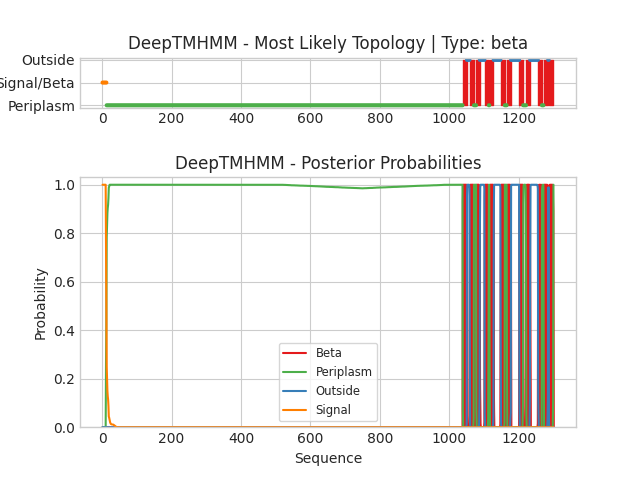
Я использовала сайт DeepTMHMM, чтобы определить траснмембранные участки.
Траснмембранные участки: OPM; DTU
1: 1039-1047| 1039-1050
2: 1061-1071| 1059-1070
3: 1077-1086| 1078-1087
4: 1103-1111| 1102-1113
5: 1118-1126| 1116-1127
6: 1149-1159| 1148-1159
7: 1167-1176| 1166-1177
8: 1202-1214| 1202-1212
9: 1221-1231| 1222-1232
10: 1258-1268| 1256-1266
11: 1272-1280| 1272-1284
12: 1290-1298| 1287-1299
Как видно, предсаказание не совсем точное, отличается на 1-2 аминокислоты. Эти отличия могут быть связаны с наличием гидрофобных аминоксилот за пределами траснмембранного участка, что может быть связано с функциями белка. Файл с результатами лежит здесь.
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Мне был выдан белок Экспортный белок стафилопин (Staphylopine export protein) с ID: CNTE_STAAM, как ясно из названия этот белок участвует в экспорте стафилопина - вторичного метаболита бактерий, необходимого для связывания ионов металла. Белок был найден у Staphylococcus aureus (strain Mu50 / ATCC 700699).
DeepTMHMM показал, что в этом белке есть 12 траснмембранных участков. Последовательность CNTE_STAAM была взята с сайте Uniprot и находиться в файле.
1: 10-30
2: 42-66
3: 74-90
4: 96-117
5: 137-154
6: 161-178
7: 214-234
8: 246-264
9: 278-297
10: 302-323
11: 339-360
12: 368-386
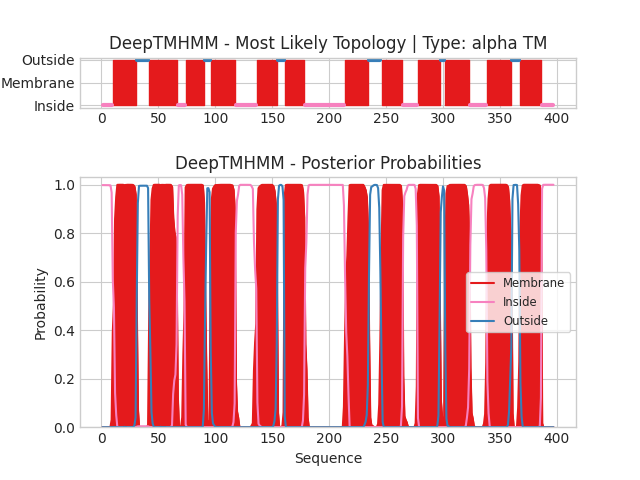

Модель белка была подана на вход программе PPM 3.0 с параметрами: Number of Membranes - 1, Type of membrane - Gramm-positive bacteria inner membrane (так как Staphylococcus aureus - грам-положительная бактерия), Topology (N-ter) - in (если судить по предсказанию DeepTMHMM). В результуте программа нашла 12 траснмембранных сегментов, координаты совпадают с предсказанными DeepTMHMM с различием в 1-2 аминоксилоты, что означает довольно высокую точность.
1: 11-34| 10-30 2: 41-63| 42-66 3: 73-94| 74-90 4: 95-115| 96-117 5: 135-155| 137-154 6: 159-179| 161-178 7: 213-239| 214-234 8: 246-264| 246-264 9: 276-297| 278-297 10: 301-322| 302-323 11: 340-361| 339-360 12: 366-386| 368-386
