Выравнивание геномов
Построение DotPlot
Я решила сравнить бактериальные геномы. Хотела выбрать что-то простое, но не модельный организм, поэтому взяла
Staphylococcus aureus - возбудителя пневмонии. В NCBI выбрала штамм JP080 (AP017922 - complete genome). Штаммов у этой
бактерии было много, но на нескольких выравниваниях генома я поняла, что ничего интересного там нет, поэтому
зашла в NCBI Taxonomy и нашла род Jeotgalicoccus. Затем в Advanced поиске Nucleotides и по запросу
(Jeotgalicoccus[Organism]) AND complete genome
выбрала Jeotgalicoccus sp. ATCC 8456 (NZ_CP066224 - complete genome)
По АС в BLAST выравняла геномы и DotPlot мне показался достаточно интересным. Сначала брала blastn:
При поиске с word size = 11 DotPlot показался мне "пустым" в некоторых частях геномов, поэтому я подумала, что blastn может найти выровненные участки при меньшей длине слова. Но алгоритм не работает про длину слова 7 (ни в какой день ни в какое время...)
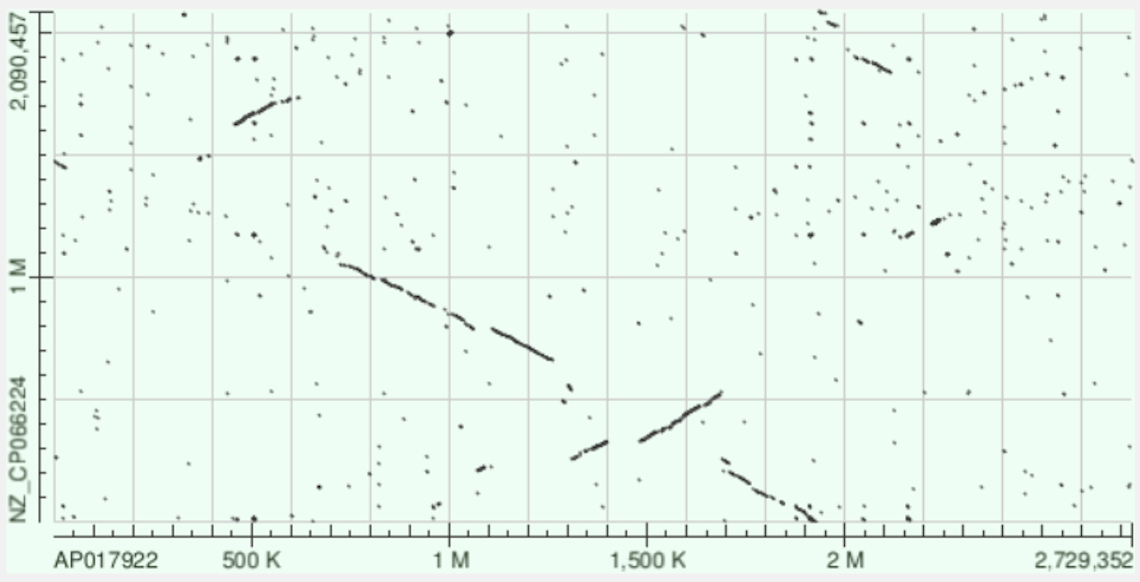
При поиске megablast тоже сделала два запроса с разной длиной слов: 28 (по умолчанию) и 16 (для более четкого DotPlot, то есть хочу увидеть более короткие выровненные последовательности).
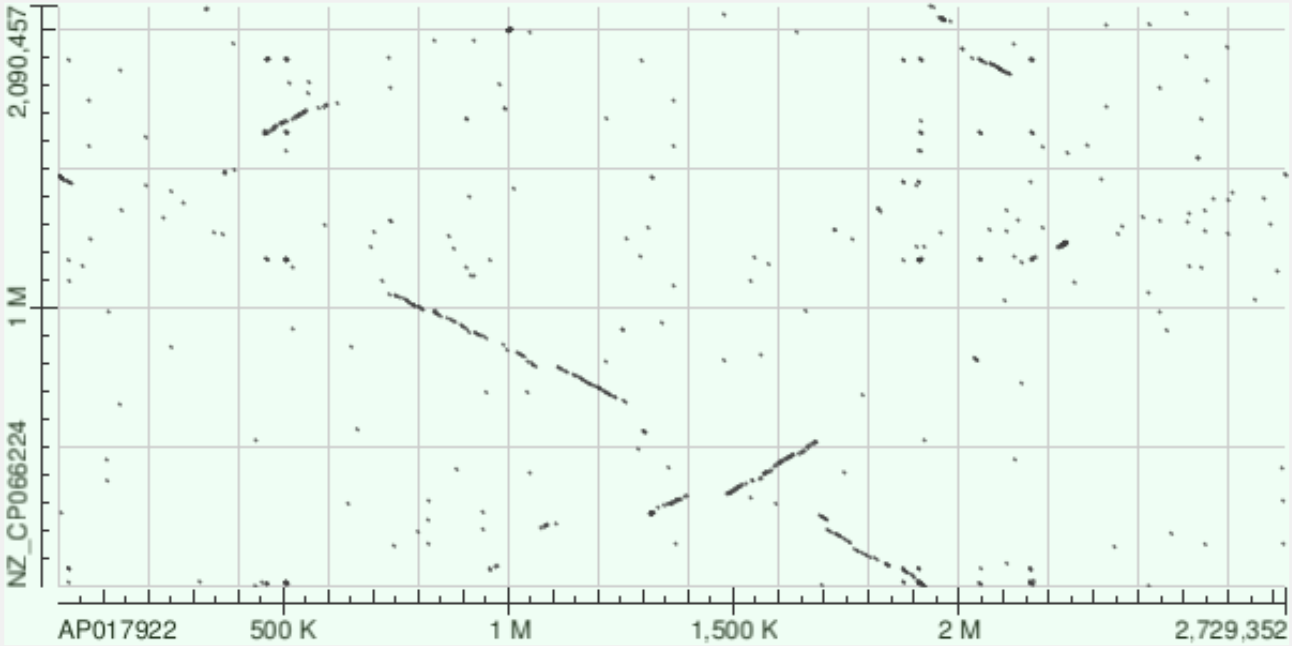
Обсуждение DotPlot
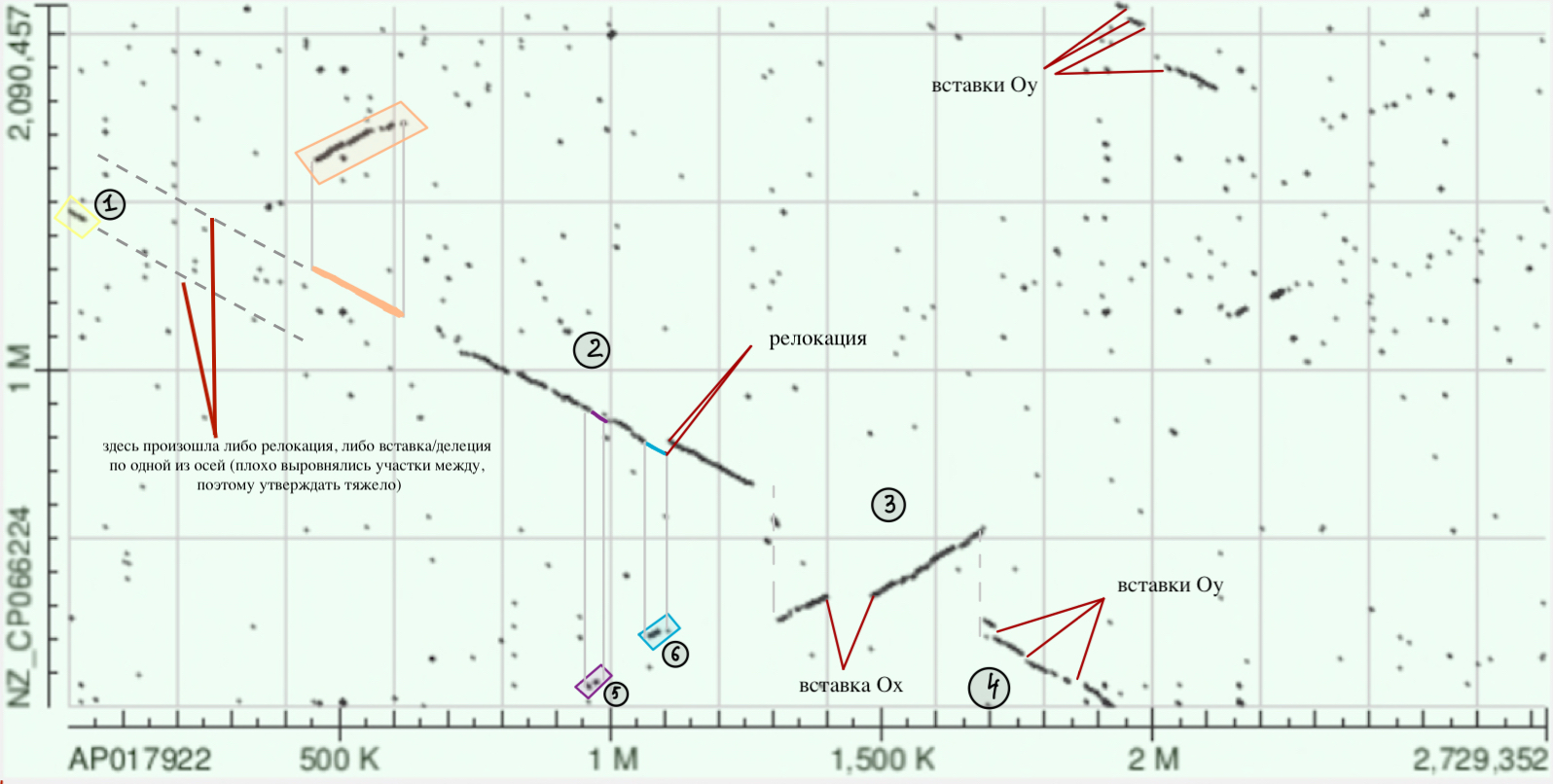
По оси X расположен геном Staphylococcus aureus, по оси Y - Jeotgalicoccus sp..
Основная линия выравнивания идет не по главной диагонали, а по побочной, это значит, что у бактерий выбраны разные цепи
Первый гомологичный участок (Рис. 4 - 1, желтый), длиной около 50К нулкеотидов, расположен на Ox 0 : 50K и Oy 1.5M : 1.45M. Это говорит о том, что точка "разрезания" цепи ДНК выбрана по-разному.
Сразу бросается в глаза достаточно крупный выровненный участок длиной около 500К нуклеотидов (Рис. 4 - 2), затем инвертированный гомологичный участок (Рис. 4 - 3), и продолжение гомологичного участка еще около 350К нуклеотидов (Рис. 4 - 4).
Геном Staphylococcus aureus примерно на 700К нуклеотидов крупнее, поэтому все точки после 2.1М по Ох - случайные совпадения.
Теперь более подробно о глобальных перестройках. Крупная инверсия произошла на участке 1250К - 1700К (Рис. 4 - 3), при этом в самой инверсии видно 2 отдельных крупных гомологичных участка. Причем они находятся не на одной диагонали, а на параллельных прямых, поэтому скорее всего в геноме Staphylococcus aureus произошла вставка участка (длиной около 100К нуклеотидов, координаты 1400К-1500К).
Небольшую вставку можно заметить и у Jeotgalicoccus sp. с координатами 1800К-1900К.
Большая инверсия с реклокацией находится на участке Staphylococcus aureus с координатами 450К-600К (Рис. 4 - оранжевый).
В целом, если принять во внимание все краткие участки, то можно нафантазировать много крупных последовательных перестроек геномов. Небольшие участки с координатами по Staphylococcus aureus 900К-950К (Рис. 4 - 5, фиолетовый) и 1075К-1100К (Рис. 4 - 6, голубой) очень красиво могут встать в крупный гомологичный участок. Скорее всего на этом участке произошло много инверсий с последующими вставками/делециями и релокациями (см. Рис.4).