Нуклеотидный BLAST
Задание 1
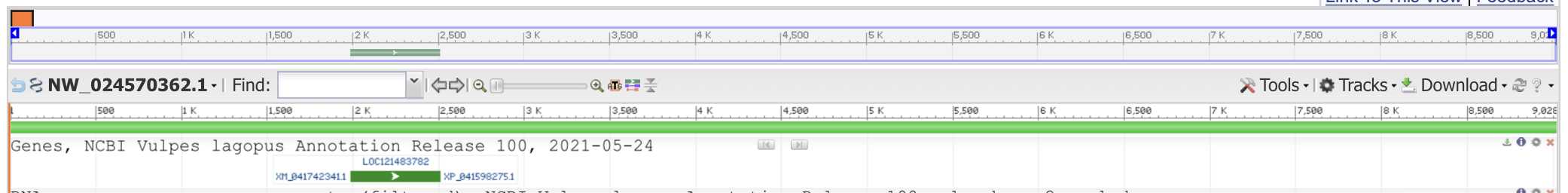
Я выбрала контиг JADWOQ010000542.1 (локус NW_024570362.1, найти graphics получилось только по
идентификатору локуса, хотя указано, что референсная последовательность идентична контигу).
Размер 9028 пн, здесь находится:
CDS 1984..2511
в features указаны еще mRNA и gene с такими же координатами (очевидно)
Ссылочка на fasta контига JADWOQ010000542.1
А теперь BLAST
Я попробовала найти что-нибудь среди Anura (лягушьки и жабьки). Mammalia и Anura относятся к Tetrapoda (но относятся к разным надклассам Amniota и Amphibia соответственно). Но, на удивление, (при word size = 16) blastn ничего не нашел. Тогда я попробовала поискать среди любых организмов, не относящихся к семейству Canidae. Потом я зашла в раздел Taxonomy и посмотрела, к каким таксонам относятся найденные организмы. Оказалось, что из всех всего несколько организмов относились к птицам, еще меньше - черепахи и крокодилы (всего 5), а все остальные - млекопитающие. Поэтому я решила выбрать просто другой таксон млекопитающих: bats (летучие мыши ("the BAT" by NCT)).
Параметры запуска: организмы - 'bats', show - 50 (так как всего организмов было немного #испр. я это проверяла, то есть я знаю сколько всего там находок. Больше всего их было для blastn и я поставила 50, чтобы убрать последовательности с покрытием 0-1%. Если поставить show 5000, то в bkastn выдастся всего 215 последовательностей, из них 164 имеют Query Cover 0-2%. Поэтому я указываю show 50 уже зная, сколько их там, потому что перепроверяла все, но наверное надо было сразу описать первый запуск))#). Остальные настройки по умолчанию.
blastn: word size - 11. 50 значимы находок (среднее покрытие 7%, что соответствует проценту последовательности CDS в контиге). Выравнивания с CDS встречаются реже, чем с некодирующей областью (где-то 1:2). Используется, чтобы сравнить геномы организмов (близкородственных), а также для определения таксона организма.
megablast: word size - 28. Всего 25 последовательностей с покрытием 1% (т.е. участок длиной 150-180 нуклеотидов, который находится в середине CDS). Используется для поиска последовательностей близкородственных организмов (например можно понять их таксономическое отношение близкородственных организмов).
blastx: word size - 5. Всего 25 последовательностей с покрытием 1% (angiogenic factor with G patch and FHA domains 1 и гипотетические белки). Используется для определения кодирующих последовательностей, гомологов белков, мутаций.
tblastx: при word size = 2 и 3 находок не было :( Используется для поиска гомологов белков (с нуклеотидной последовательности транслиурется белковая последовательность и по ней ищутся гомологи, помогает справиться с вырожденностью генетического кода).
Сравним находки разных BLASTов.
У blastn и megablast похожая выдача: организмы одинаковые, но порядок следования разный: blastn - Hipposideros armiger - Rhinolophus sinicus - Molossus molossus; в megablast - Molossus molossus - Rhinolophus sinicus - Hipposideros armiger. При поиске в blastx первыми идут Rhinolophus sinicus, затем Hipposideros armiger, а вот Molossus molossus находится в конце выдачи. В целом выдача всех алгоритмов схожа, но порядок следования разный (из-за отличий в алгоритмах).
Задание 2
Индексация последовательности генома
makeblastdb -in GCF_018345385.1_ASM1834538v1_genomic.fna -dbtype nucl
В файле лежат последовательности 16S rRNA и 23S rRNA - входят в состав малой и большой субъединиц рибосомы прокариот соответственно. Их функции - структурная (положение рибосомальных белков), связывают большую и малую субъединицы рибосомы; 16S rRNA узнает последовательность Шайн-Дальгарно на мРНК; 23S частично входит в PTC (#испр peptidyl transferase center#) большой субъединицы.
Буду использовать blastn (так как гены rRNA не CDS и используются последовательности совсем неродственных организмов).
blastn -task blastn -query 16S_rRNA.fa -db GCF_018345385.1_ASM1834538v1_genomic.fna -out blastn_16S.out -evalue 0.05 -outfmt 7
blastn -task blastn -query 23S_rRNA.fa -db GCF_018345385.1_ASM1834538v1_genomic.fna -out blastn_23S.out -evalue 0.05 -outfmt 7
(ставлю -evalue=0.05, чтобы отбросить совсем плохие находки, а -outfmt=7 для выдачи в виде таблицы)
Результаты:
16S: 15 находок. 2 хромосомы и 13 скэффолдов. Файл
23S: 36 находок. Митохондриальная хромосома, 12 хромосом, 23 скэффолда. Файл
Интересно, что в топе и 16S, и 23S, находится NC_054830.1 - 7 хромосома (в 16S score 1149, в 23S - 1482) и скэффолд NW_024571137.1 (score 16S = 475, 23S = 1141). У большинства находок аннотированы rRNA (у некоторых есть просто ncRNA, но как я поняла, NCBI относит rRNA к ncRNA). Это вполне логично.
#испр. "почему-то упомянуто о митоДНК только в контексте поиска по 23S" - ну, потому что в контексте поиска по 16S митоДНК нет в находках)) NC_026529.1 - 'Vulpes lagopus mitochondrion, complete genome.', все скэффолды - 'Vulpes lagopus strain Blue_001 unplaced genomic scaffold'.