Домены и профили
Выбор домена и подсемейства
Из PFam я выбрала домен PF17106 - Sigma domain on NACHT-NTPases ( сигма-домен NACHT-NTP-азы). Некоторая информация об этом домене:
- ID: NACHT_sigma
- AC: PF17106
- seed: 33
- Uniprot: 475
- full: 180
- Domain architectures: 83
Я выбрала подсемейство белков, содержащих два домена: SesA и NACHT_sigma (B6HH10). SesA - PF17107, N-terminal domain on NACHT_NTPase and P-loop NTPases. Функция подсемейства неизвестна. Всего белков с такой архитектурой 156. Сначала в белках идет домен SesA, затем NACHT_sigma.
Далее я выровняла последовательнсти подсемейства в Jalview. С помощью Remove redundancy я удалила последовательности с порогом 90%. Осталось 113 последовательности. Выравнивала я с помощью Mafft. Оказалось, что есть 6 последовательности, которые плохо выровнялись с остальными (у них есть последовательность между очень консервативными участками), поэтому я их удалила. Итого осталось выравнивание из 107 белков, которое можно скачать ниже:
- Выравнивание Mafft 107 белков подсемейства
- Длина выравнивания 276
Создание HMM профиля
Далее я построила HMM-профиль с помощью следующих команд:
hmm2build hmmout pr11_for_hmm.fa
hmm2calibrate hmmout
hmm2search --cpu=1 hmmout pr11_full.fasta > hmm_results.txt
- HMM-профиль здесь
- Результаты поиска здесь
- Длина профиля 230 (в Pfam заявлено, что длина данной доменной архитектуры 208)
Анализ HMM-профиля
Для обработки результатов сначала пришлось обработать в Excel таблицу с результатами hnn_results.txt, так как в ней плохо настроена табуляция. Таблица, которая нужна для обработки, находится в файле hmm_out.txt . Ссылки на все файлы ниже:
- Список AC из выравнивания full
- Список AC доменной архитектуры
- Список AC белков, использлованных для создания HMM-профиля
- Таблица hmm_out.txt (все то же самое, что есть в hmm_results.txt, только кратко и с номральной табуляцией)
Вся обработка была выполнена в блокноте в коллабе (ссылка тут). Гистограммы весов и длин белков:
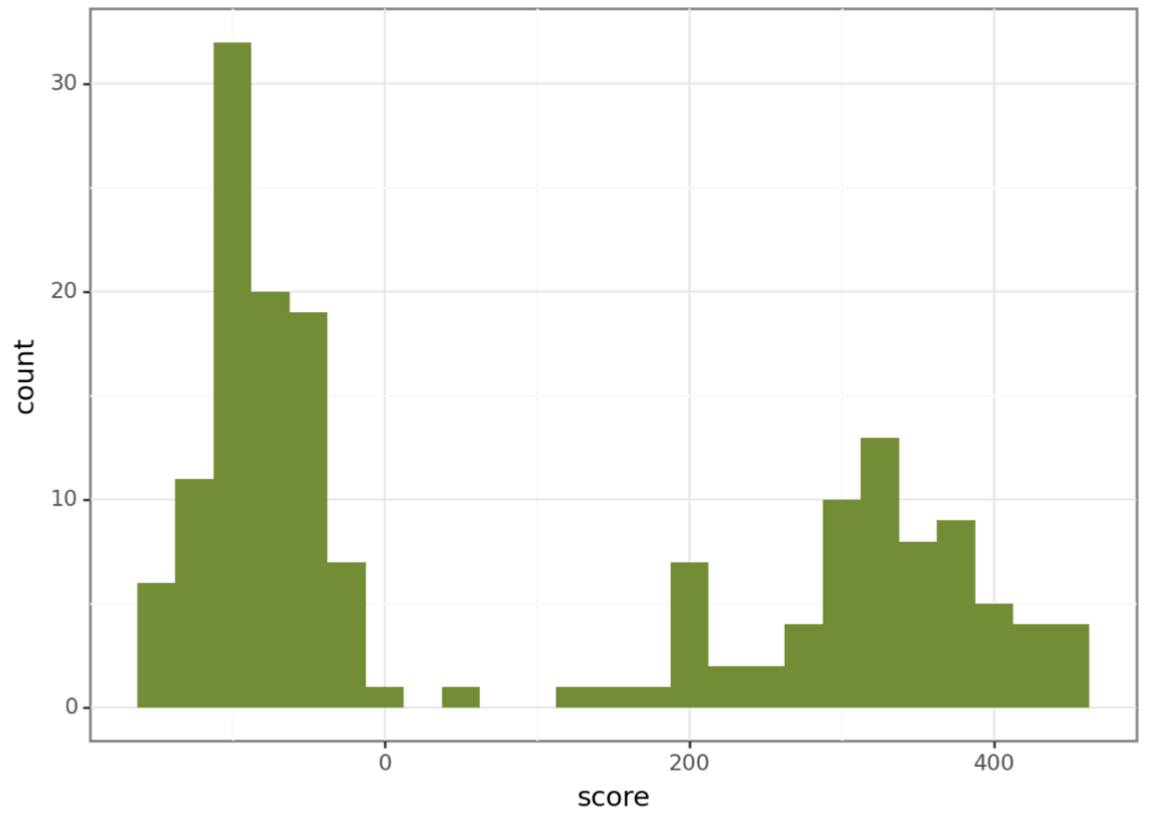
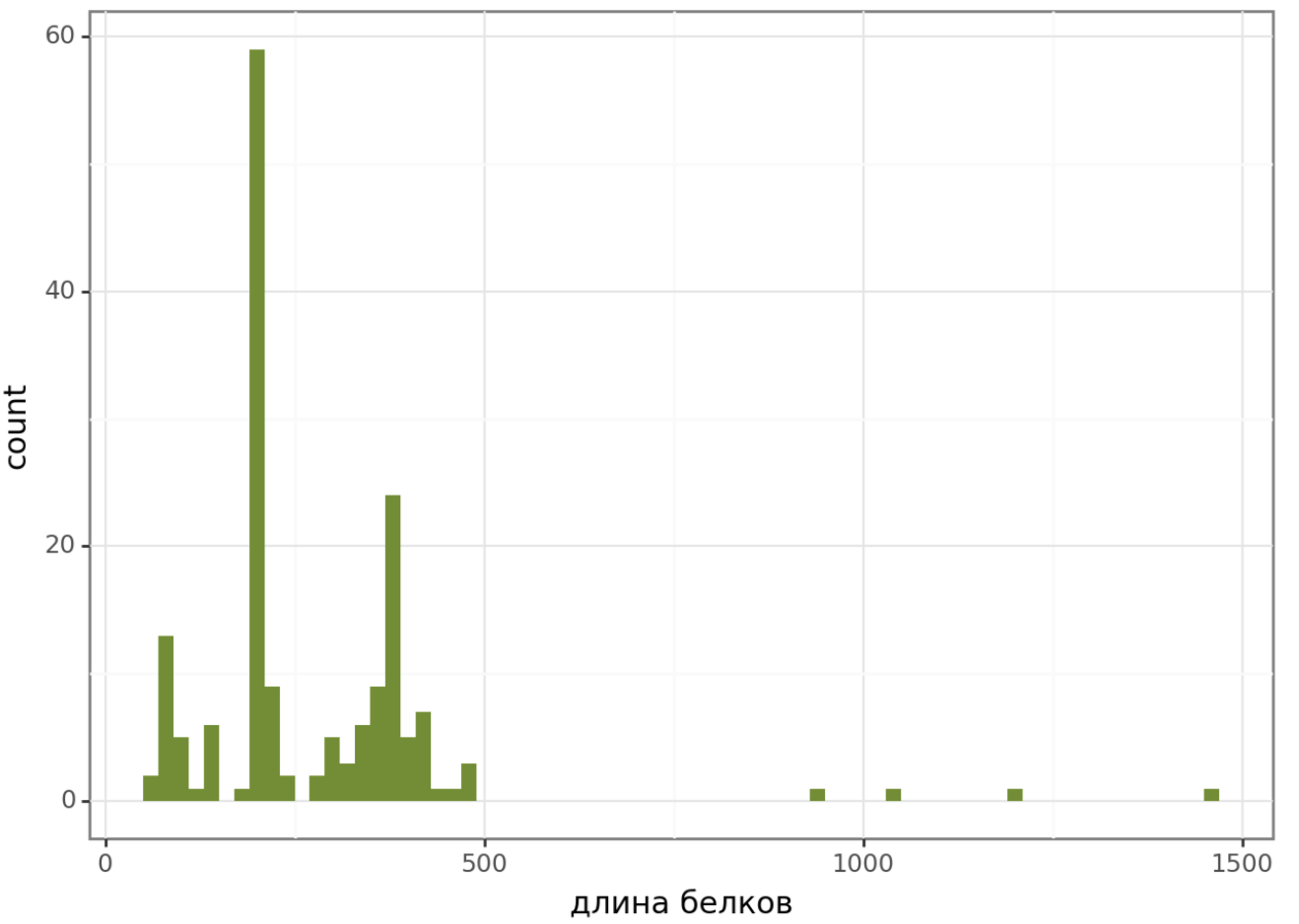
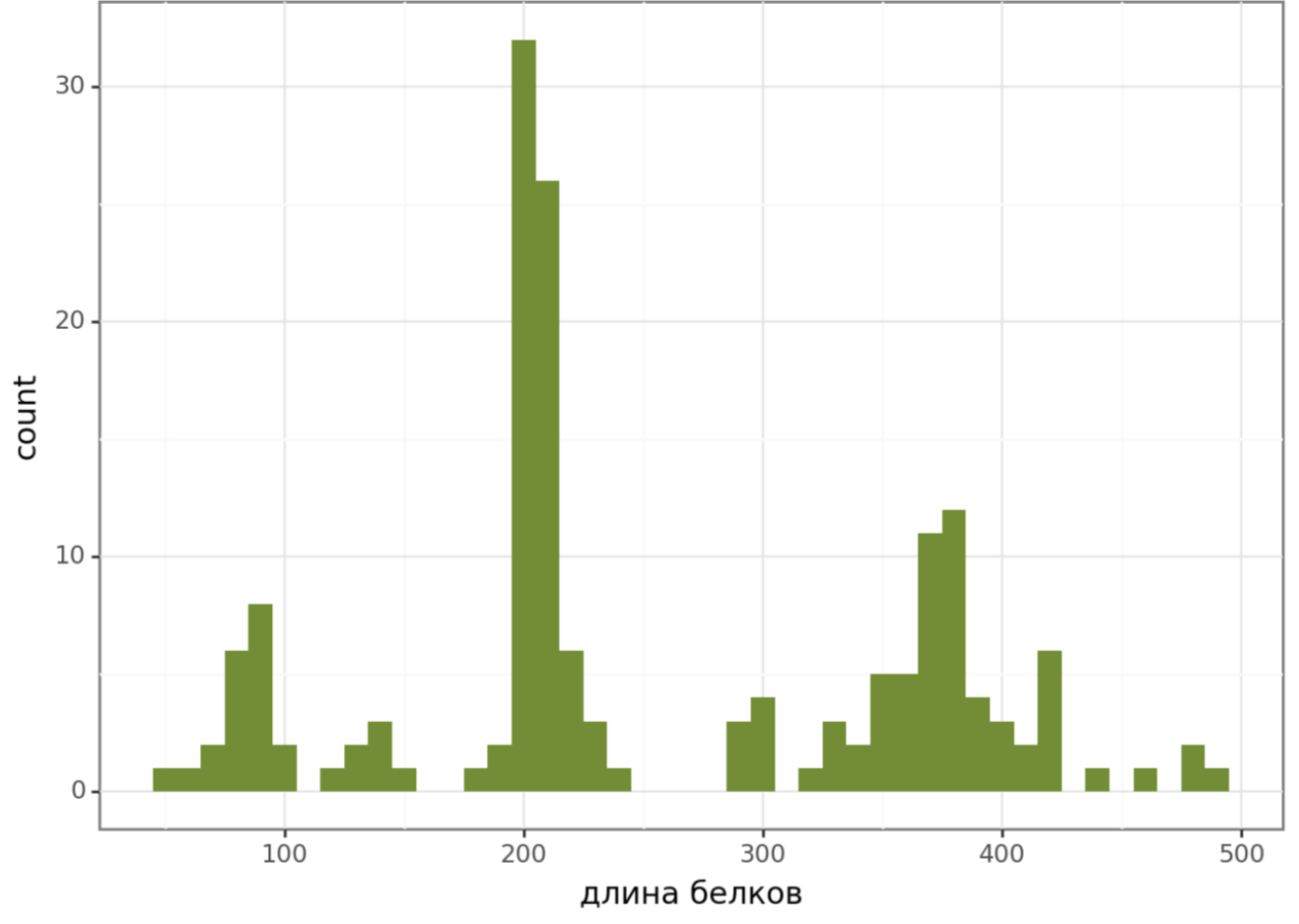
К таблице с помощью скрипта были добавлены 2 колонки: 'Has domain architecture' и 'Was used for HMM-profile', значения в столбцах 0 (нет) и 1 (да).
Домен нашелся в 168 последовательностях из 180 (из выборки full). Я добавила в таблицу последовательности, не вошедшие в hmm_results.txt, значения в строках для этих белков все проставлены как NaN, так как здесь 0 и отсутствие значения - разные вещи. Для построения ROC-curve и F1 я использовала все тот же скрипт.
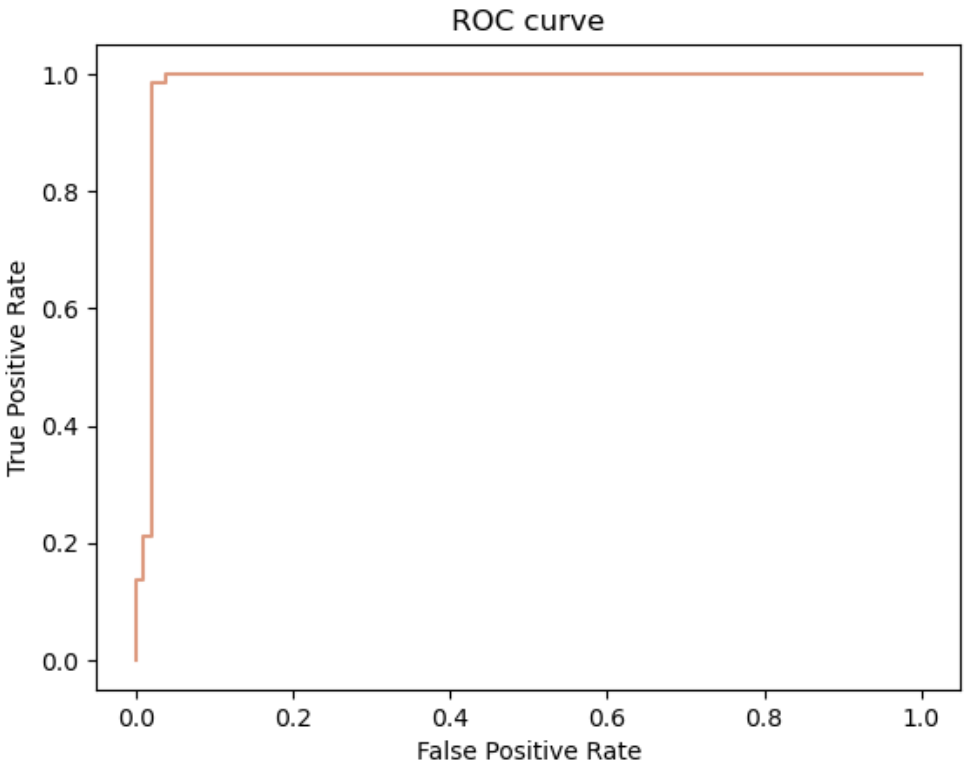
Оптимальное соотношение специфичности и чувствительности по графику ROC: чувствительность 98.5%, специфичность = 97.1% . Также по графику можно сказать, что ROC-AUC score большой, то есть модель хорошо распознает домен NACHT_sigma в белках.
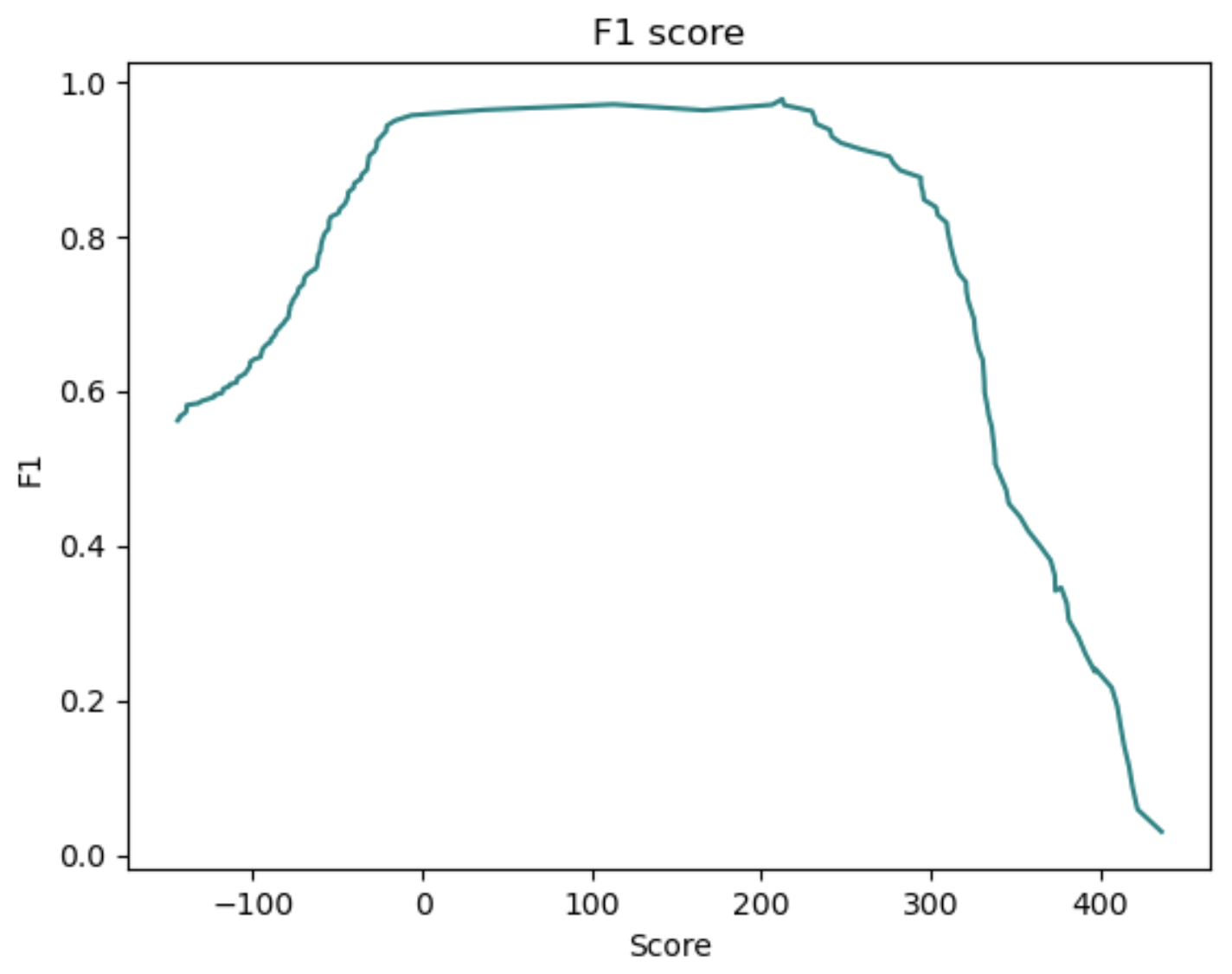
Что касается графика зависимости F1 от веса, то можно сказать, что пик наблюдается при весе 200 (как я и выбрала на гистограмме весов). Затем параметр F1 начинает падать.
то есть порог веса установим равным 200!