Ортологи и паралоги
Выбранные организмы
| Название бактерии | Мнемоника |
|---|---|
| Aromatoleum aromaticum | AROAE |
| Burkholderia mallei | BURMA |
| Pasteurella multocida | PASMU |
| Polynucleobacter asymbioticus | POLAQ |
| Pseudomonas aeruginosa | PSEAE |
| Roseobacter denitrificans | ROSDO |
| Shewanella denitrificans | SHEDO |
Поиск гомологов blastp
Алгоритм поиска гомлогов белка CLPX_ECOLI в протеомах выбранных организмов:
1. Перенесла последовательность CLPX_ECOLI в ecoli_clpx.fasta2. Создание fasta-файла с протеомами всех выбранных бактерий
cat AROAE.fasta BURMA.fasta PASMU.fasta POLAQ.fasta PSEAE.fasta ROSDO.fasta SHEDO.fasta > ~/proteomes.fasta3. Создание БД для поиска blastp из протеомов выбранных бактерий
makeblastdb -dbtype prot -in proteomes.fasta -out clpx_db4. Поиск гомологов по запросу ecoli_clpx.fasta в созданной ДБ с порогом e-value 0.001, результаты в виде таблицы в results.txt
blastp -query ecoli_clpx.fasta -db clpx_db -evalue 0.001 -out results.txt
Выдача ( полный файл тут ):
sp|Q12LA2|CLPX_SHEDO ATP-dependent Clp protease ATP-binding subun... 712 0.0sp|Q9I2U0|CLPX_PSEAE ATP-dependent Clp protease ATP-binding subun... 654 0.0
sp|Q5P160|CLPX_AROAE ATP-dependent Clp protease ATP-binding subun... 619 0.0
sp|Q62JK8|CLPX_BURMA ATP-dependent Clp protease ATP-binding subun... 617 0.0
sp|A4SXD7|CLPX_POLAQ ATP-dependent Clp protease ATP-binding subun... 613 0.0
sp|P57981|CLPX_PASMU ATP-dependent Clp protease ATP-binding subun... 612 0.0
sp|Q165G0|CLPX_ROSDO ATP-dependent Clp protease ATP-binding subun... 582 0.0
sp|Q9HUC5|HSLU_PSEAE ATP-dependent protease ATPase subunit HslU O... 95.9 1e-21
sp|Q12IT8|HSLU_SHEDO ATP-dependent protease ATPase subunit HslU O... 94.0 5e-21
sp|Q16CY2|HSLU_ROSDO ATP-dependent protease ATPase subunit HslU O... 93.6 8e-21
sp|Q5P503|HSLU_AROAE ATP-dependent protease ATPase subunit HslU O... 93.6 8e-21
sp|P57968|HSLU_PASMU ATP-dependent protease ATPase subunit HslU O... 93.2 1e-20
sp|Q62F00|HSLU_BURMA ATP-dependent protease ATPase subunit HslU O... 82.4 4e-17
sp|Q168A2|RUVB_ROSDO Holliday junction branch migration complex s... 45.4 3e-05
tr|A0A0H2WJ72|A0A0H2WJ72_BURMA ATP-dependent zinc metalloprotease... 43.9 9e-05
tr|A4SXL5|A4SXL5_POLAQ ATP-dependent zinc metalloprotease FtsH OS... 43.5 2e-04
tr|Q9HV48|Q9HV48_PSEAE ATP-dependent zinc metalloprotease FtsH OS... 43.5 2e-04
tr|Q9CNJ2|Q9CNJ2_PASMU ATP-dependent zinc metalloprotease FtsH OS... 43.1 2e-04
tr|Q9CKU5|Q9CKU5_PASMU ComM OS=Pasteurella multocida (strain Pm70... 41.6 6e-04
tr|Q16C81|Q16C81_ROSDO Chaperone protein ClpB OS=Roseobacter deni... 41.2 7e-04
tr|Q167Z2|Q167Z2_ROSDO ATP-dependent zinc metalloprotease FtsH OS... 41.2 7e-04
tr|Q12QI8|Q12QI8_SHEDO ATP-dependent zinc metalloprotease FtsH OS... 41.2 8e-04
Реконструкция и визуализация
Из всех записей оставила только мнемоники и загрузила в JalView, полученные последовательности закинула в NGPhylogeny.fr
Параметры:
Multiple Alignment: MAFFT
Tree Inference: FastME
Gamma distributed rates across sites: No
Starting tree: BIONJ
Number of bootstrap replicates: 100

В принципе, тут уже видно ортоголоические группы и примеры ортологов/паралогов, но лучше укоренить.
Укореняю в middlepoint и включаю отображение бутстрэп анализа (в двух вариантх, дальше будет только кружочками)
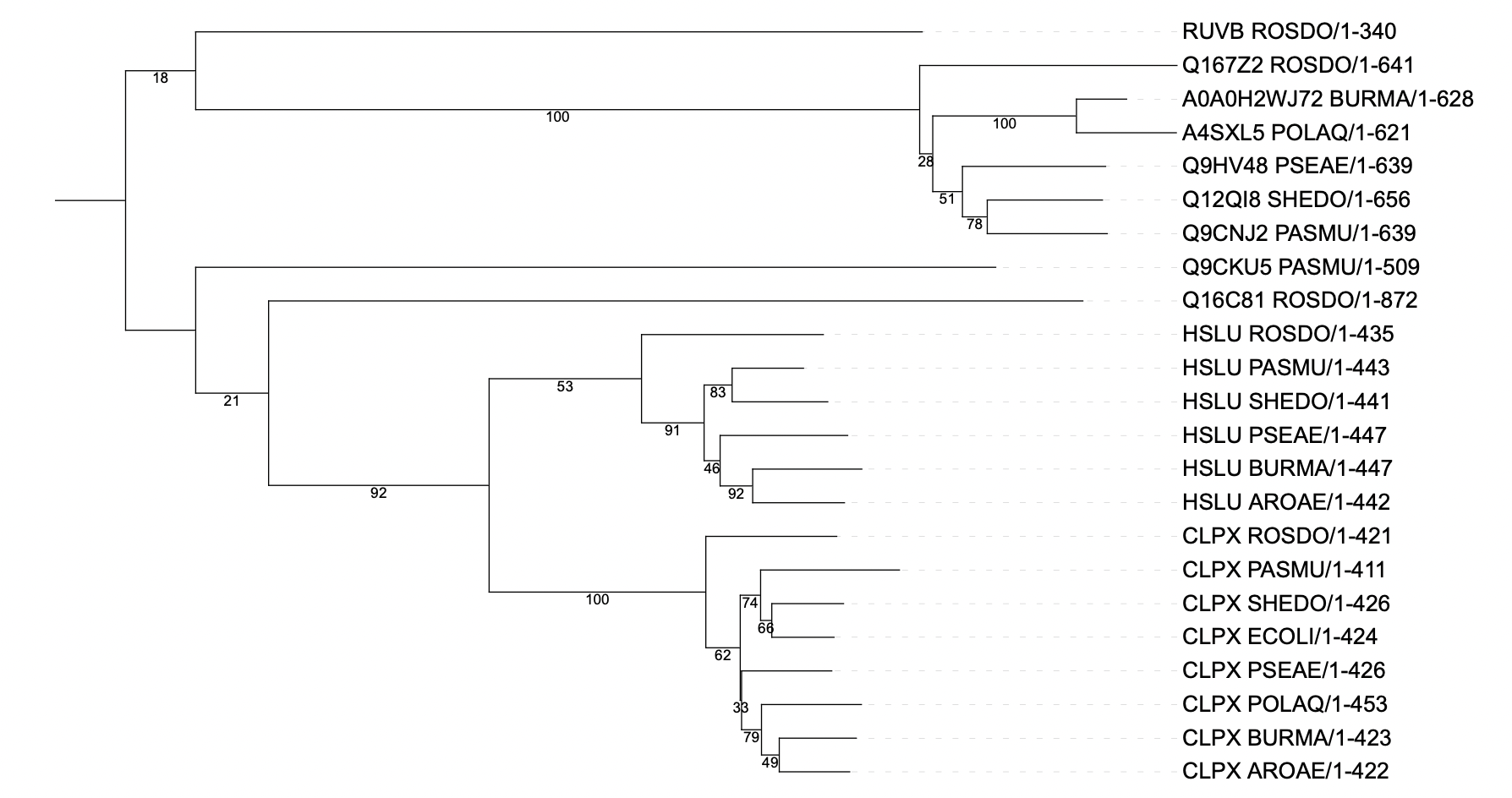
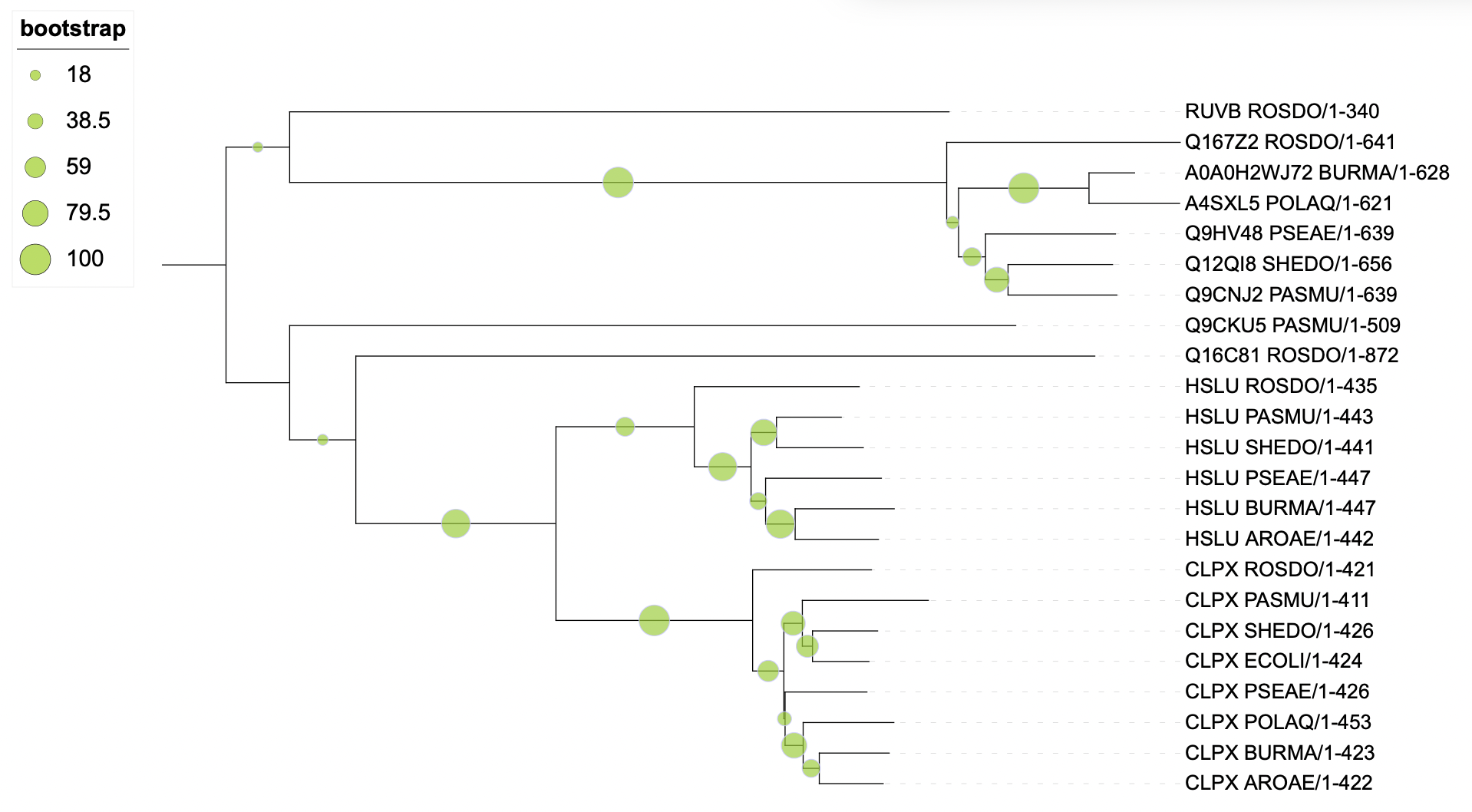
Примеры:
Ортологи: CLPX_BURMA и CLPX_AROAE; HSLU_PASMU и HSLU_SHEDO; Q9CNJ2_PASMU и Q12QI8_SHEDO
Паралоги: HSLU_PASMU, Q9CNJ2_PASMU, CLPX_PASMU, Q9CKU5_PASMU
Выделение ортологических групп
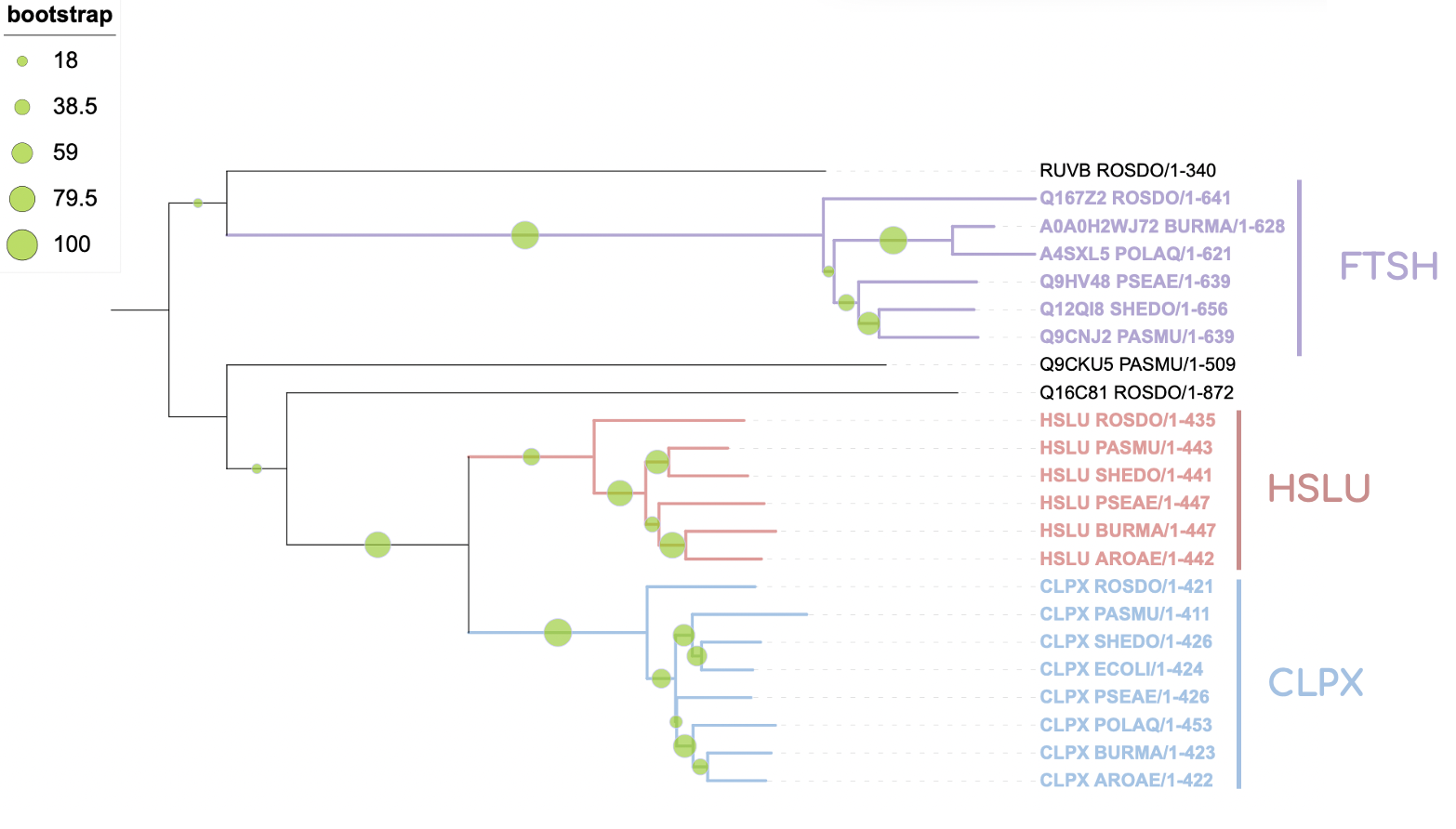
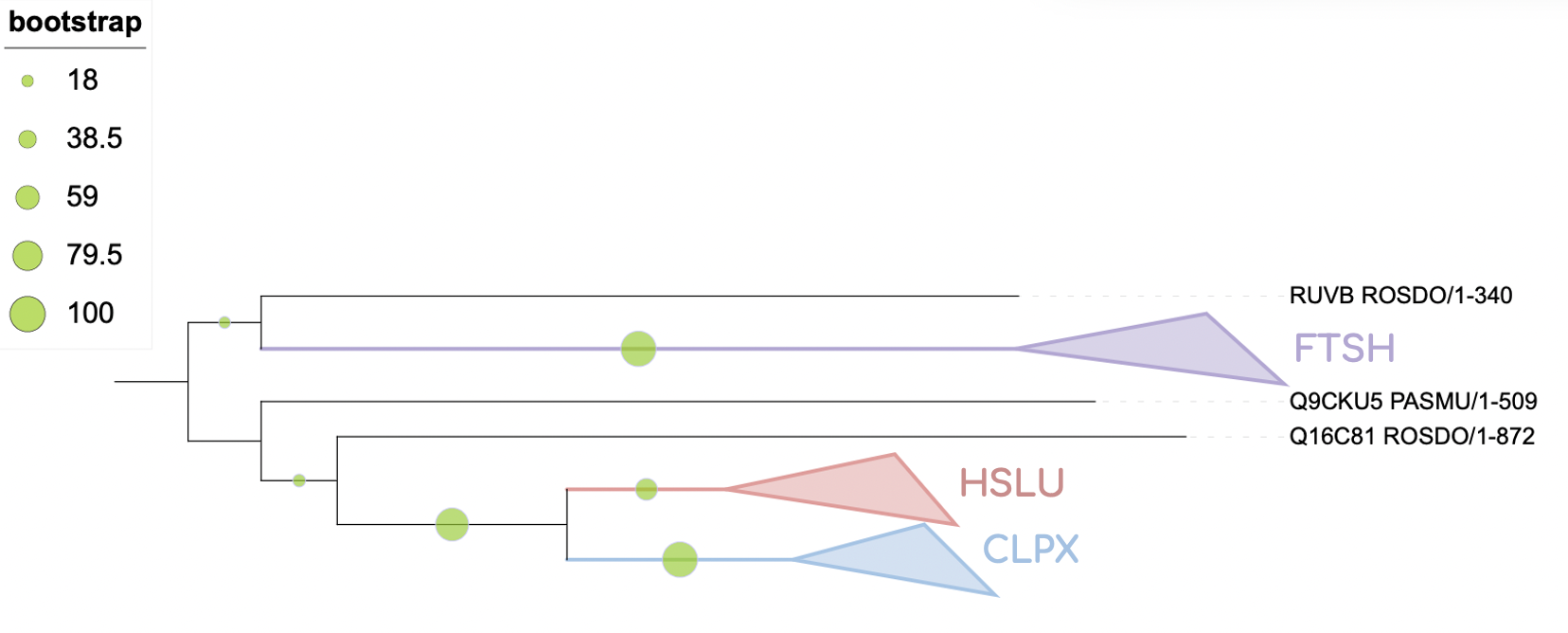
В этом дереве можно выделить 3 ортологические группы:
CLPX - АТФ-связывающий сабъюнит АТФ-зависимой протеазы Clp (ATP-dependent Clp protease ATP-binding subunit СlpX). В эту группу входят все 7 (8 включая ECOLI) белков выбранных бактерий. Топология совпадает с исходынм деревом.
HSLU - АТФ-зависимый сабъюнит АТФ-зависимой протеазы HslU (ATP-dependent protease ATPase subunit HslU). В эту группу входят 6 белков выбранных бактерий, то есть все, кроме POLAQ. Топология соответствует исходной.
FTSH - АТФ-зависимая цинковая металлопротеаза FtSH (ATP-dependent zinc metalloprotease FtsH). В эту группу входят 6 белков выбранных бактерий, то есть все, кроме AROAE. Топология совпадает с исходным деревом.