Трансмембранные белки
Сравнение предсказаний трансмембранных участков в бета-листовом белке
Описание белка
В базе данных OPM выбрала белок со следующими характеристиками:
type: transmembrane (трансмембранные белки)
class: β-barrel transmembrane (белки с β-листами в трансмембранной части)
superfamily: Ligand-gated protein channels (лиганд-зависимые ионные каналы)
family: outer membrane receptor (OMR) (рецепторы внешней мембраны)
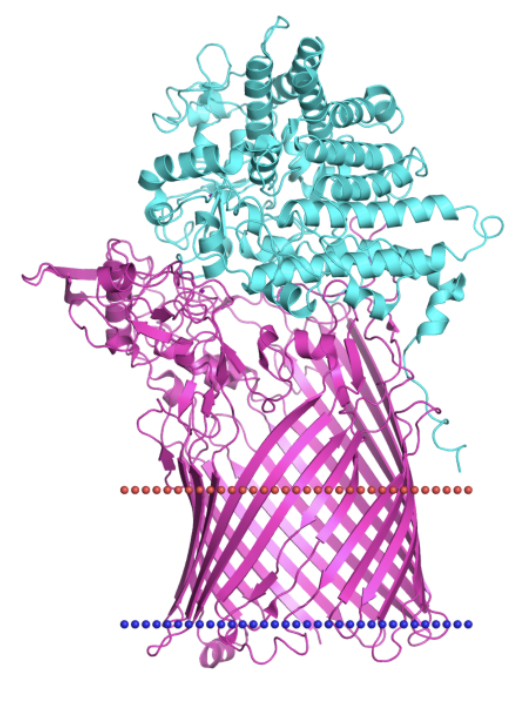
name: SusD-SusC complex (комплекс SusD-SusC, см. ниже)
pdb: 5t3r
uniprot: Q8A6W3_BACTN, Q8A6W4_BACTN
function: Комплекс состоит из двух белков: SusD и SusC. Структура комплекса есть в pdb, но в
Uniprot они распологаются разными записями. SusD - крахмал-связывающий белок (Starch-binding protein SusD) -
связывает крахмал, а его транспорт в периплазму для деградации (расщепления полимерных олигосахаридов крахмала)
проводит белок SusC (TonB-dependent receptor SusC).
Из записи PDB комплекса (5t3r) скачала fasta-последовательность и загрузила ее в DeepTMHMM. Но при всей fasta комплекса DeepTMHMM работал как-то не очень (не выдавал картинки), поэтому я загрузила только fasta SusC (потому что рецептор SusD связан с трансмембранным белком, но сам им не является, что и смущало работу алгоритма). Таблицу с данными β-листов пришлось брать из работы из полного fasta (чтобы сохранилась нумерация АК), а картинку расположения вторичных структур - из работы только по fatsa SusC.
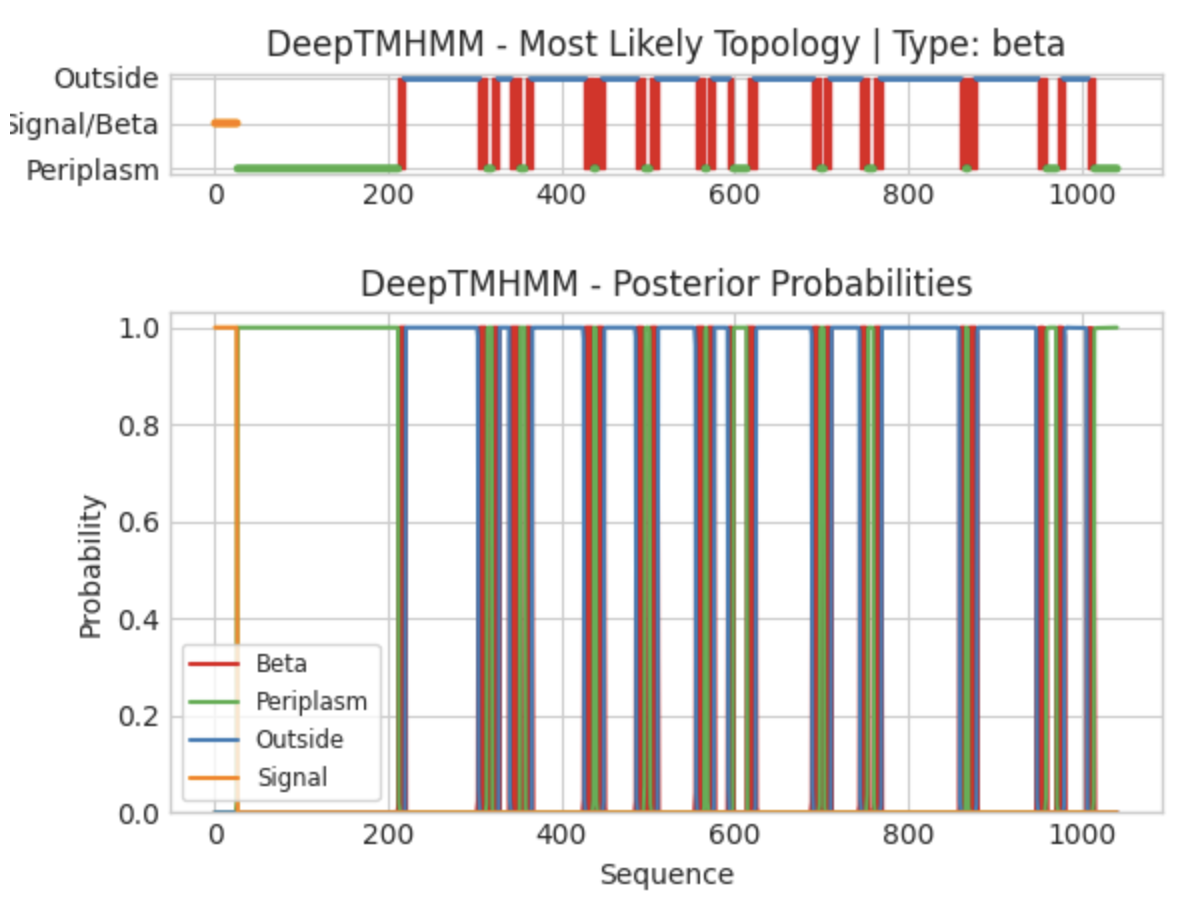
После загрузки fasta только SusC DeepTMHMM заработал нормально и нашел 22 трансмембранных β-листа. Текстовая выдача в формате .gff3
Описание трасмембранных участков
номер петли: OPM | DeepTMHMM
1: 213- 221 | 213- 220
2: 305- 314 | 305- 314
3: 321- 328 | 321- 328
4: 341- 352 | 341- 352
5: 359- 366 | 359- 366
6: 427- 437 | 427- 437
7: 441- 449 | 441- 449
8: 487- 496 | 487- 496
9: 503- 511 | 503- 511
10: 556- 565 | 556- 565
11: 569- 576 | 569- 576
12: 593- 598 | 593- 598
13: 615- 624 | 615- 624
14: 690- 699 | 690- 698
15: 704- 711 | 704- 711
16: 745- 754 | 745- 754
17: 761- 769 | 761- 769
18: 860- 867 | 860- 866
19: 870- 879 | 870- 879
20: 949- 959 | 949- 959
21: 972- 980 | 972- 980
22: 1007-1014 | 1007-1014
По "таблице" с координатами β-листов видно, что их количество по обоим алгоритмам одинаковое и равно 22. Единственные отличия я выделила жирным шрифтом в таблице: в 3 из 22 листов DeepTMHMM определяет длину листа на 1 меньше. Я подумала, что, может быть, все "выделяющиеся" остатки обращены в конкретную сторону от мембраны, но в случае 1 листа 221-я АК находится снаружи клетки, а в 14 и 18 листах 699-я и 867-я АК - в периплазме. Поэтому, я думаю, что это просто погрешность предсказания структур.
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Описание белка
В базе данных OPM выбрала белок со следующими характеристиками:
swiss-prot AC: Q9I540 (KUP_PSEAE)
name: Probable potassium transport system protein Kup (вероятный белок системы транспорта калия Kup)
organism: Pseudomonas aeruginosa (Грам-)
function: Транспорт ионов К+ в клетку; возможно осуществляет симпорт
К+ и Н+ в клетку.
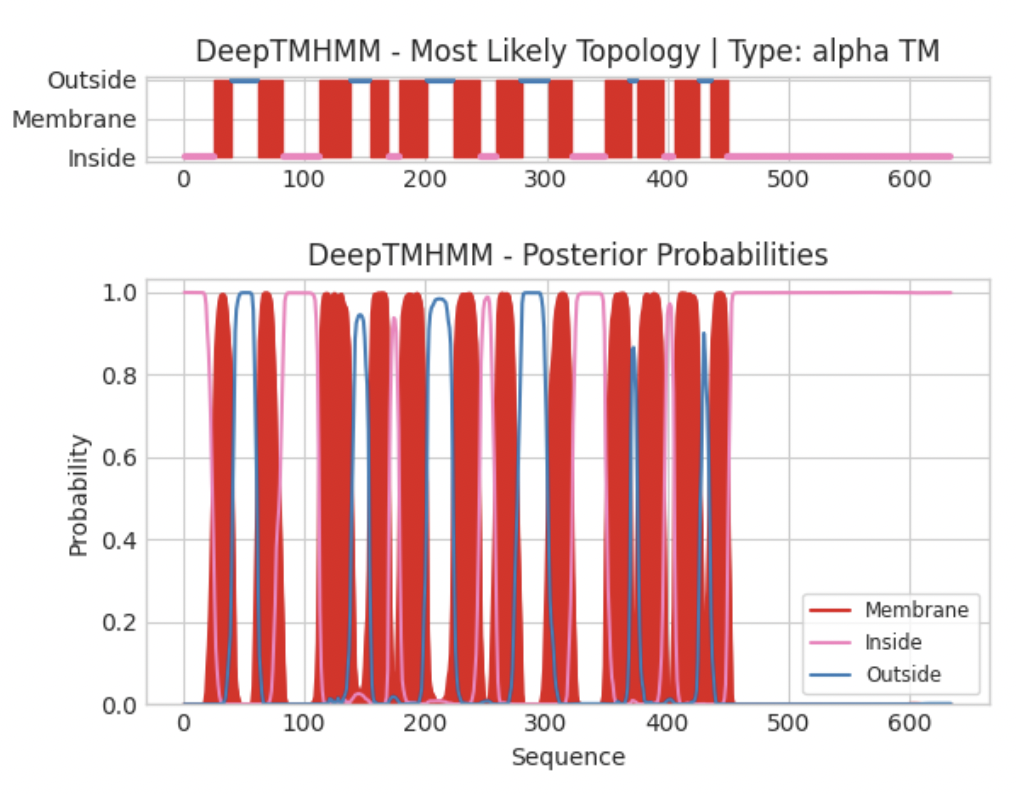
DeepTMHMM нашел 12 трансмембранных α-спиралей. Текстовая выдача в формате .gff3
Чтобы получить описание трансмембранных участков из AlphaFold, скачала файл с описанной структурой и загрузила его
в PPM 3.0. Параметры запуска алгоритма:
type: transmembrane (трансмембранные белки)
number of membranes: 1
type of membrane: Gram-negative bacteria inner membrane (по аннотации Uniprot)
allow curvation: no (пусть будет дефолтное значение, так как сильно глубокой задачи
расположения белка у нас нет, а структуры должны определиться и так)
topology N-ter: in (чтобы N-конец был направлен в цитоплазму. По предсказанию DeepTMHMM (inside 1-25))
Описание трасмембранных участков
номер петли: DeepTMHMM | PPM 3.0
1: 26-40 | 23-45
2: 62-82 | 60-83
3: 113-138 | 116-147
4: 154-169 | 152-170
5: 179-201 | 177-201
6: 223-245 | 224-246
7: 259-279 | 254-275
8: 302-321 | 295-321
9: 349-369 | 352-374
10: 375-396 | 375-395
11: 406-436 | 406-433
12: 436-449 | 434-450
Количество найденных α-спиралей совпадает у алгоритмов (12), однако границы более размыты, чем в задании с β-листами. Все участки перекрываются (с разной точностью), но не так точно. Например, PPM совем не предполагает петлю между 9 и 10 спиралями. AlphaFold предсказывает все спирали внутри с уверенностью 'very high', но по краям спиралей есть участки 'high' и даже 'low', например, 174-176 (один из краев 5й петли). Но в целом, предсказание "довольно уверенное", поэтому сослаться на него для объяснения отличий было бы не совсем правильно.