Сигналы и мотивы (v1)
Описание сигнала: 5'ss (и немного 3'ss)
Краткое описание
Название сигнала: 5' splice signal (сигнал сплайсинга на 5' конце интрона мРНК)
В чем состоит сигнал: данный сигнал необходим для сплайсинга мРНК
Кому адресован: сплайсосома: U1 мяРНК сплайсосомы (если мы говорим об альтернативных интронах, то U11 мяРНК минорной сплайсосмы)
Предназначение: U1 мяРНК узнает последовательность на 5' конце интрона, связывается с ней и изгибает мРНК для сплайсинга
Сила сигнала: сигнал должен быть умеренно сильным, так как каждая мРНК эукариот подвергается сплайсингу, но в то же время иногда интрон должен вставляться в мРНК - при альтернативном сплайсинге
Описание в более свободном виде
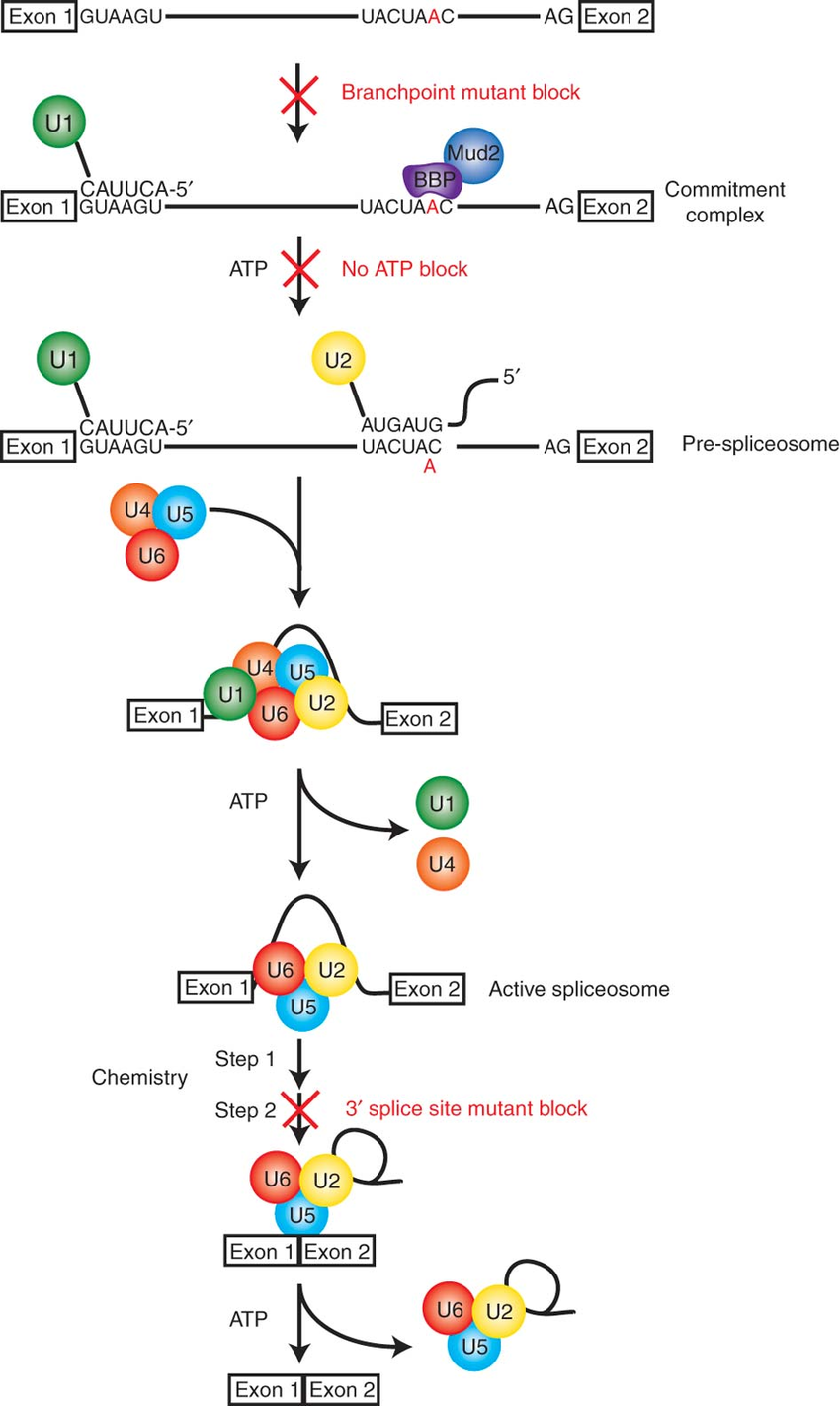
Я выбрала сигнал 5'ss - 5' splicing signal - это последовательность на 5' конце интрона. Все пре-мРНК эукариот подвергаются сплайсингу с образованием мРНК. Сплайсинг осуществляет сплайсосома - это комплекс мяРНК (см. Рис1). Вообще, для сплайсинга можно выделить три основные сигнала на пре-мРНК:
- 5'ss - 5' конец интрона
- branchpoint - точка ветвления, в котором находится аденин (необходим для изгибания интрона в форму лассо)
- 3'ss - 3' конец интрона
На самом деле, существует несколько типов интронов, которые отличаются ак раз консервативными последовательностями на 5' и 3' концах. В зависимости от типа интрона в процессе сплайсинга могут участвовать два типа сплайсосом: классическая (U2-зависимая) и минорная (U12-зависимая) сплайсосмы. Я буду говорить в основном про интроны классического типа: 5'-GU-......-А-...-AG-3'
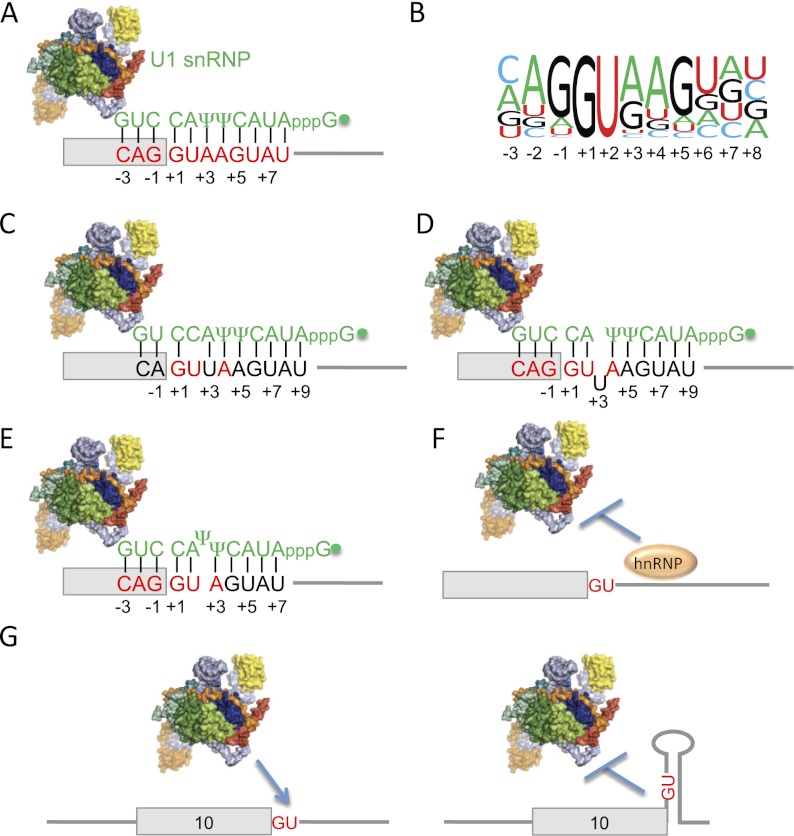
Сплайсосома узнает 5'ss с помощью комплементарного связывания U1 мяРНК с последовательностью на 5'ss. Сила сигнала влияет на силу комплементарных связей в спирали dsRNA, образованной пре-мРНК и U1 мяРНК. Длина этой петли 11 пар оснований. Самыми консервативынми позициями являются первые два нуклеотида интрона (GU) (ну и два последних нуклеотида - там находится 3'ss). Консенсусные последовательности представлены на Рис.2.
Изначально 5'ss комплементарно связывается с 5'-последовательностью U1. Когда U2 связывается с бранчпоинтом и активирует 2'OH аденозина в нем (за счет псведоуридина в составе U2), к нему присоединяется комплекс U4/U6*U5. После этого необходимо сблизить в пространстве 5'ss и бранчпоинт: U1 уходит из комплекса, а 5'ss после перегруппировки комплементарно связывается с U6 - интрон изгибается и образует форму "лассо" - он вырезается, а экзоны остаются в составе мРНК.
Список литературы
- Xavier Roca, Adrian R. Krainer and Ian C. Eperon "Pick one, but be quick: 5′ splice sites and the problems of too many choices", doi: 10.1101/gad.209759.112
- JOHN ROGERS AND RANDOLP HWALL, "A mechanism for RNA splicing", https://doi.org/10.1073/pnas.77.4.1877
- Xavier Roca, Martin Akerman, Hans Gaus, Andrés Berdeja, C Frank Bennett, Adrian R Krainer "Widespread recognition of 5' splice sites by noncanonical base-pairing to U1 snRNA involving bulged nucleotides", DOI: 10.1101/gad.190173.112
Построение PWM последовательности Козак человека
Для выполнения задания я использовала скрипт, данный свыше (любезно предоставленный нашими старшими товарищами).
Скрипт составляет PWM по последовательности вида:
7 нуклеотидов - ATG - 3 нуклеотида
При этом составляются три выборки: 100 последовательностей из генов с известной координатой стартового ATG для построения PWM, 200 последовательностей тестовой выборки (аналогичны) и 200 последовательностей аналогичного вида (7 нукл - ATG - 3 нукл), где ATG является случайным.
Полученная PWM представлена в таблице 1:
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| А | -0.249 | -0.494 | -0.551 | -0.126 | 0.142 | -0.017 | -0.676 | 1.218 | -5.691 | -5.691 | -0.494 | 0.050 | -0.551 |
| С | 0.312 | 0.024 | 0.347 | 0.535 | -0.187 | 0.668 | 0.741 | -5.327 | -5.327 | -5.327 | -0.248 | 0.506 | 0.198 |
| G | 0.312 | 0.808 | 0.347 | 0.071 | 0.668 | -0.312 | 0.535 | -5.327 | -5.327 | 1.582 | 0.987 | -0.381 | 0.506 |
| T | -0.340 | -0.676 | -0.166 | -0.551 | -1.187 | -0.612 | -1.438 | -5.691 | 1.218 | -5.691 | -0.986 | -0.340 | -0.206 |
Посмотрим на гистограммы весов:
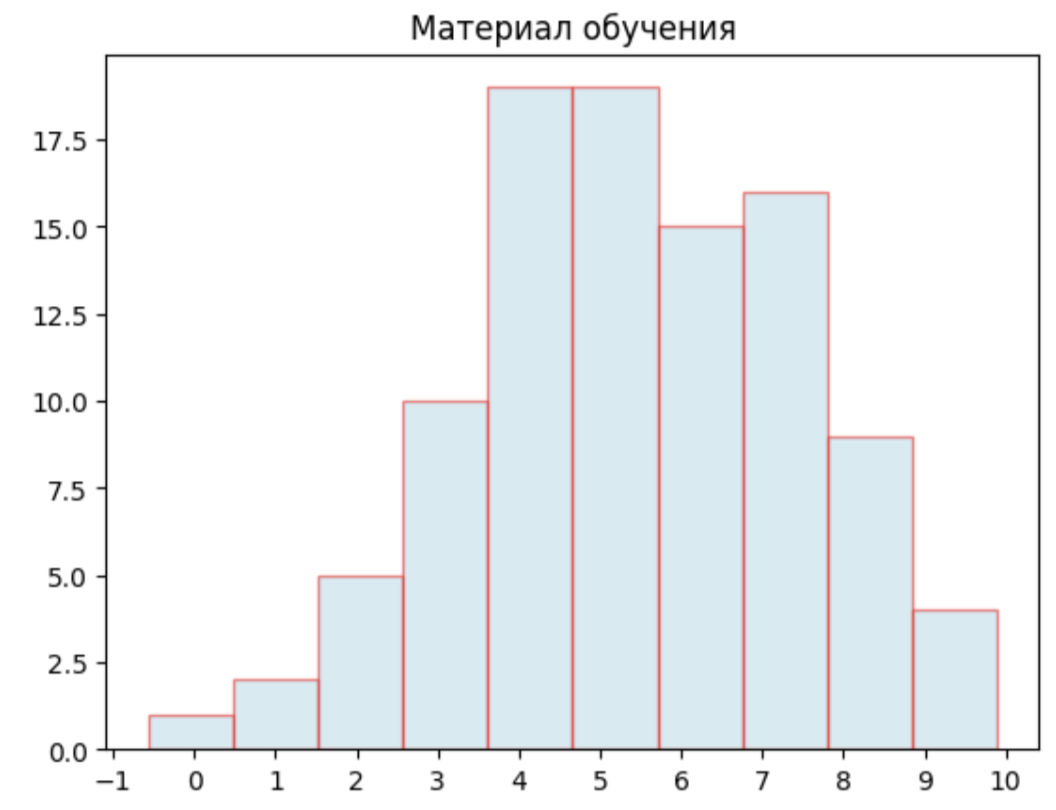
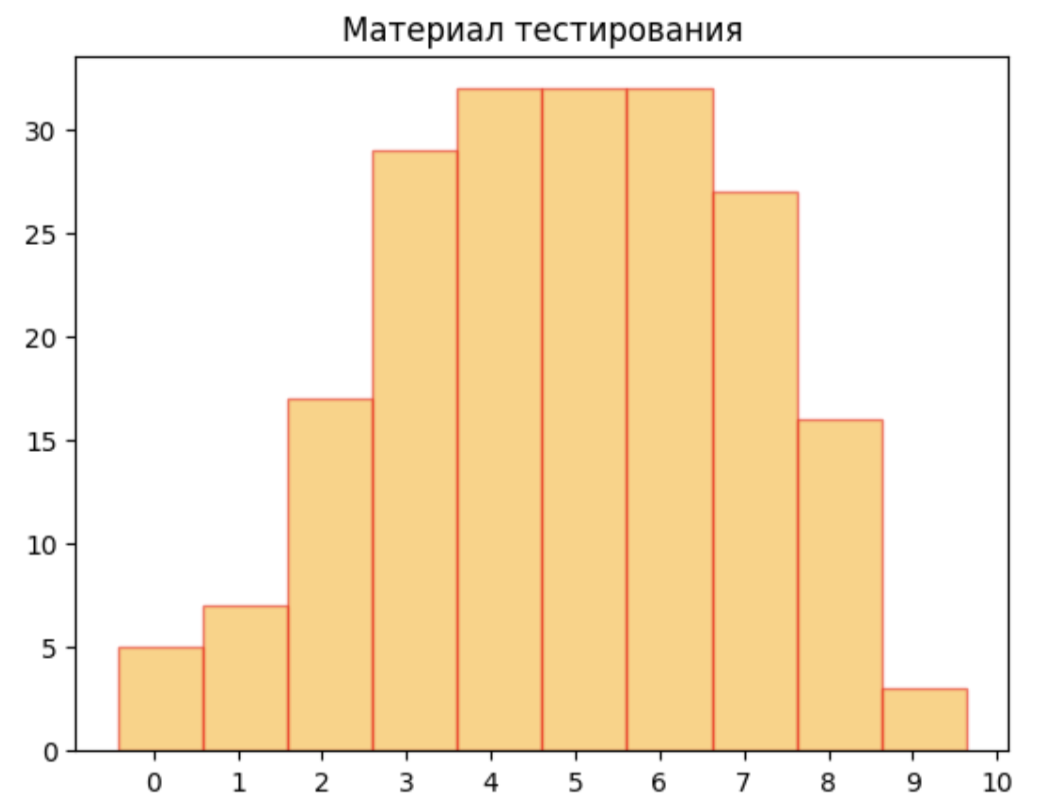
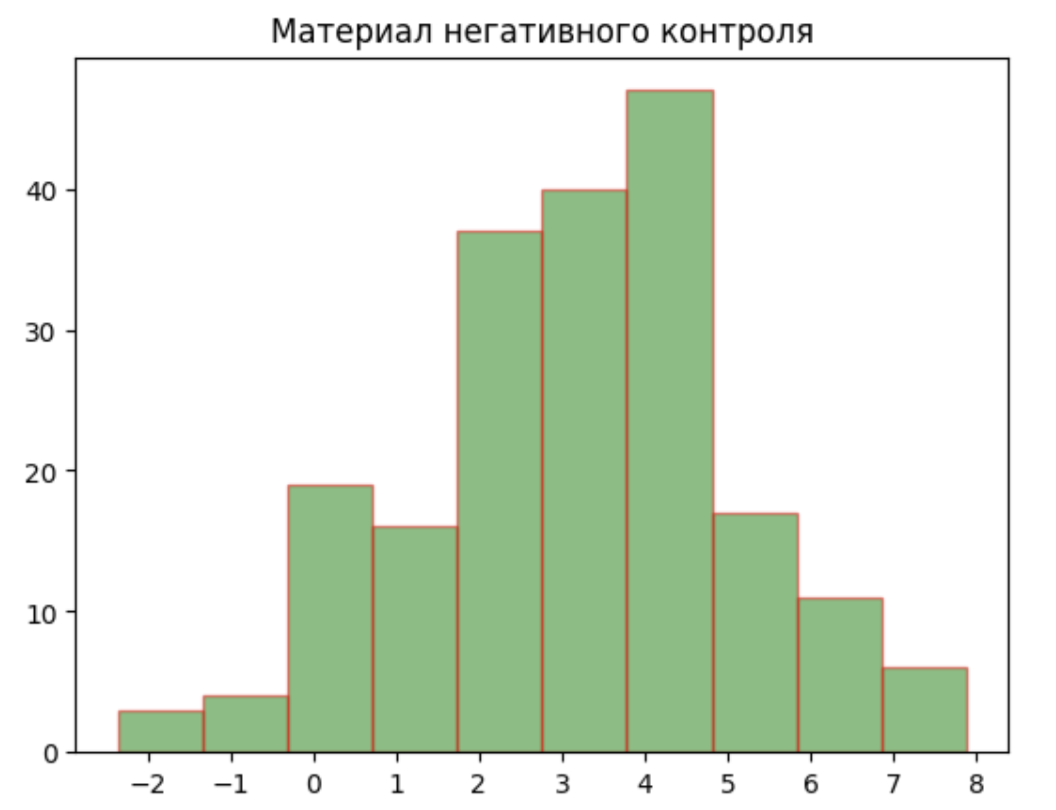
Я выбрала порог веса, равный 4, так как на гистограмме отрицательного контроля самый крупный пик находится на значении веса 4, и там же находится пик тестовой выборки.
Результат проверки представлен в таблице ниже:
| -сигнал | +сигнал | всего | |
|---|---|---|---|
| обучающая выборка | 27 | 73 | 100 |
| тестовая выборка | 72 | 128 | 200 |
| отрицательный контроль | 133 | 67 | 200 |
Построение информационной матрицы и LOGO
Вычисленное информационное содержание IC выравнивания
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| А | -0.093 | -0.112 | -0.103 | -0.103 | 0.234 | 0.121 | -0.093 | 1.761 | 0.000 | 0.000 | -0.121 | 0.022 | -0.135 |
| С | 0.022 | 0.022 | 0.165 | 0.165 | -0.068 | 0.185 | 0.185 | 0.000 | 0.000 | 0.000 | -0.046 | 0.145 | 0.055 |
| G | 0.270 | 0.315 | 0.107 | 0.165 | 0.227 | -0.068 | 0.292 | 0.000 | 0.000 | 2.286 | 0.671 | -0.021 | 0.485 |
| T | -0.103 | -0.103 | -0.093 | -0.121 | -0.154 | -0.135 | -0.157 | 0.000 | 1.761 | 0.000 | -0.154 | -0.103 | -0.146 |
| IC(j) | 0.096 | 0.122 | 0.076 | 0.106 | 0.239 | 0.103 | 0.227 | 1.761 | 1.761 | 2.286 | 0.350 | 0.043 | 0.259 |
Посмотрим на logo из тестовой выборки:
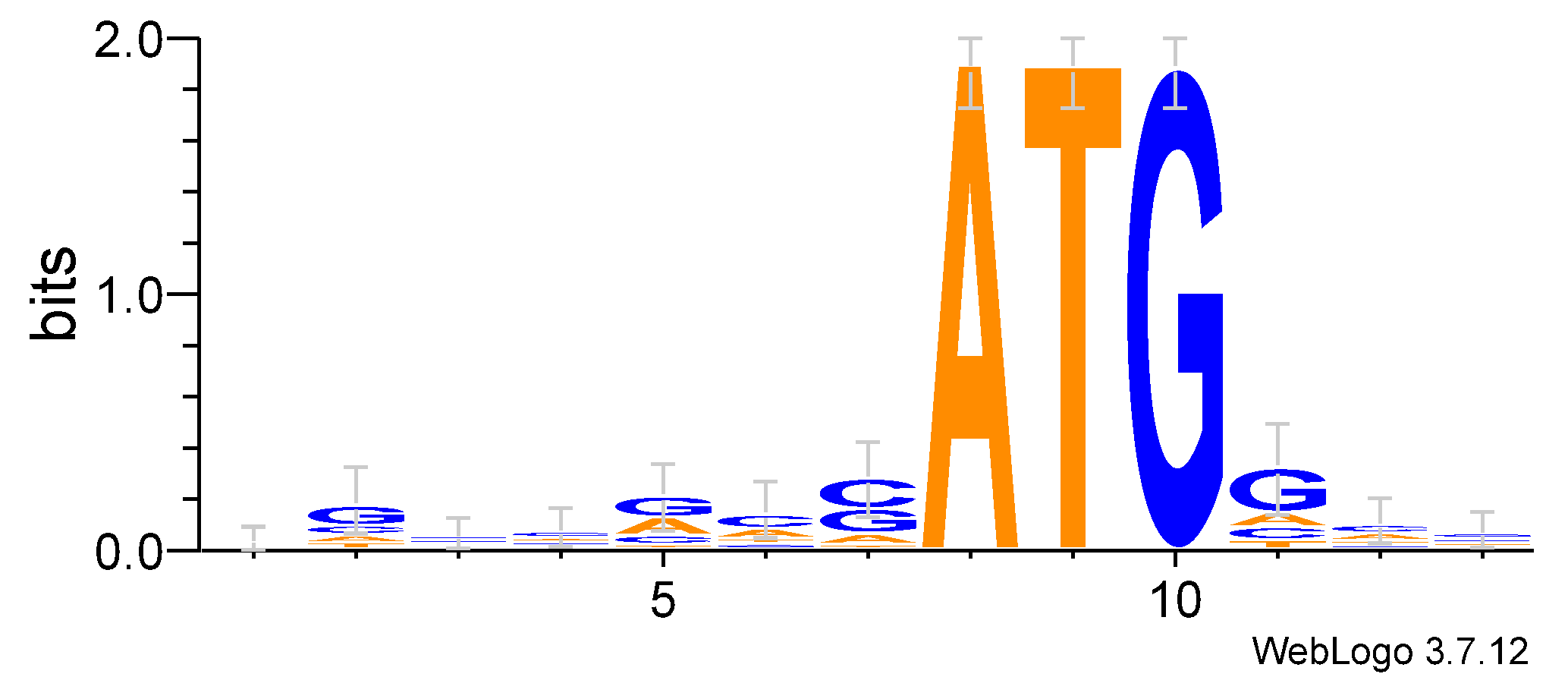
Очень хорошо видно консервативные ATG, при этом также наиболее заметными являются позиции, окружающие ATG (5-7 и 11).