PyMOL: электронная плотность
Задание 1. ЭП: хорошая и плохая расшифровки
Мне достались два PDB айдишника: 7BE3 и 2NMN. Это две кристаллографические расшифровки сахаросвязывающего белка галектина-3. Поподробнее про каждую расшифровку:
| 7BE3 | 2NMN | |
|---|---|---|
| Разрешение | 1.25 Å | 2.45 Å |
| Дисахарид | LacdiNAc | β - Lactose |
| Характеристики белка | 138 АК (16.29 kDa) | 138 АК (16.04 kDa) |
| Доп лиганды | глицерол, хлор | - |
Посмотрим на структуры:
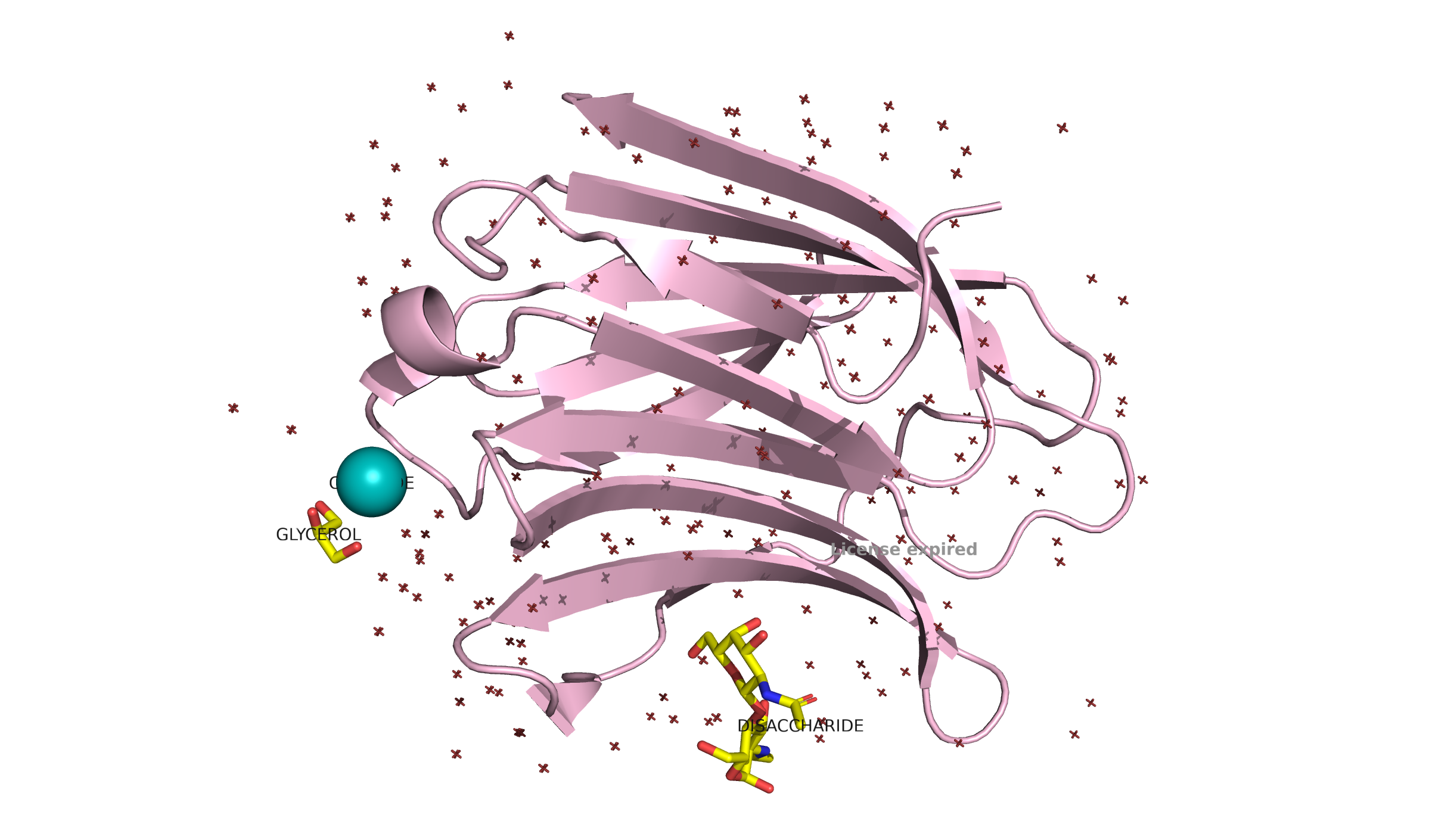
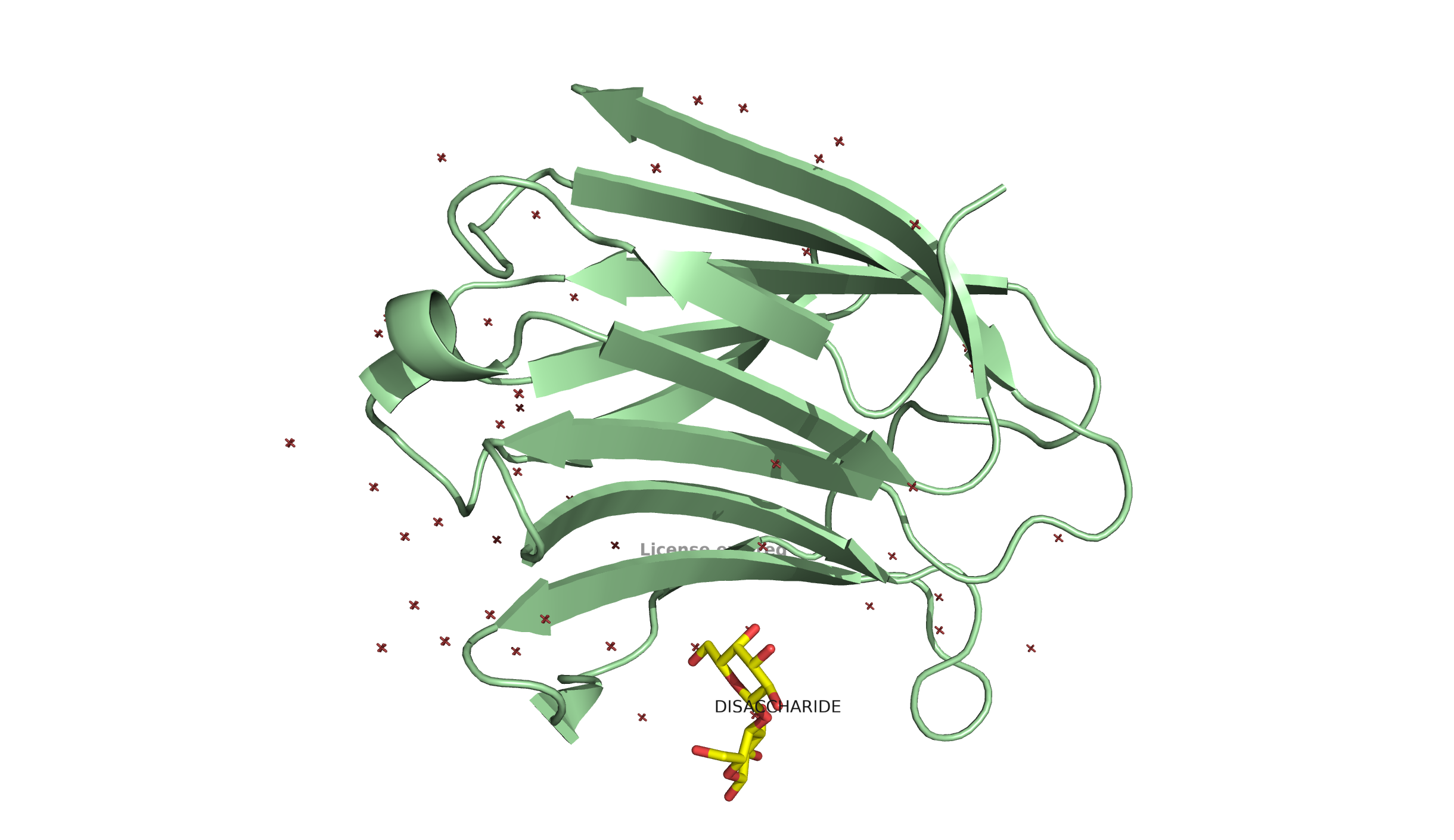
Чтобы визуально оценить, насколько сборки похожи, я убрала лиганды и воду и выровняла сборки.
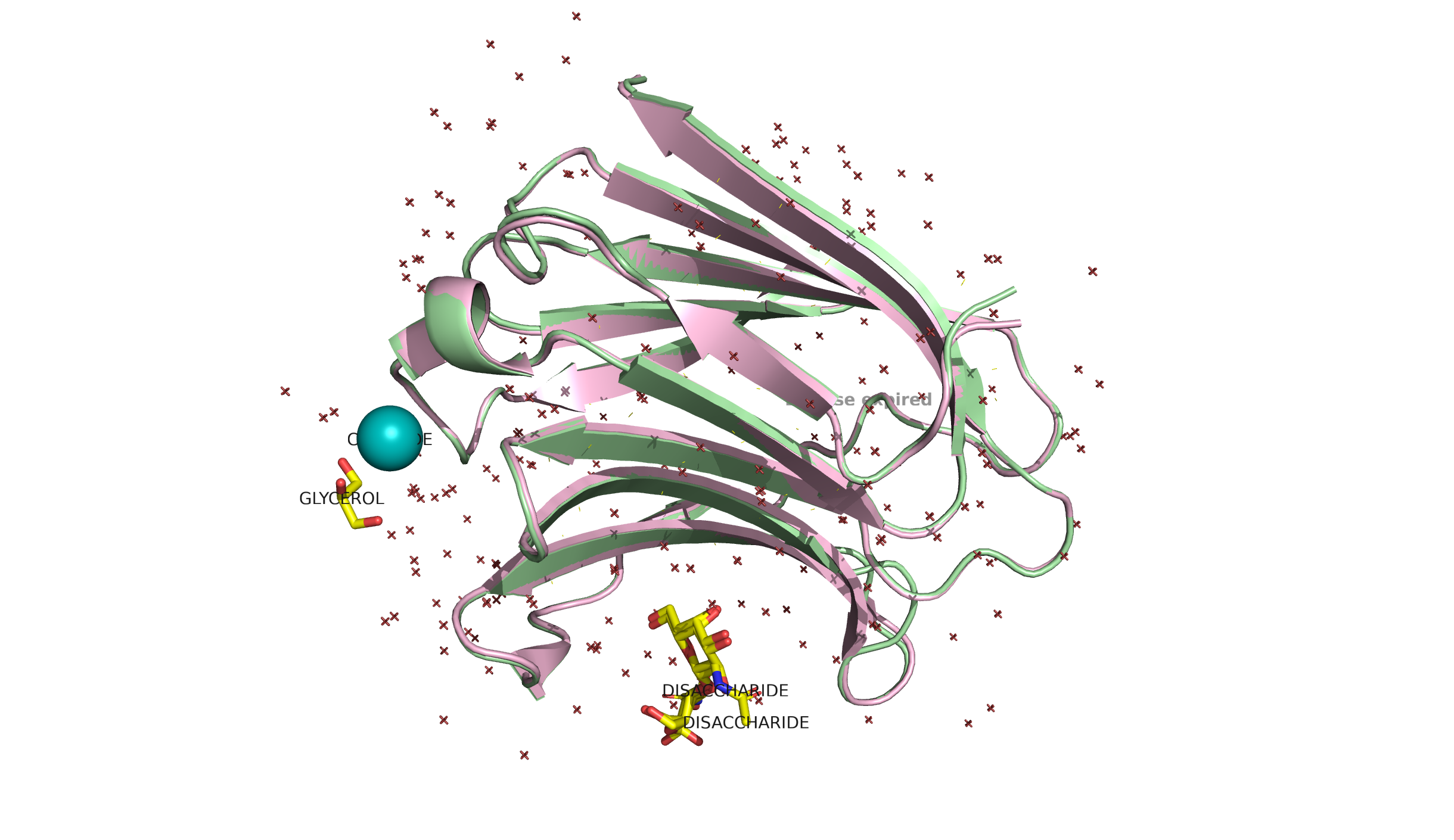
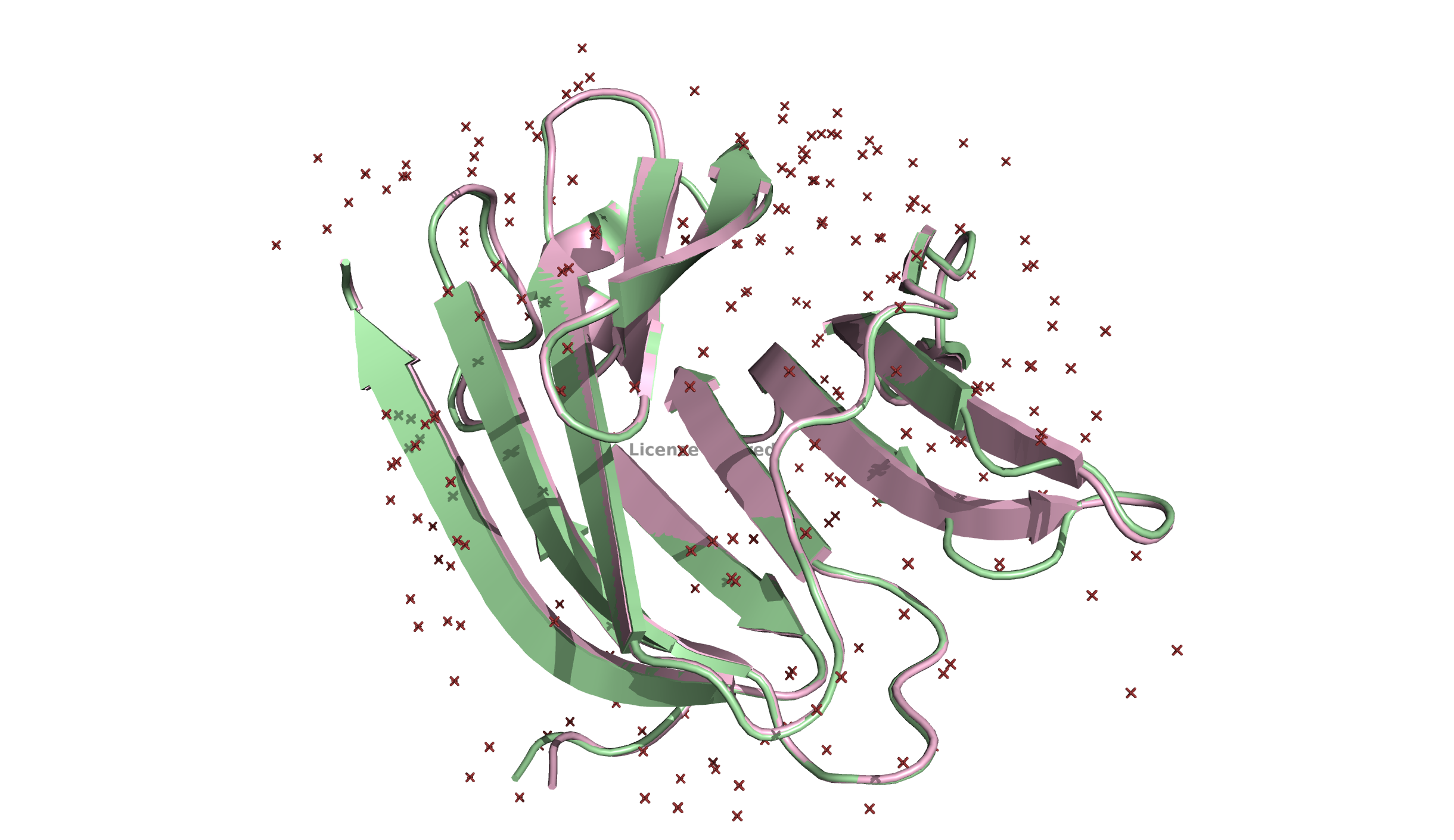
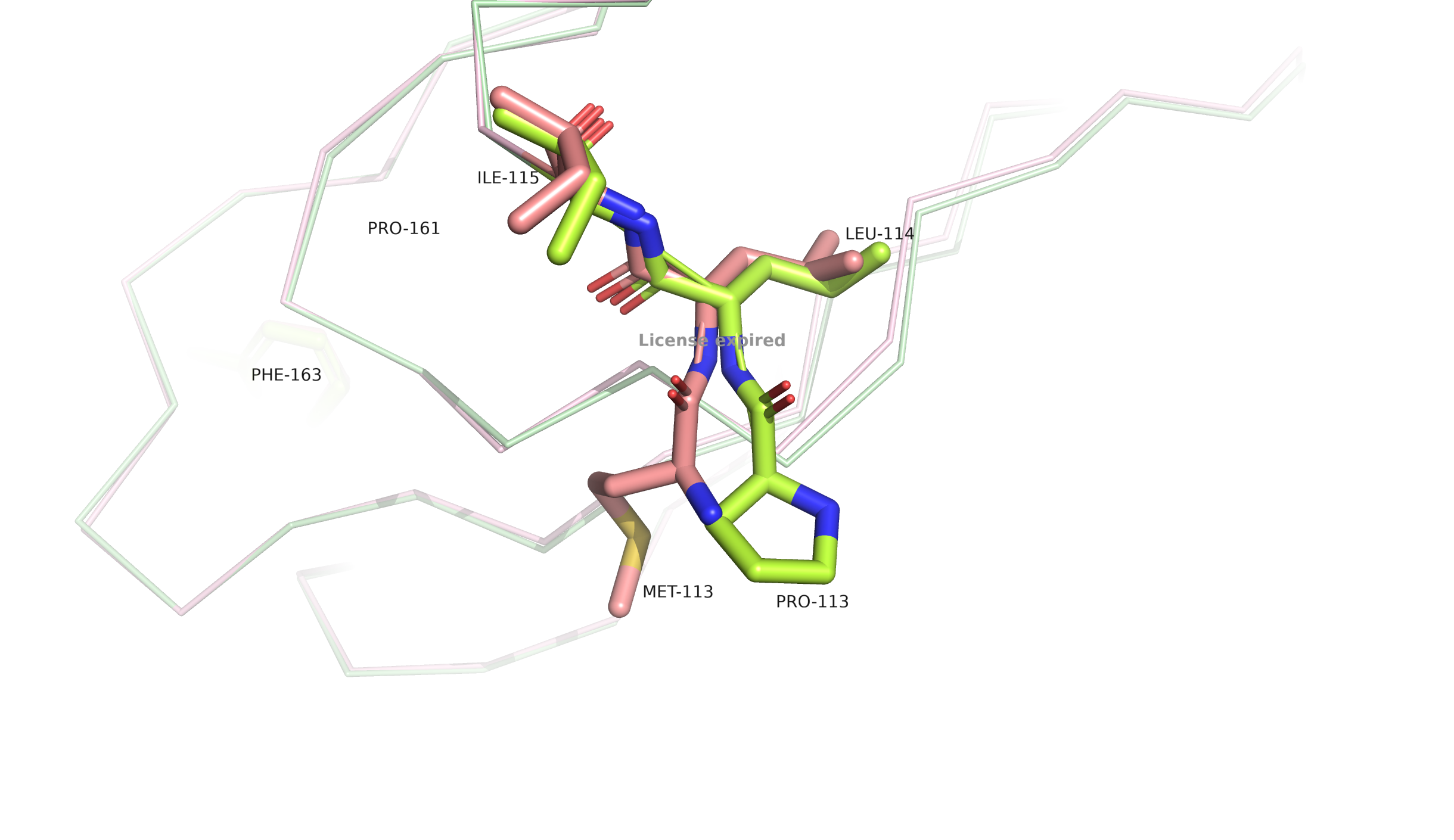
По рисунку 3 видно, что расшифровки хорошо согласуются друг с другом и по вторичной структуре белков, и по расположению лигандов. Единственный участок, который визуально можно различить между сборками, показан на рисунке 4 справа. У сборки с высоким разрешением β-листы длиннее, а на их месте у сборки с низким разрешением находится петля. Ниже представлены фото всех АК, входящих в составэтого участка.
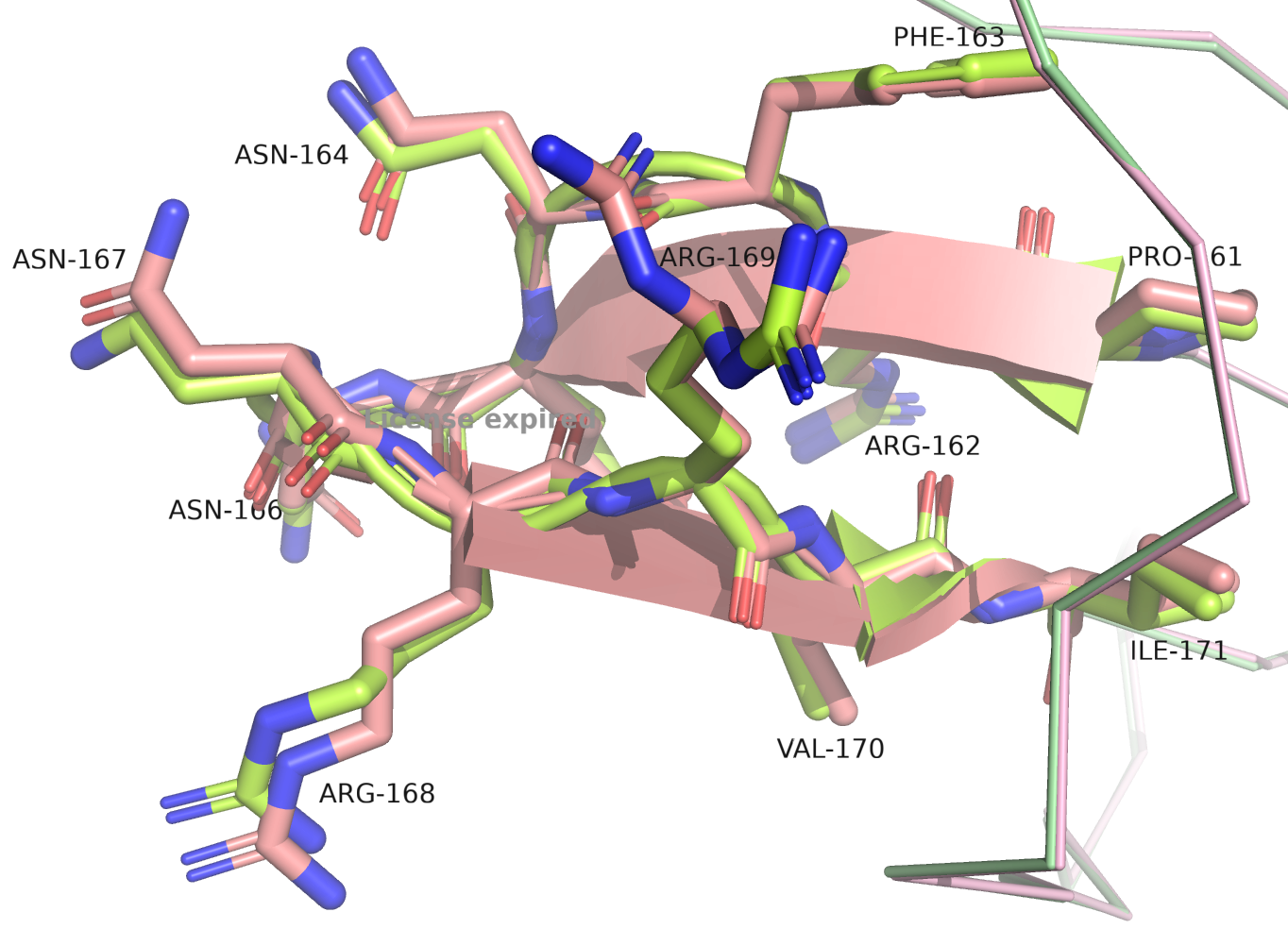
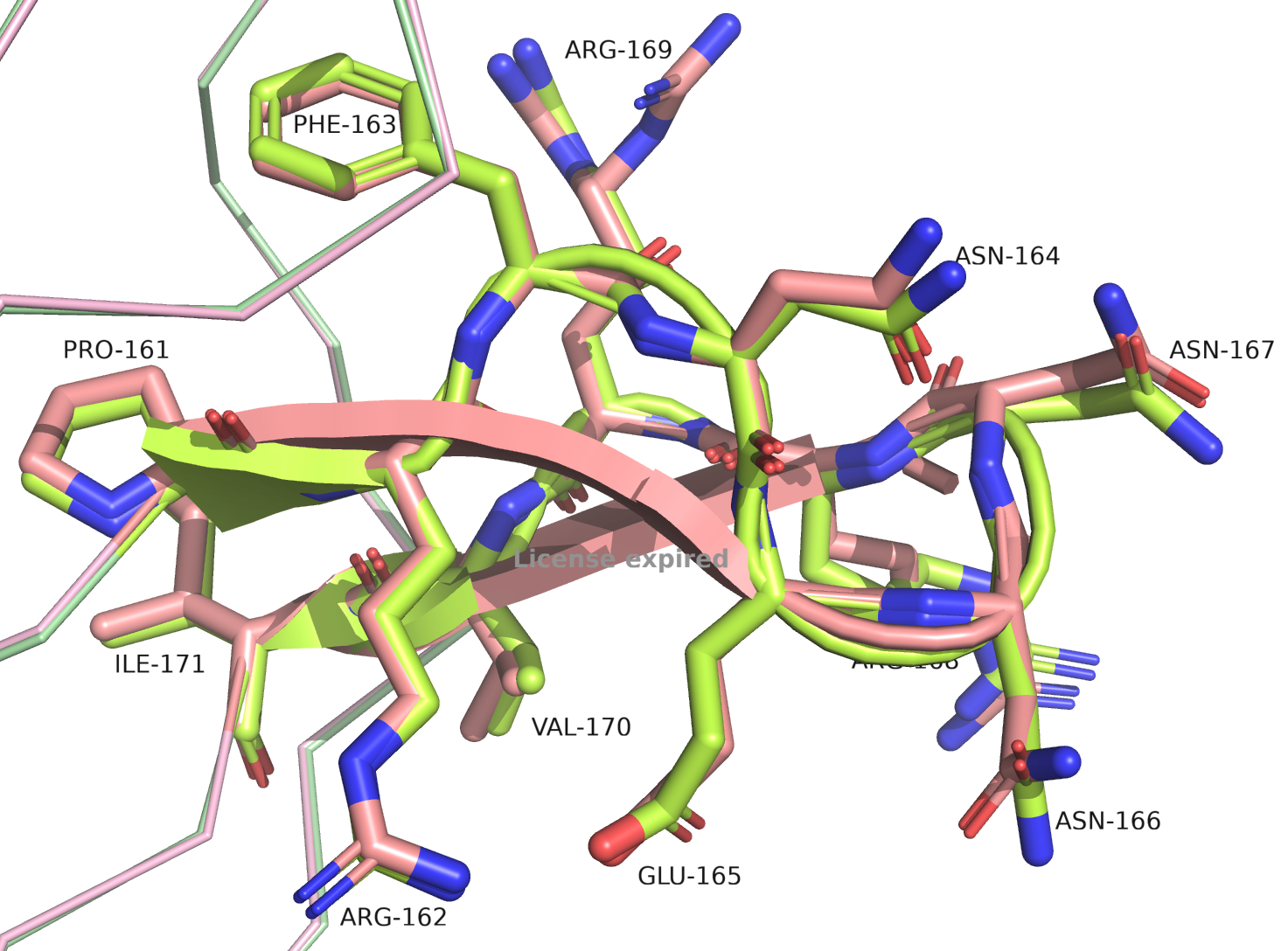
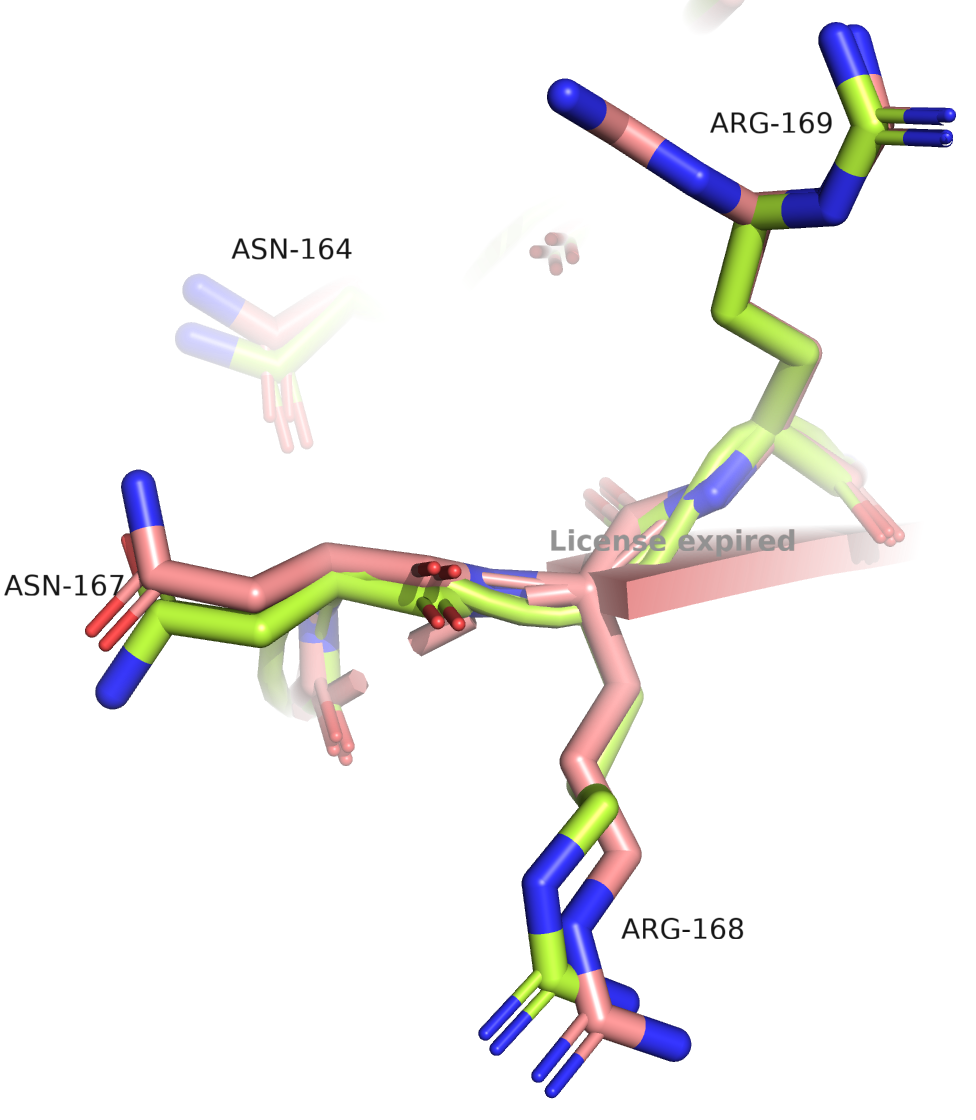
На рисунках выше я постаралсь показать все АК в составе query-участка. Я специально не убирала весь белок в виде ribbon, чтобы визуально можно было еще раз удостовреиться в общем соответствии структур. Для большинства АК участка мы видим неплохую (ASN-164, ARG-168 - боковые радикалы слегка подвинуты друг относительно друга) и даже очень хорошую (PRO-161, ARG-162, PHE-163, GLU-165, VAL-170, ILE-171 - почти полное совпадение) согласованность расположения. Чуть похуже согласованы расположения ASN-166, ASN-167 ("перевернутые" относителное друг друга амидные группы. Но тут подряд идущие ASN, возможно из-за стабилизации такое наблюдаем). Самый интересный - ARG-169. На самой правой картинке сверху показаны одни из самых отличающихся остатков с ARG-169 в центре. Мы видим альт-локи для аргинина 169 в 7BE3 сборке (выше разрешение). Один из альт-локов совпадает с положением ARG-169 в сборке 2NMN.
Для остова этого участка я посмотрела электронную плотность для каждой сборки на трех уровнях подрезки: 1, 2 и 3.
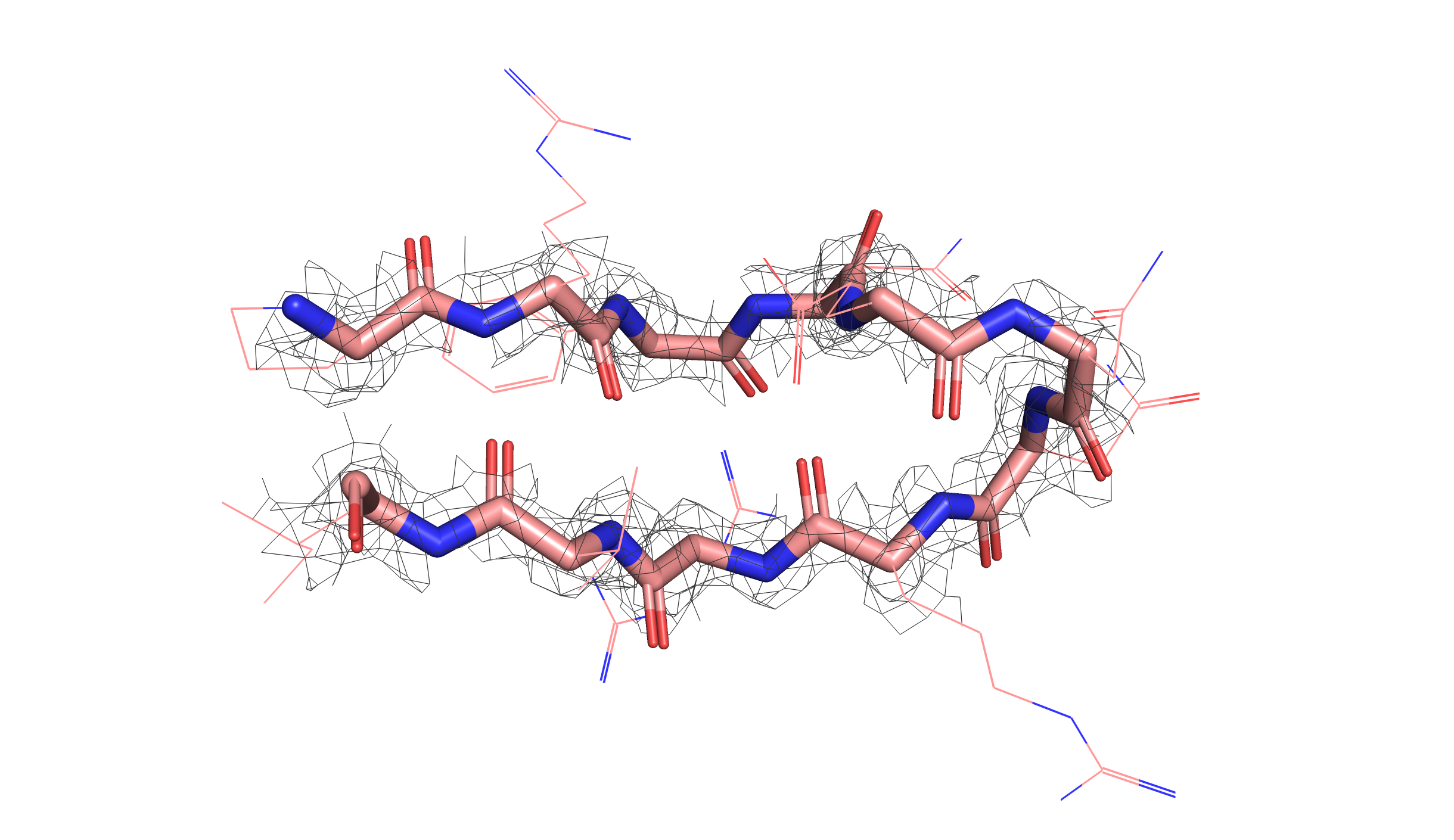
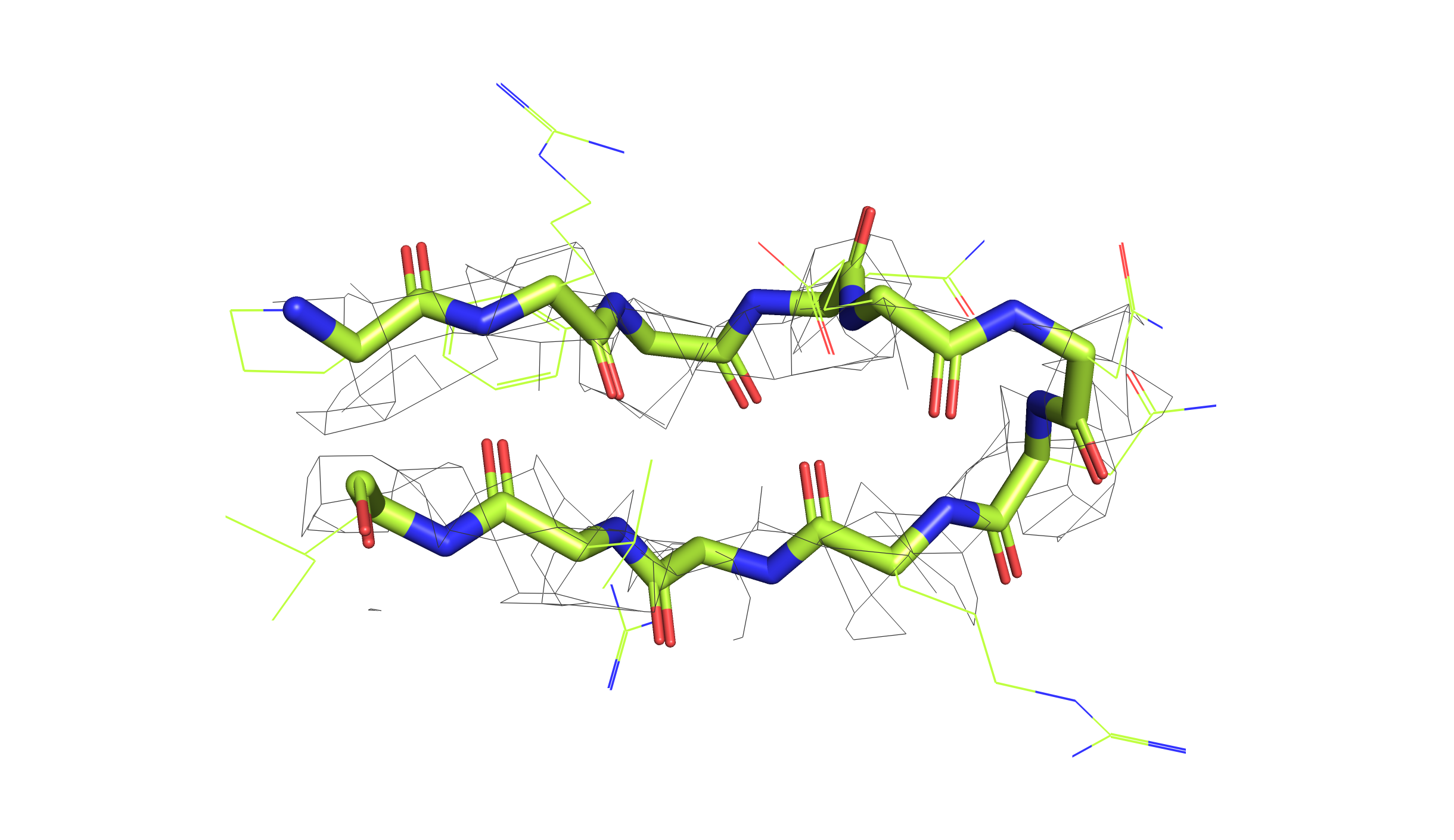
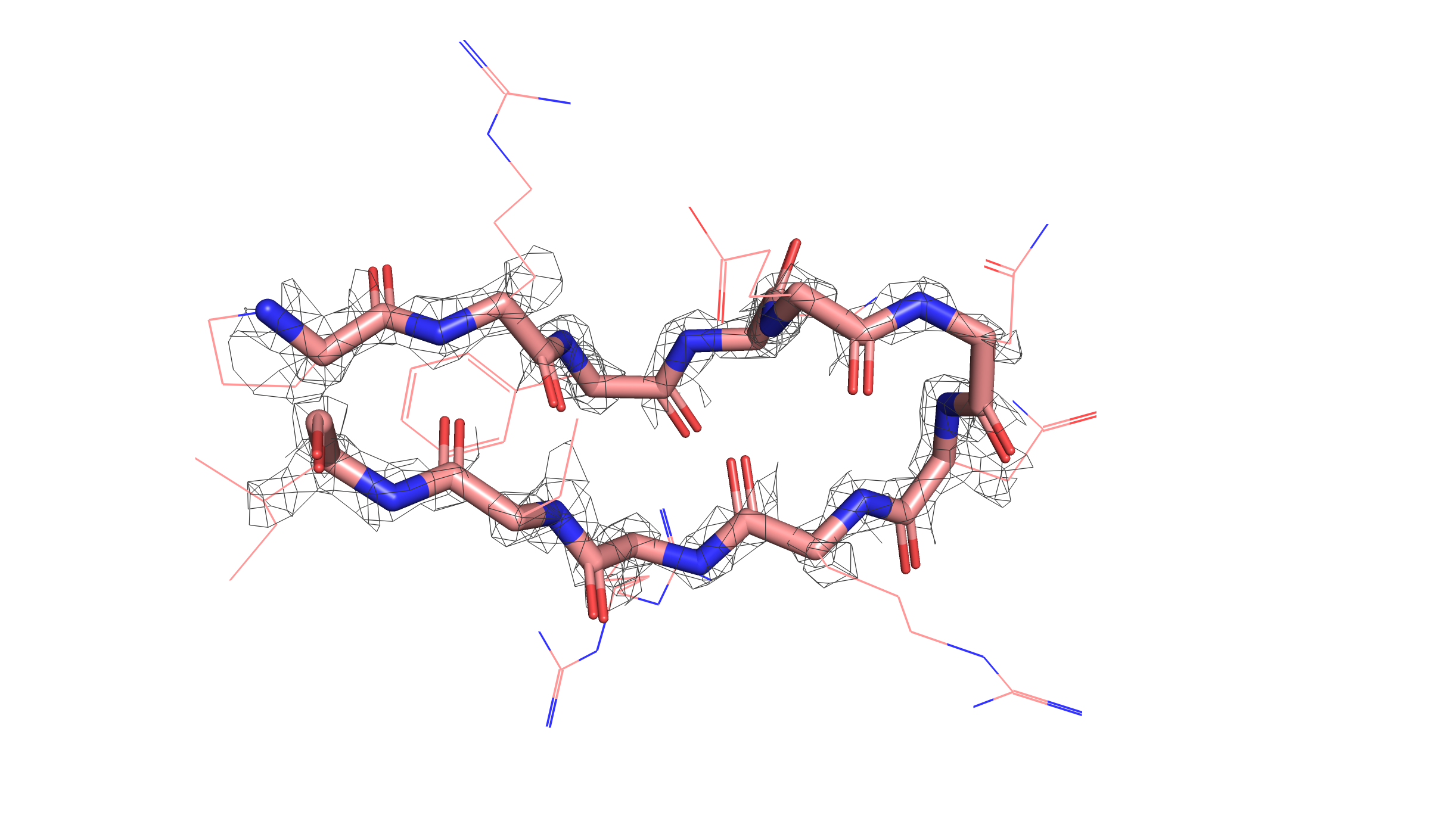
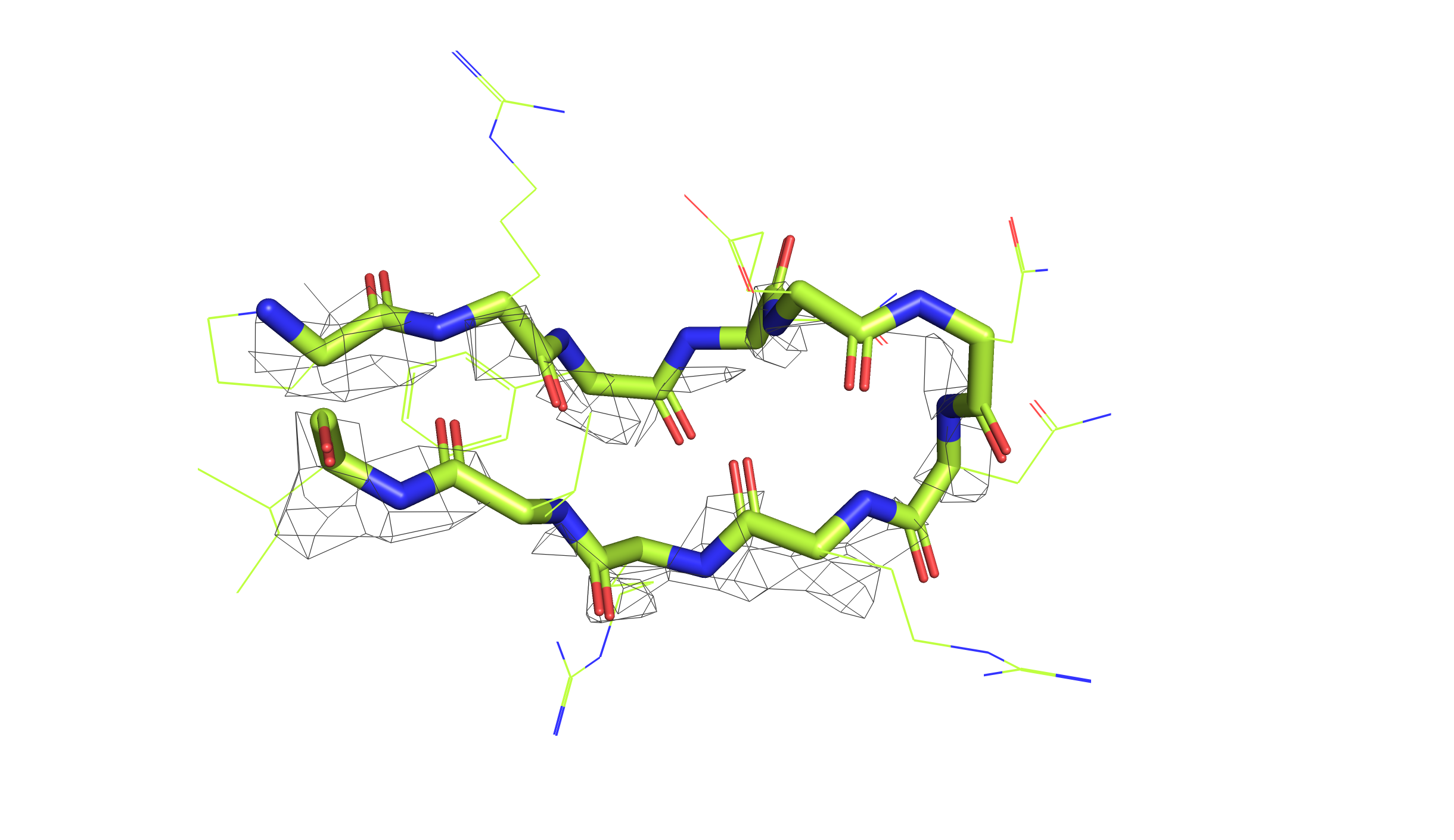
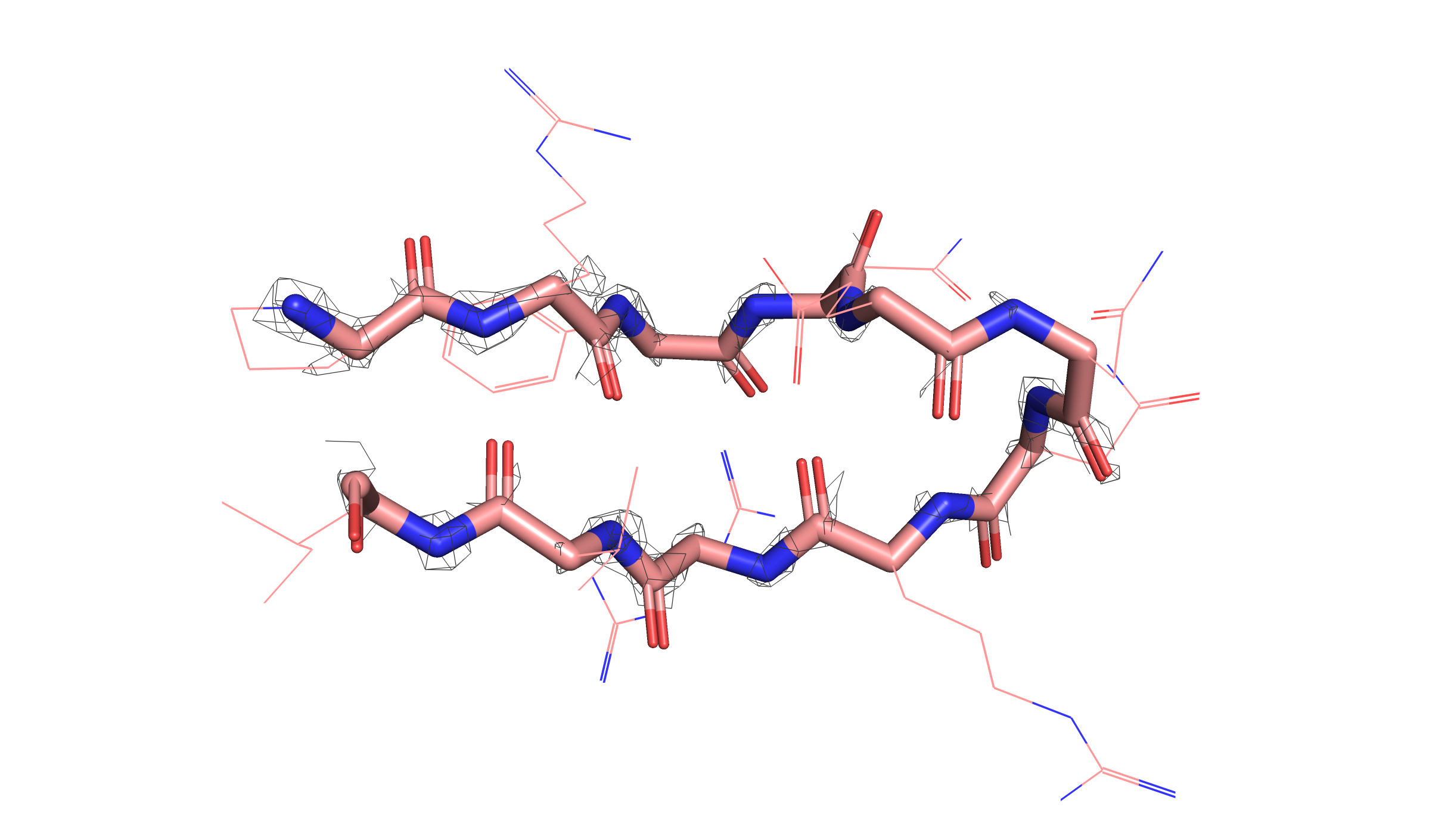
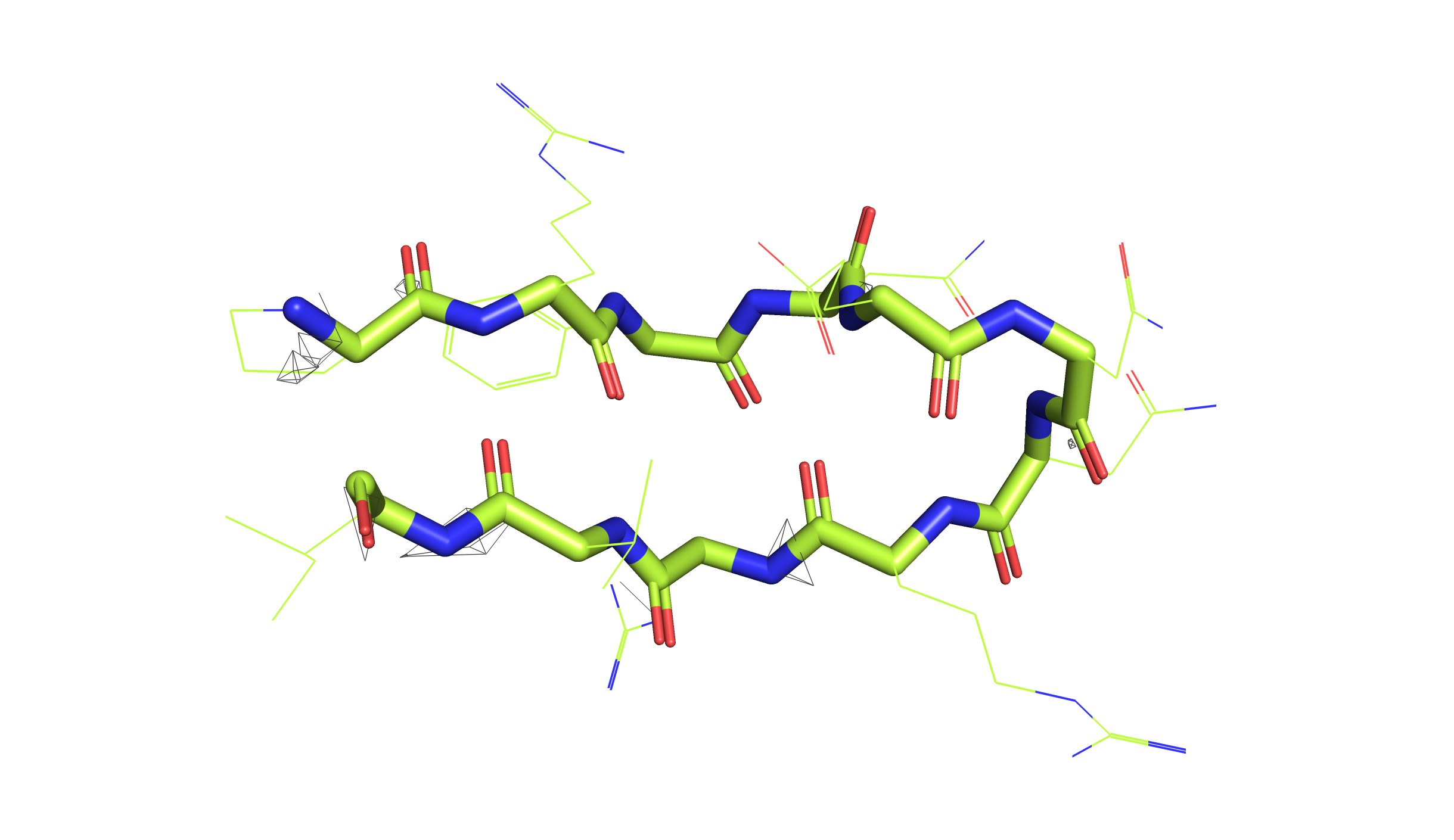
Мы видим, что электронная плотность расшифровки с более высоким разрешением (левые картинки, розовая структура, 7BE3) хорошо согласуется с остовом. Для структуры худшего разрешения (правые картинки, зеленая структура, 2NMN) электронная плотность выглядит как "примерная область, в которой находится main chain", при этом сложно сказать, что даже по ЭП первого уровня мы можем однозначно определить структуру остова. Для уровня подрезки 3 в случае 7BE3 мы видим ЭП как "шарики" идеально вокруг атомов остова. В случае 2NMN на всех уровнях подрезки, в принципе, мы видим какие-то геометрические фигуры, далекие от идеального окружения атомов.
Очевидно, что у 7BE3 разрешение выше - мы видим это по лучше упорядоченному распредлению ЭП. Этот вывод подтверждается данными PDB (см. таблицу выше). Ну и в конце можно сказать, что я бы доверилась первой структуре (7BE3) и определяла расположение β-листов (да и всего белка в принципе...) именно по этой сборке.
* мы видим, что масса структур различается. Эти различия можно увидеть в еще одном не совсем совпадающем участке двух расшифровок: на N-конце белка, в самой первой позиции под номером 113 у этих сборок находятся рахные АК: для 7BE3 определили Met, а для 2NMN - Pro. Как написано в задании, "Эти различия не стоит рассматривать как следствия из разного качества моделей.", но просто интересно
Задание 2. ЭП и положение в структуре
Мне досталась структура 2YHD - расшифровка рецептора андрогена. Структура выявлена для белка в комплексе с лигандом - тестостероном, и ингибитором. согласно PDB, разрешение этой расшифровки составляет 2.20Å, структура состоит из 249 АК (29.71 kDa)
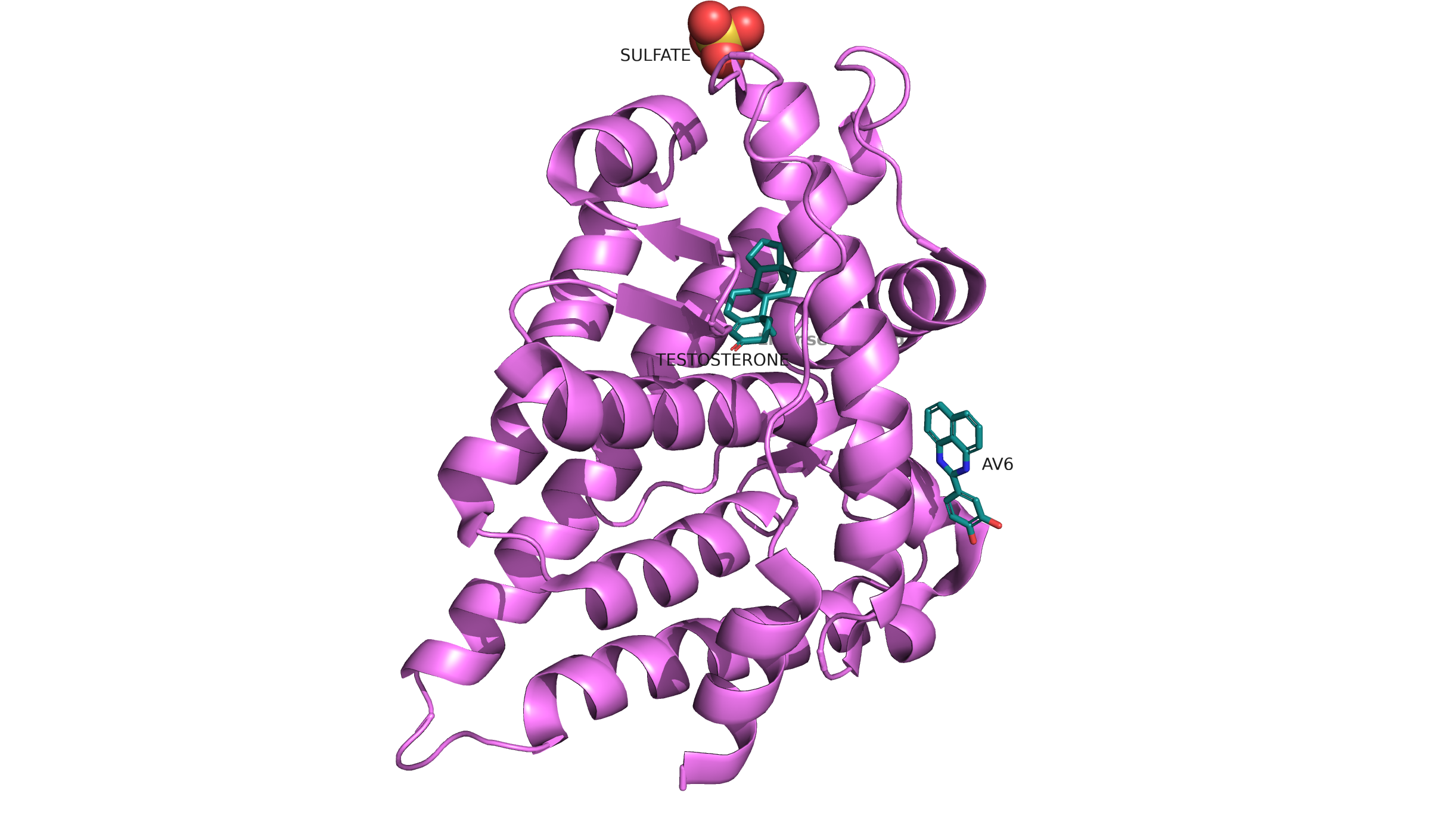
Теперь посмотрим ЭП с помощью mesh на уровнях подрезки 1, 2 и 3.
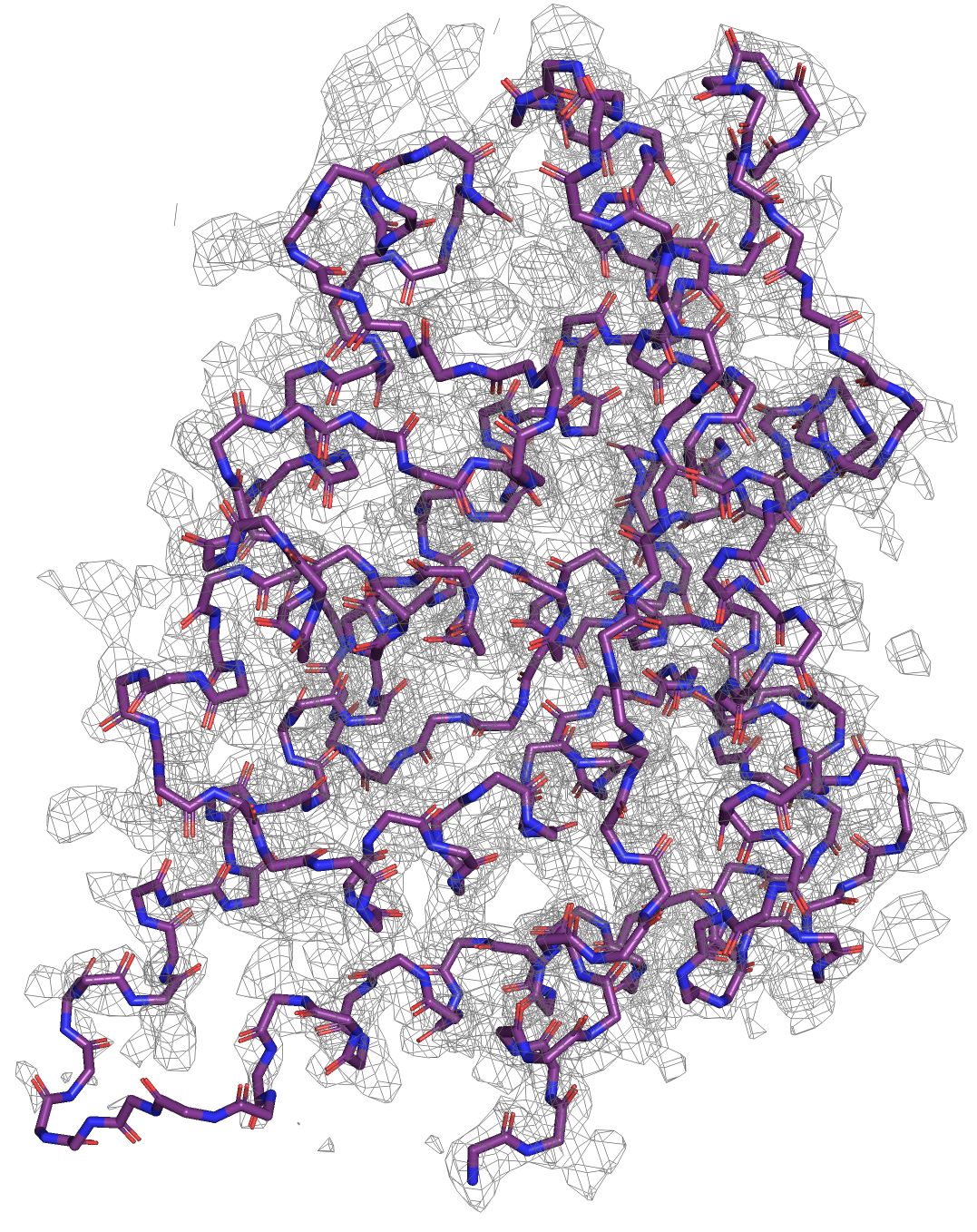
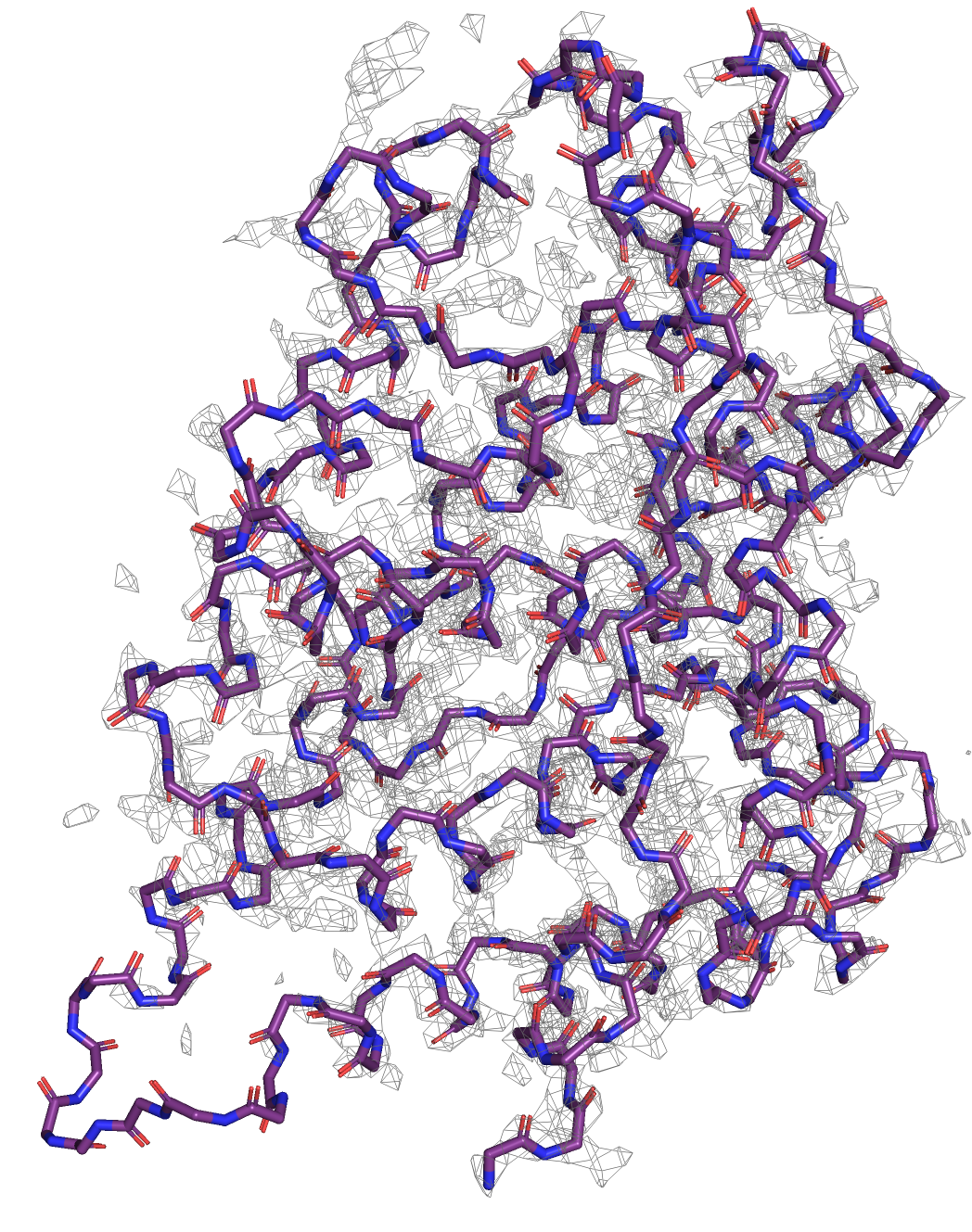
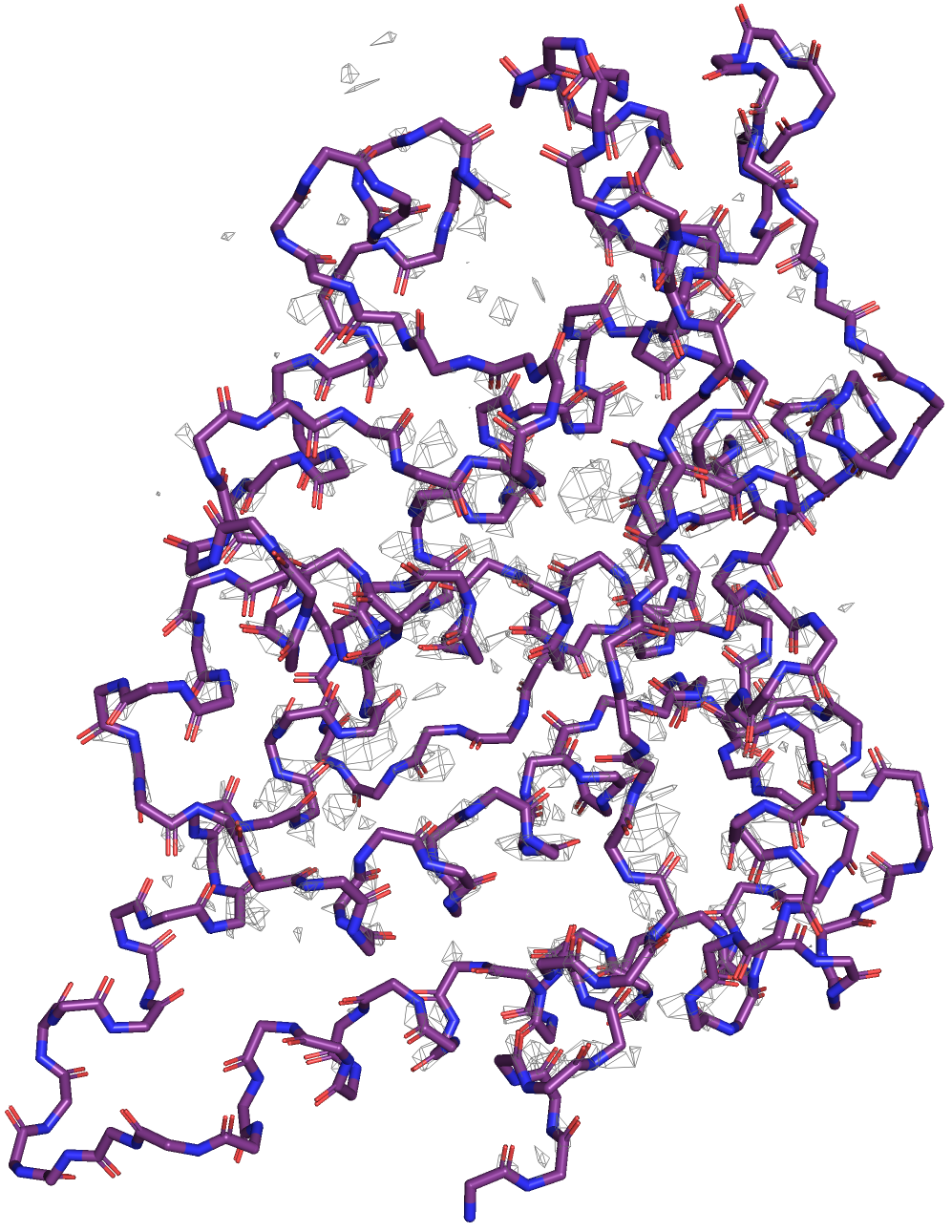
Мы видим как по мере увеличения уровня подрезки ЭП "уходит" с наших графиков. На уровне подрезки = 1 ЭП покрывает весь наш белок: мы видим ее практически на всех атомах и на всех элементах вторичной структуры. Уже при уровне = 2 видно, как ЭП "ушла" с участков белка, не имеющих вторичной структуры (у нас рецептор только с α-спиралями, поэтому с петлей между α-спиралями ЭП исчезла, а на самих спиралях еще осталась). Ну и на уровне подрезки 3 ЭП сосредоточена вокруг некоторых атомов остова α-спиралей. Можно заметить, что ЭП в основном расоположена на N и O в составе пептидной связи. Сα атомы обделены ЭП на уровне подрезки = 3.
Задание 3. Альтернативные положения
В этом задании я рассмотрю альт-локи GLU-114 в структуре 7KR0 (структура домена белка NSP3 коронавируса SARS-CoV-2). Этот остаток находится на α-спирали, поэтому на рисунке ниже я ее оставила. Взаимодействие альт-локов GLU-114 возможно с боковыми радикалами АК той же α-спирали: ASN-117 для альтлока А и LYS-110, SER-111 для альтлока B

ВАЖНО: тут две связи от ASN-117 до кислородов GLU'A-114 -- это НЕ значит, что водородные связи на каждый кислород. Я просто не знала как показать, что остаток GLU будет ионизирован и ЭП будет равномерно распредлена между этими двумя кислородами остатка (резонанс). Поэтому дополнительно напишу: между ASN-117 и GLU'A-114 - водородная связь (2.9-3.4 Å); между SER-111 и GLU'B-114 тоже возможно образование водородной связи (но я не уверена, расстояние от -OH серина до карбоксильной группы GLU'B-114 составляет 4.3-5.7 Å, на самом деле это как будто многовато) + ионные взаимодействия между отрицательно заряженной карбоксильной группой GLU'B-114 и положительной заряженным остатком LYS-110 (2.9-3.3 Å).
Таким образом мы видим по одному хорошему стабилизирующему взаимодействию (водородная и ионная связь соответственно), а в случае GLU'B-114 есть еще возможность образования водородной связи с остатком серина. Мне кажутся эти взаимодействия более-менее эквивалентными для стабилизации расположения аль-лока. Подтверждением этой мысли мы видим на рисунке - я подписала населенность (occupancy) на карбонильных атомах аль-локов. Для A альт-лока (1 водородная связь) населенность = 0.45, а для B альт-лока (1 ионная связь + слаббая водородная) = 0.55, что почти одно и то же, с небольшим предпочтением в сторону двух стабилизирующих связей. Населенность еще подписала для LYS-110 и SER-111, в случае LYS-110 ионная связь возможна между GLU'B-114 и любым альт-локом LYS-110, а вот для слабой водородной связи с SER-111 возможно взаимодействие только с одним альт-локом с населенностью = 0.6
Задание 4. B-фактор
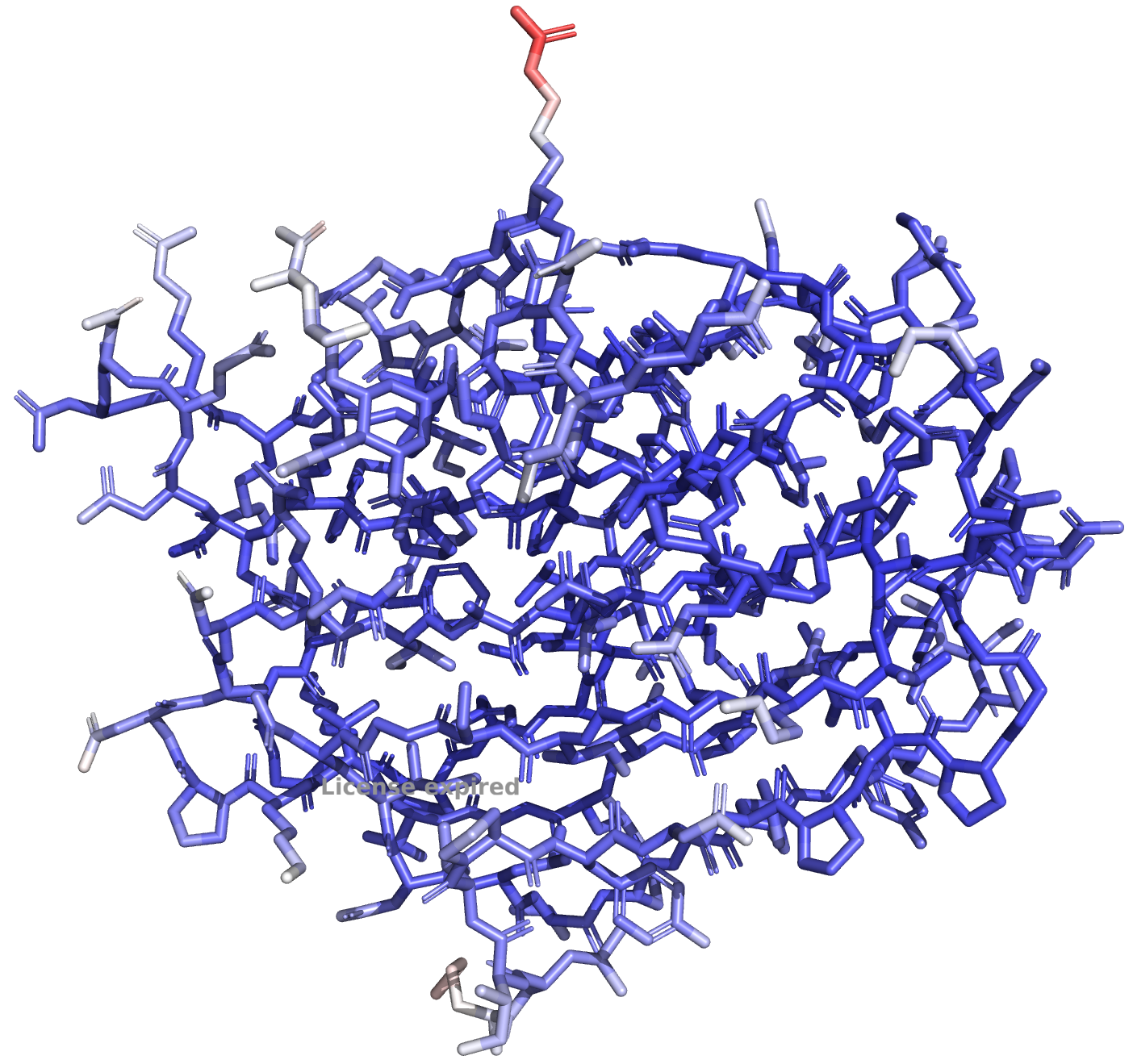
Продолжаю работать с 7KR0. Сначала покрасила по B-фактору только остов белка (Рис 17), а затем весь белок (Рис 18)
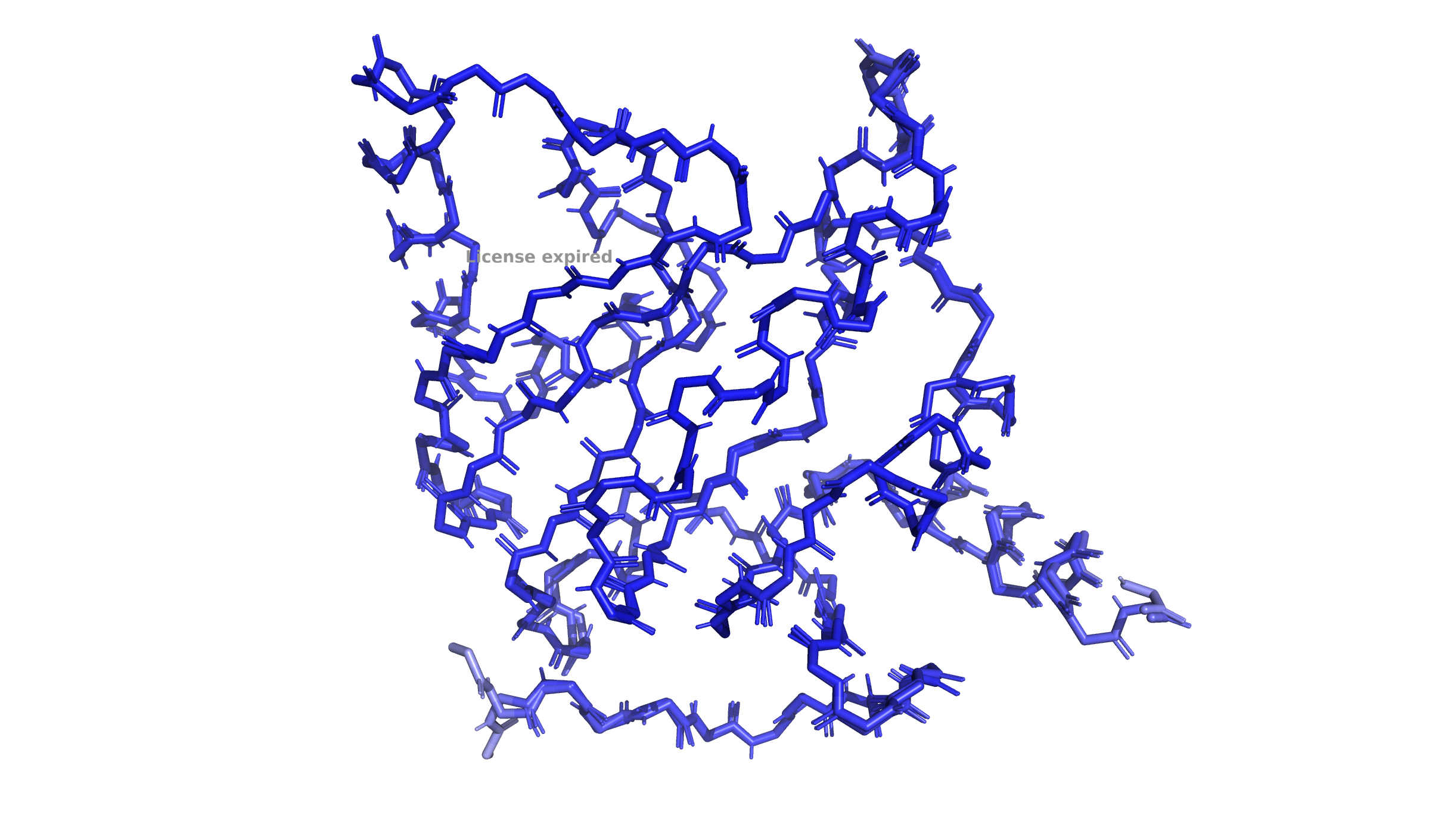
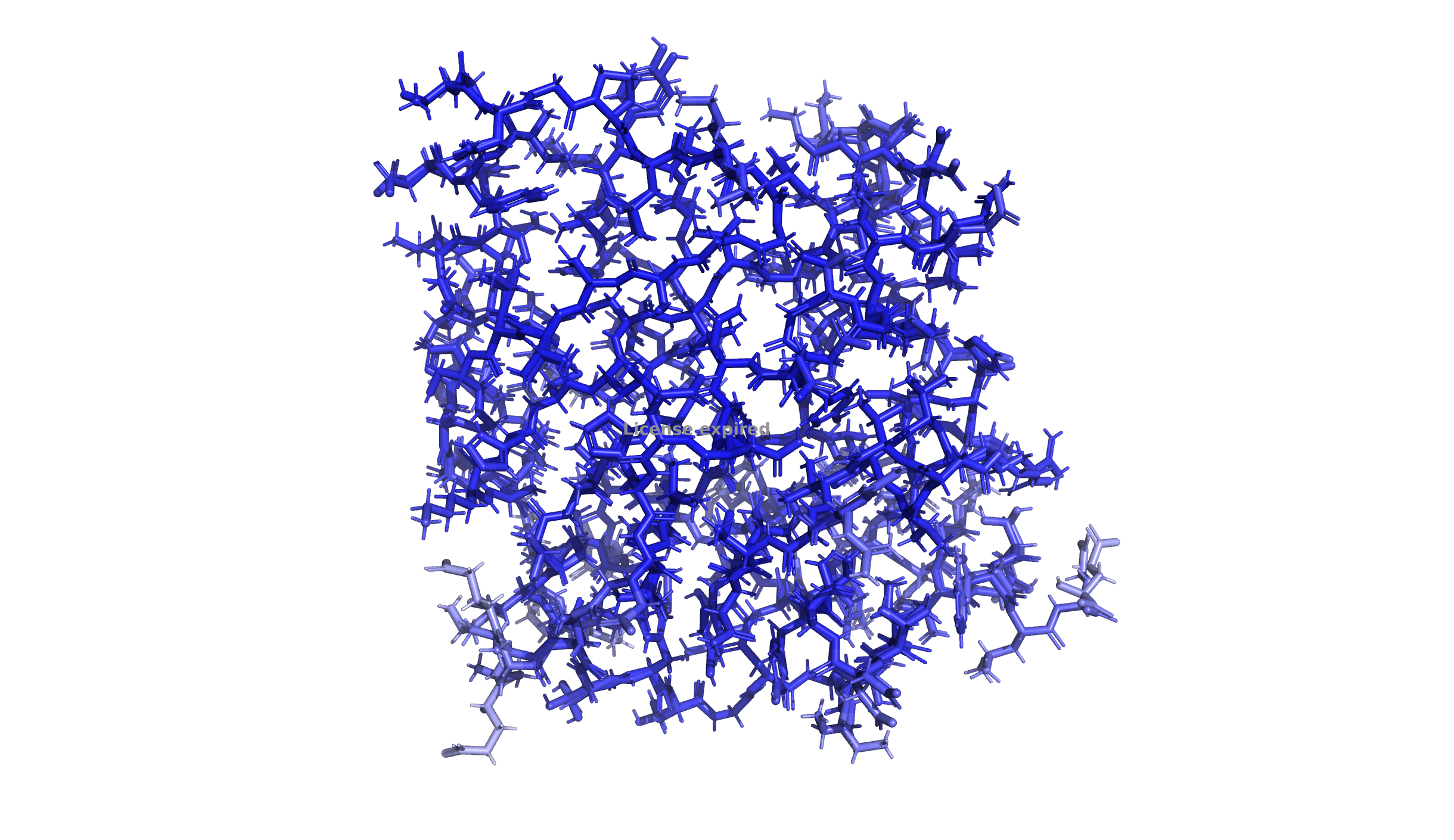
В данной структуре нет "красных" атомов с высоким B-факторов: практически весь белок темно-синий (то есть B-фактор низкий). Только методом пристального взгляда можно заметить остатки во внешних частях белка, которые находятся вне вторичых структур (то есть боковые остатки в наружних петлях белка), которые имеют голубой цвет. Я выбрала самый светлый участок. Он не красный, но такой светло-голубой... Ну и в добавок на этом участке находятся альт-локи не просто АК, а целых "дипептидов". Смотрим его ниже:
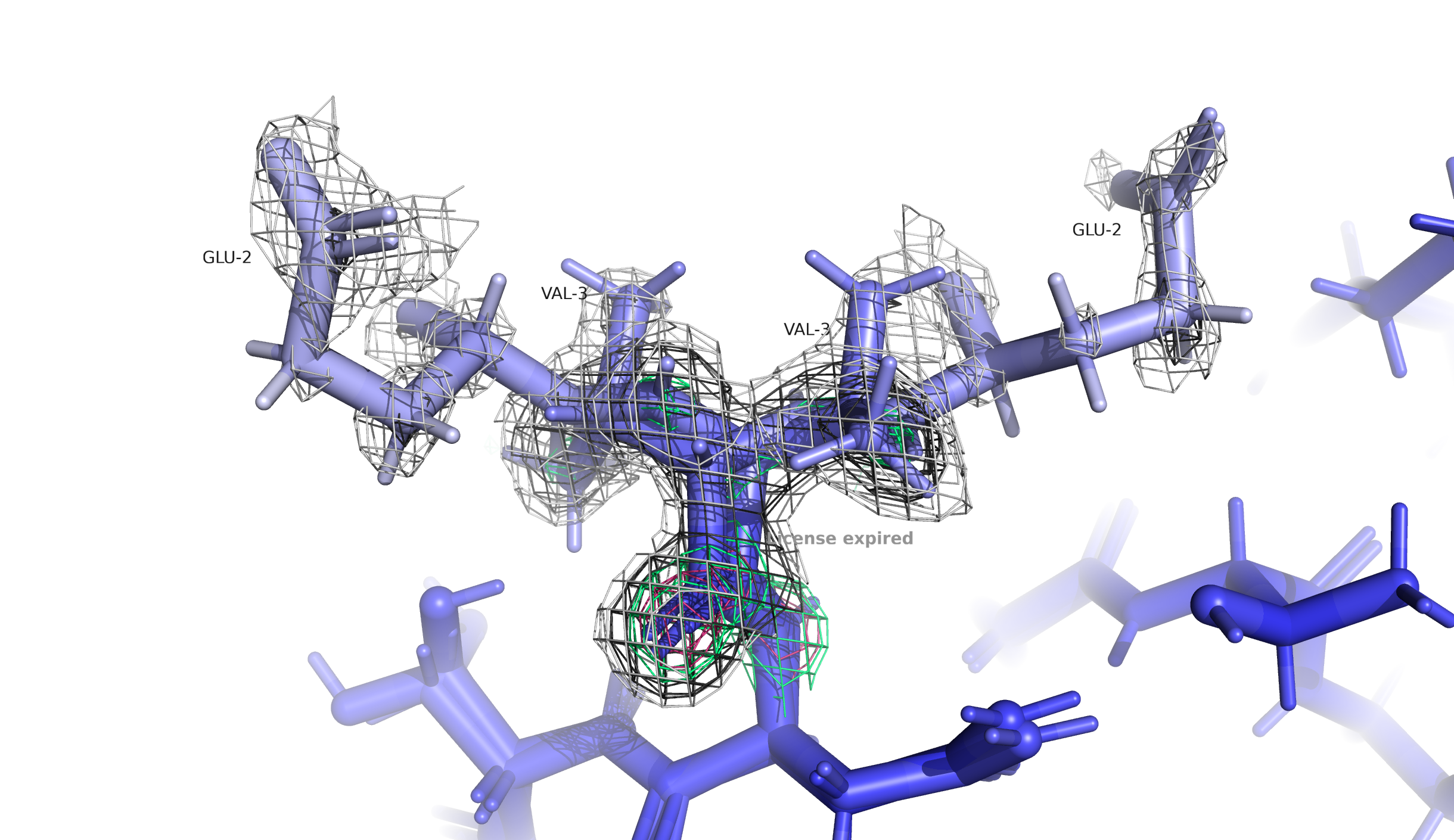
Альт-локи для GLU-2 -- VAL-3. Мы видим, что в частях, где B-фактор наибольший (по самым краям остатков), ЭП наименьшая (видим только при уровне подрезки 0.5). Поэтому B-фактор я бы описала примерно так: этот параметр показывает степень неопределенности расположения атома, возникающую вследствие его подвижности.
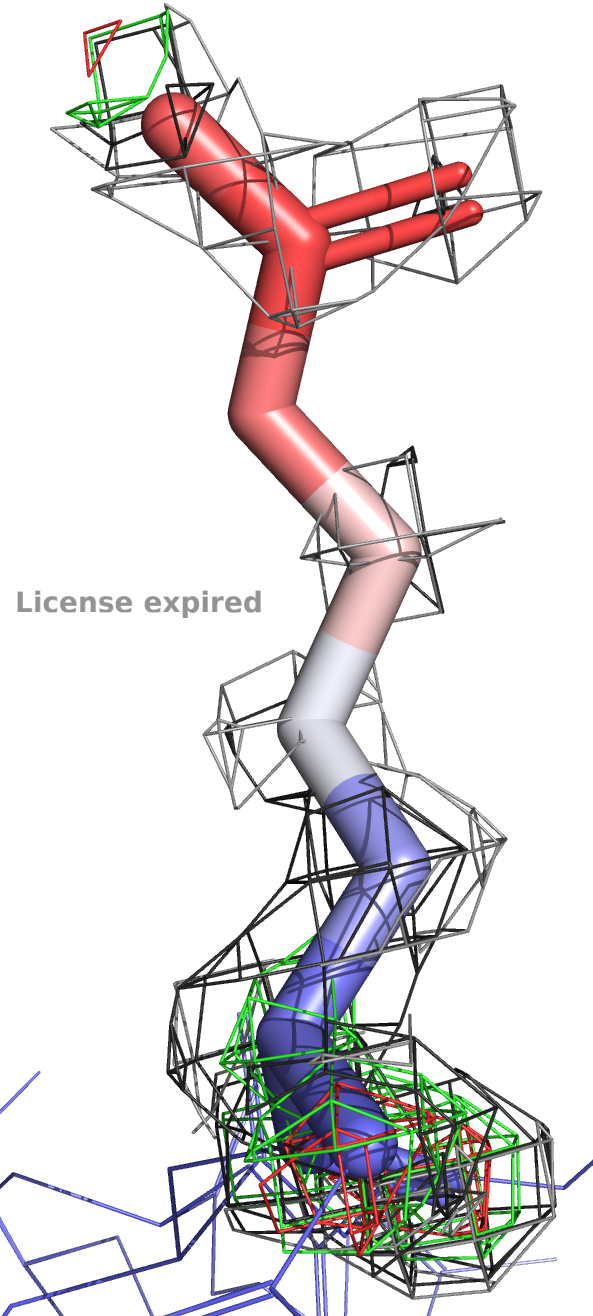
Я случайно замтеила, что в структуре из первого задания (7BE3, которая с лучшим разрешением), есть одинокий бродяга ARG, который единственный из всей структуры окраше красным. Я для подтверждения мысли покажу фотки снизу. По нему прям видно, как ЭП сьезжает вниз в область синих (низких) значений B-фактора