Протонирование
PROPKA
Структура, которая мне досталась - 5FBF. Кристаллизация происходила при pH = 3.8
Эту структуру я закинула в PDB2PQR, скачала файлы .log и .pqr; в файле .log после строки "SUMMARY OF THIS PREDICTION" находится таблица, в которой содержатся данные о 25 остатках ASP, 14 GLU, 7 HIS, 5 CYS, 17 TYR, 10 LYS, 3 ARG. Среди ASP, GLU и HIS найдем остатки с pKa > pH кристаллизации, возьмем по одному остатку каждого типа с наивысшим pKa:
| Position | pKa | model-pKa | |
|---|---|---|---|
| ASP | 83 | 3.80 | 6.69 |
| GLU | 147 | 4.50 | 9.03 |
В случае HIS максимальная предсказанная pKa была 6.3 при "норме" 6.5, поэтому остатки гистидина рассматривать не будем, ограничимся двумя - по одному для ASP и GLU
ASP-83
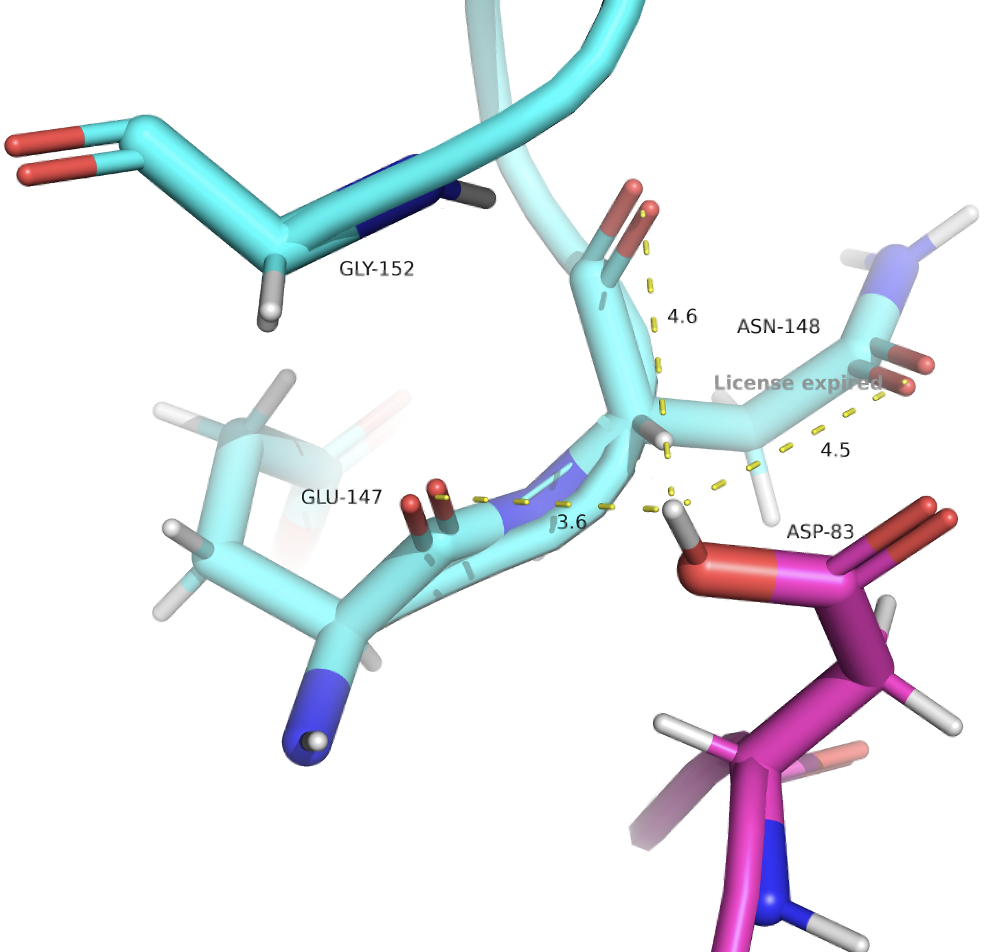

Посмотрим ASP-83. Я показала только те остатки, которые находятся в его окружении и могут в теории участвовать в образовании H-связей (на рисунке теоретические!!!). Согласно предсказанию программы, H сидит на "левом" атоме водорода в ASP-83. В таком случае ASP-83 выступает только в качестве донора водородной связи. На риснуке я отметила предполагаемых акцепторов, хотя отмечу, что правдоподобной является только связь с C=O пептидной связи GLU-147. Остальные связи слишком большие для образования стабильной водородной связи. Итого: в таком положении мы видим только одну стабильную водородную связь, где ASP-83 выступает в качестве донора. Плохо
Предположим, что модель угадала с фактом протонирования, но поставила водород не на тот атом. Я хз как удалить водород и переместить его на другой атом, но предположим, что водорд теперб сидит на "правом" кислороде остатка. В таком случае мы видим более стабильный набор связей, где ASP-83 выступает как в качестве донора (связь ASP-83 и ASN-148), а также может быть акцептором предположительно двух связей: в первой донором выступает GLY-152, во второй - GLU-147. Я все-таки больше поверю в связь с GLY-152, так как ее длина поменьше.
GLU-147

В данном случае все оч красиво получается. PyMOL сам присует связь GLU-147 с ASP-86. В целом мы видим, что здесь сам GLU-147 образуется 2 водородные связи: в одной (с ASP-86) он выступает в качестве донора, во второй (с LEU-129) - в качестве акцептора. Соседний ASP-86 также участвует в 2х связях: одна с нашим GLU-147 (тут ASP акцептор, GLU - донор), а вторая - с GLY-93 в качестве акцептора. Тут все связи супер-хорошие
Здесь мне кажется, что PROPKA здесь угадала и с фактом протонирования, и местонахождением протона.
все вместе + вспомнила про лиганд
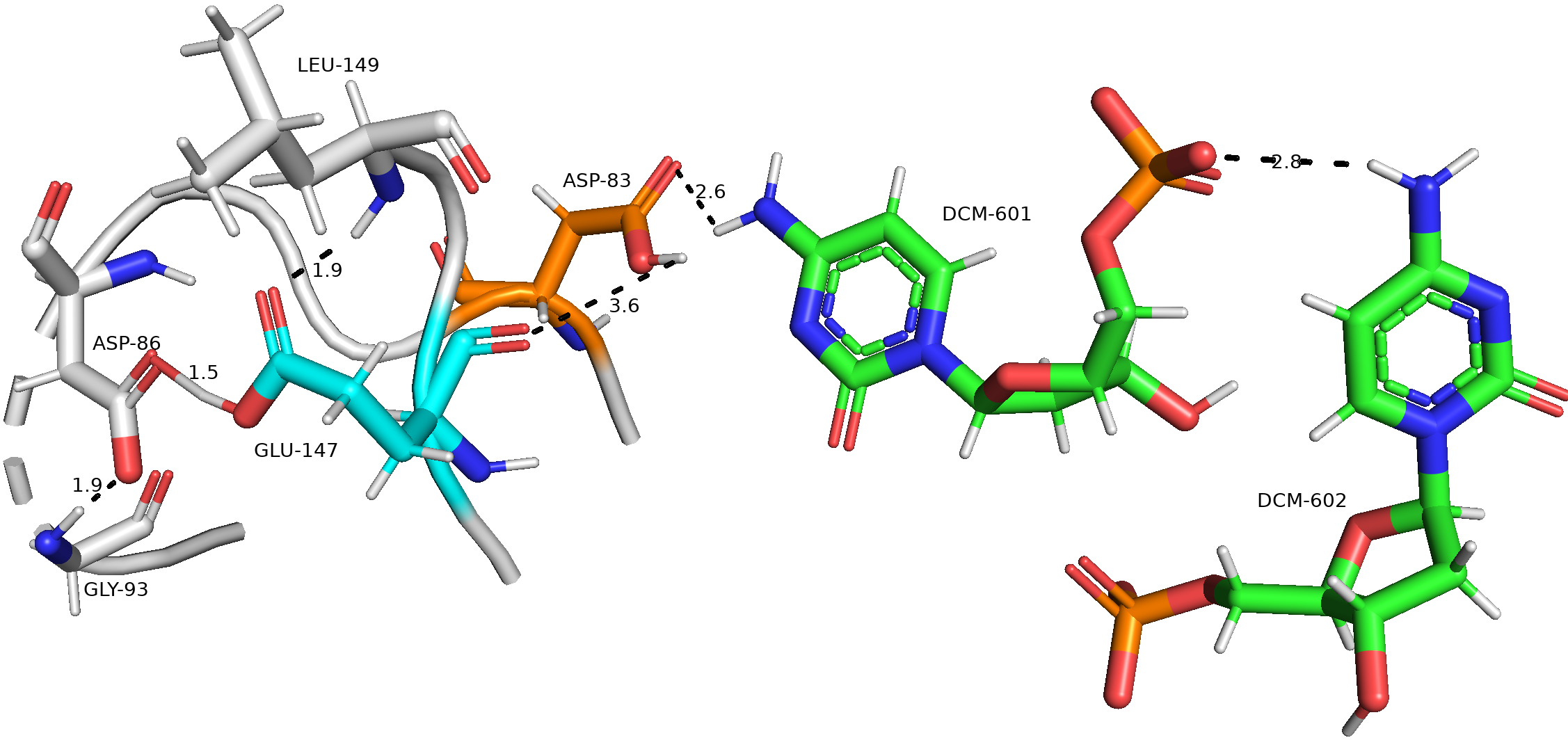
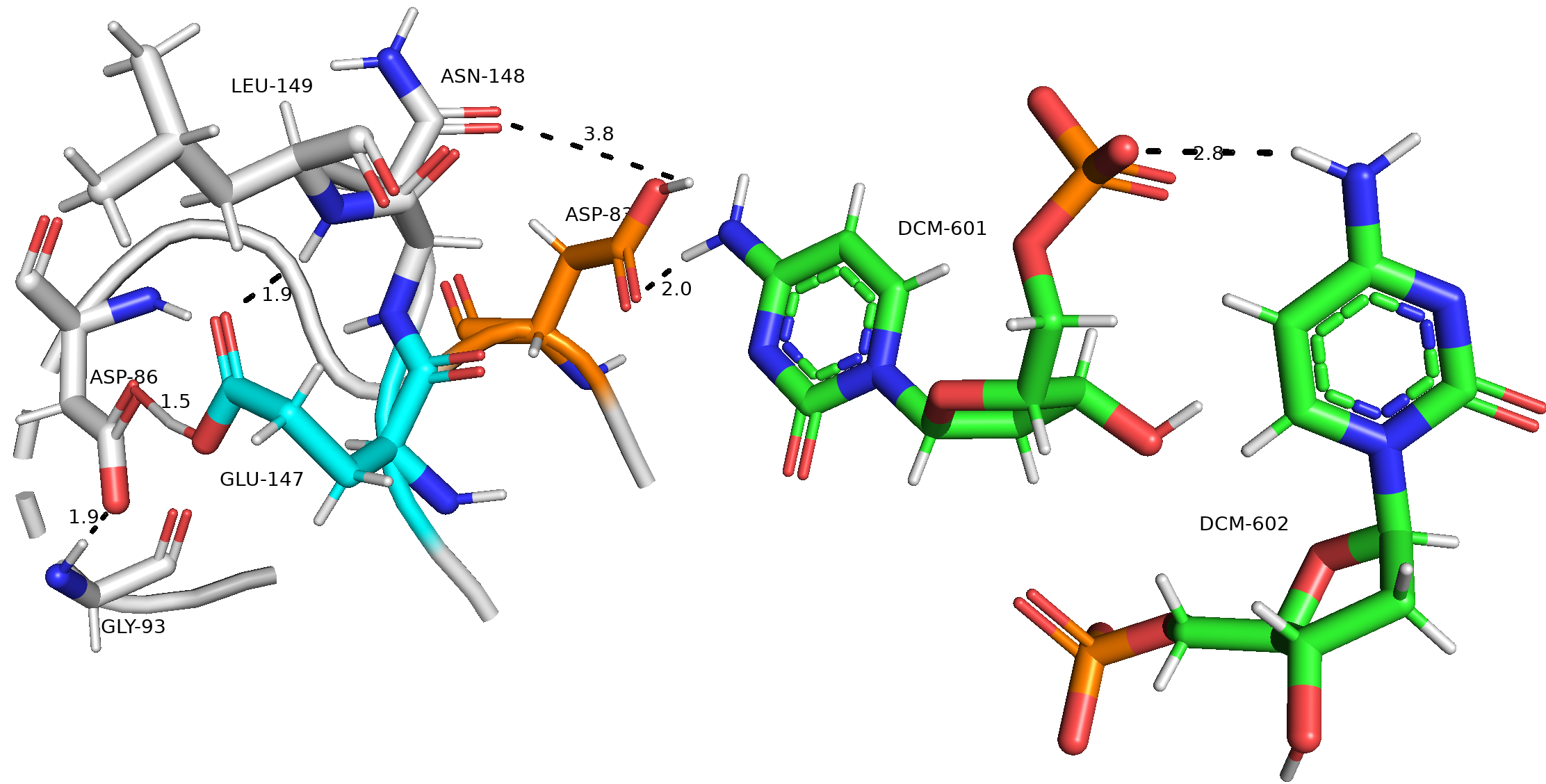

НО!!!!! оказалось, что я пропустила лиганд нашего белка - он в корне меняет дело. К тому же, ASP-38 и GLU-147 находятся очень близко друг к другу. Поэтому на картинках ниже я показываю оба остатка на одной картинке.
Опять же, в случае глутамата модель отлично справилась - протонирование верное и протон находится в нужном месте. В случае ASP, на мой взгляд, имеет место неопределенность. На рисунке 3A показаны связи при оригинальном предсказании модели, на риснуке 3B - при смене предсказанного H между кислородами ASP, а на рисунке 3C - при убранном протонировании. В случае протонирования ASP выступает в качестве акцептора водородной связи от NH2 лиганда, а также донором водородной связи с C=O группой различных остатков (на рисунке А - GLU-147, на рисунке B - ASN-148). Но донорные связи в обоих случаях значительно длиннее остальных связей. Также имеет место отсутствие протонирования: мы можем надеяться на наличие ионной связий между COO- аспартата и NH3+ лиганда. В целом ионные связи сильнее водородных. В общем, я думаю, что здесь однозначно установить корректную структуру нельзя.
PROTOSS
Наученная предыдущим опытом, я сразу покажу общую структуру для обоих остатков, предсказанную PROTOSS

PROTOSS подтверждает рассуждения о роли ASP выше. PROTOSS не протонирует ASP-83, то есть действительно имеет место предположение о резонансной структуре остатка (COO-) и наличие ионной связи с NH3+ лиганда. Интересно, что PROTOSS также не протонирует GLU-147, но мы видим всю тоже мощнейшую водородную связь с ASP-86, только в этот раз протонируется уже ASP-86. Здесь я лично особой разницы не вижу. GLU также продолжает быть акцептором водородной связи от NH LEU-149
ВЫВОД
Вывод обеих программ позволил проанализировать все возможные варианты протонирования и местонахождения протона на остатках ASP-86 и GLU-147 в структуре 5FBF. Важную роль здесь играл лиганд. Я думаю, что ASP-147 все-таки должен быть депротонирован и удерживать лиганд ионными связями. В случае GLU-147 важно протонирование одного из двух остатков: GLU-147 или ASP-86.