AlphaFold + DOMAK
Задание 1B Helices, последовательности B11
В задании 1 я выбрала подпункт B (Helices), в котором даны три последовательности. В первой (оригинальной последовтаельности, seqA) находится структура из альфа-спиралей; в seqB и seqC находится та же последовательностб, только с полиPro стрэтчем (разной длины) внутри одной из альфа-спиралей. Я ожидаю, что такие пролиновые вставки будут нарушать структуру спирали: спираль удерживает свою структуру за счет водородных связей NH i-го остатка и C=O i+4 остатка (в самой дефолтной спирали), для такой укладки должно быть возможно образование характерных φ и ѱ торсионных углов. Для пролина, вследствие его цикличности с участием N, при образовании пептидной связи N оказывается связан с 3мя атомами углерода, то есть не протонирован, а значт, не может быть донором водородной связи. Я посмотрела карты Рамачандрана и заономерно обнаружила, что в целом, для Pro допустимая (и особенно предпочитаемая) области заметно меньше, чем для остальных остатков, поэтому ему сложно встраиваться в альфа-спирали.
Я выбрала последовательности B11. Оригинал нашла в PDB: 1Y9B, длина последовательности 90 АК. В seqB остатки 66-69 заменены на Pro, в seqC - остатки 59-69.
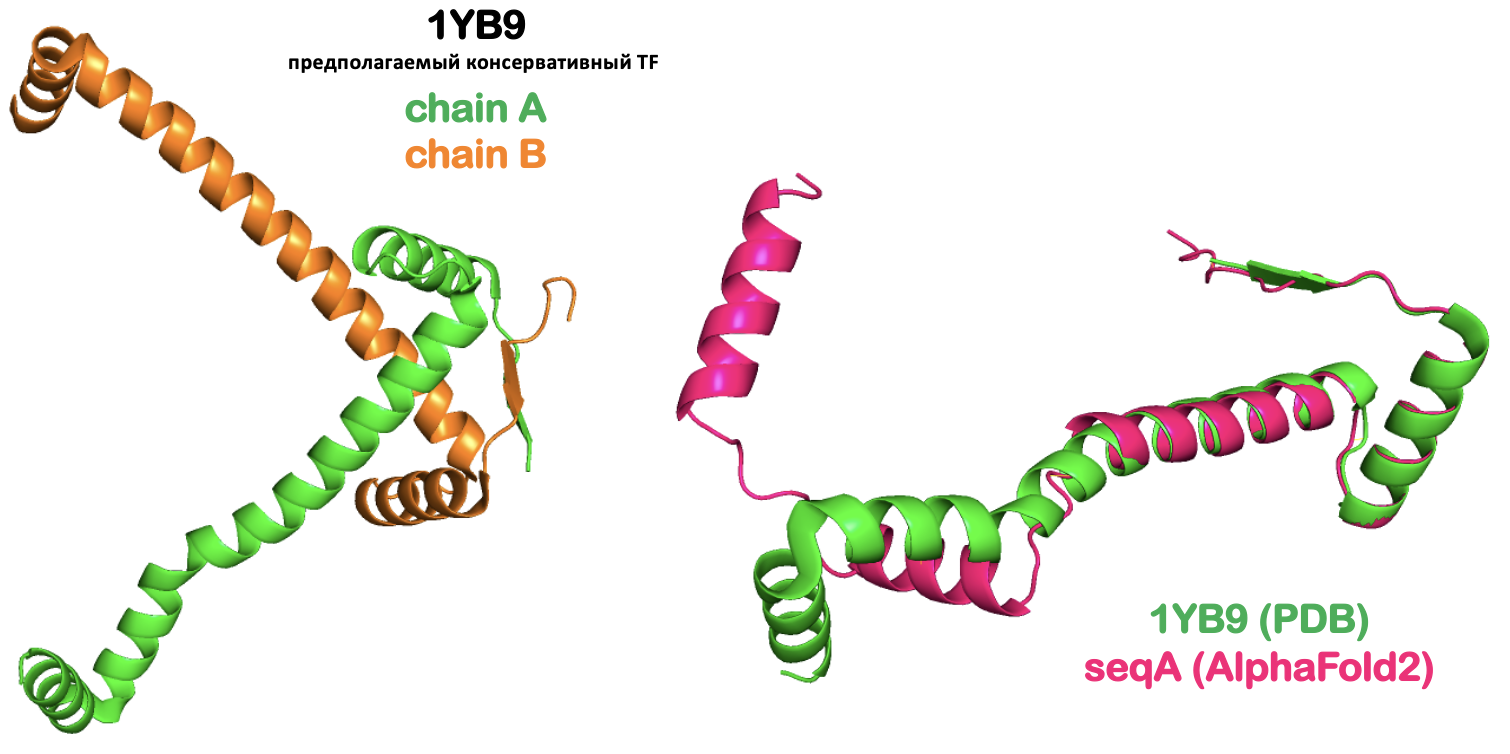
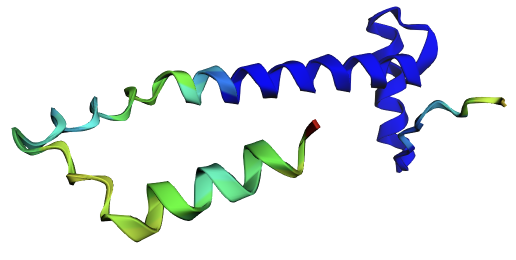
В PDB структура описана как "предполагаемый консревативный домен транскрипционного фактора, выделенного из холерного вибриона". На рисунке 1 мы видим, что предсказание AF2 в целом неудачное, выровнялась только часть структуры с plDDT > 0.9 (рис 3A). Почему-то AF2 не захотел предсказывать единую длинную спираль и разбил ее на две, вставив между кусками disorder region. Из-за этого вторая часть спирали развернулась, и мы видим, что левая часть совсем не выровнена.
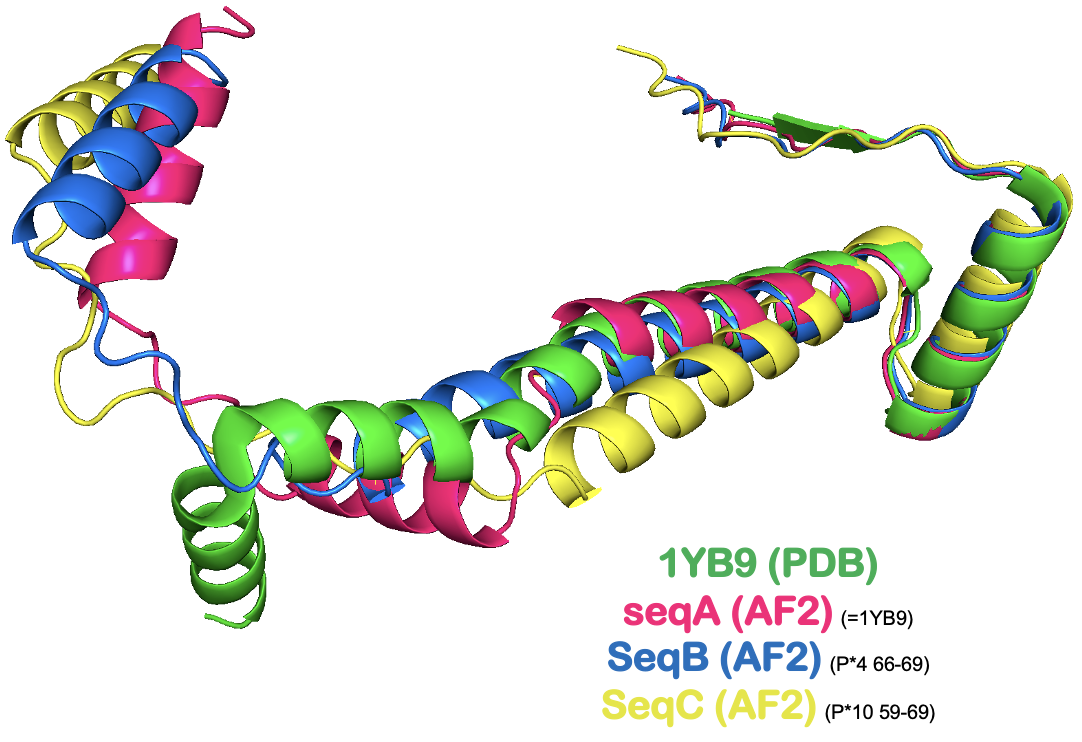
На рисунке 2 показано выравниваие всех предсказанных структур с структурой из PDB. интересно, что для seqB (синяя) AF2 предсказывает более длинную спираль посередине, чем для SeqA. В принципе для самой правой (N-концевой) альфа-спирали выровнено хорошо, на риснуке 3 эта область помечена как high confidence (plDDT > 0.9) для всех последовательностей. Интересно, что мы видим, что disorder region seqC длиннее, чем у seqB (в центре), так как именно здесь находится полипролиновая замена, но тем не менее в SeqA эта область тоже есть, просто потом собирается во вторую часьт спирали.
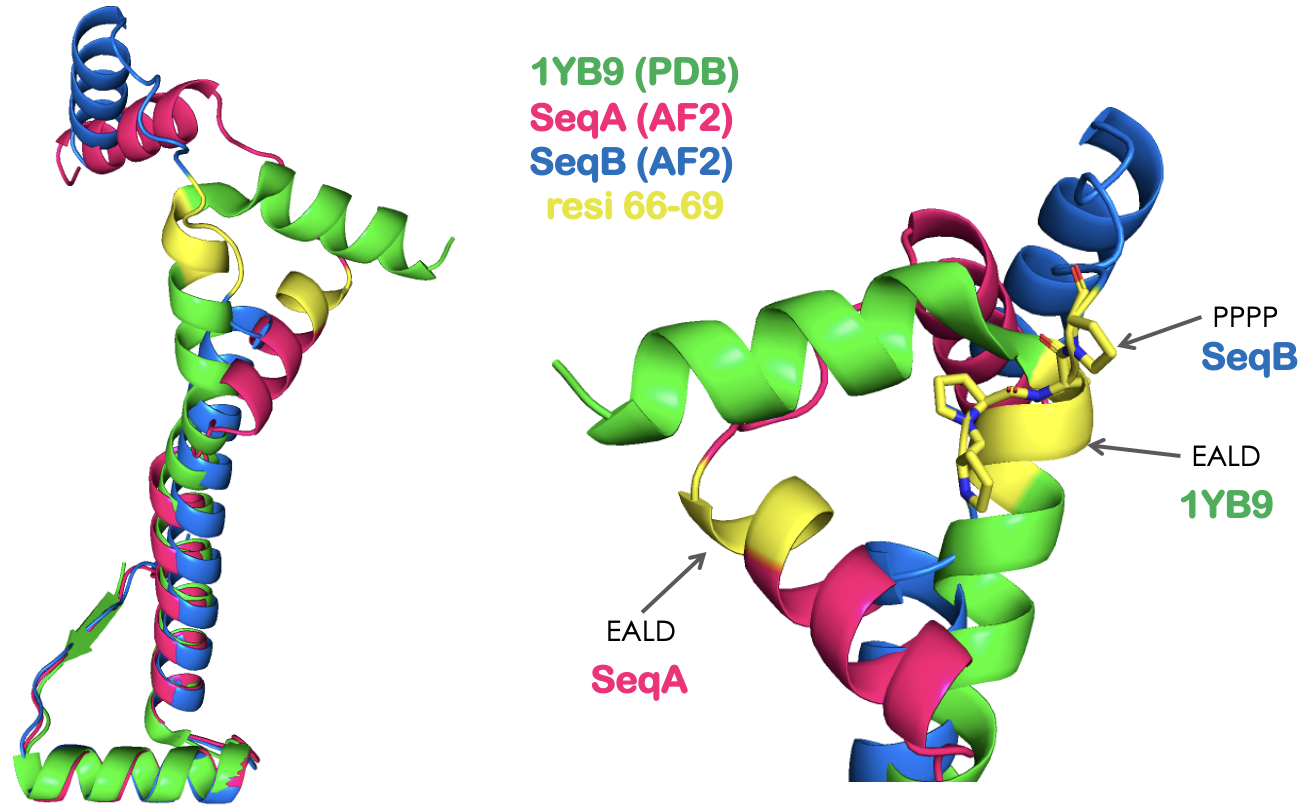

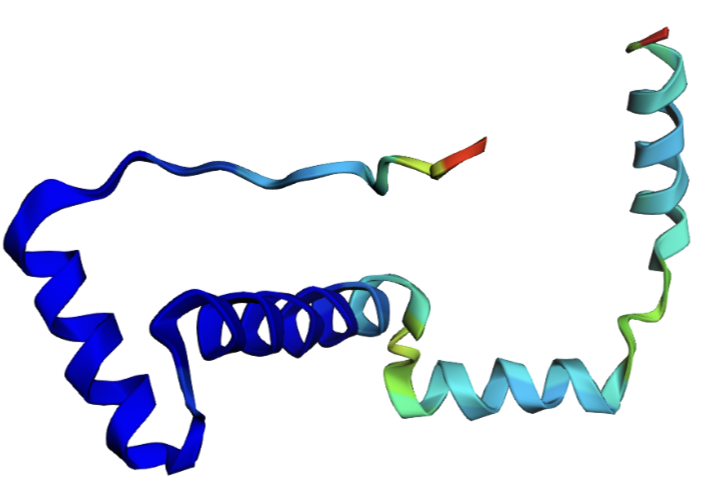
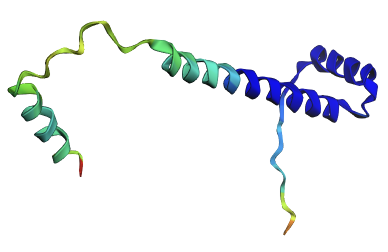
На рисунках 4-5 наблюдаем за влиянием замены C-конца альфа-спирали на пролины. Как и ожидалось, видим, что в оригинальной структуре и SeqA на месье этих остатков (66-69) находится C-конец альфа-спирали, а в SeqB, где resi 66-69 == PPPP, спираль не собралась, вместо нее disorder region. Ожидаемое поведение

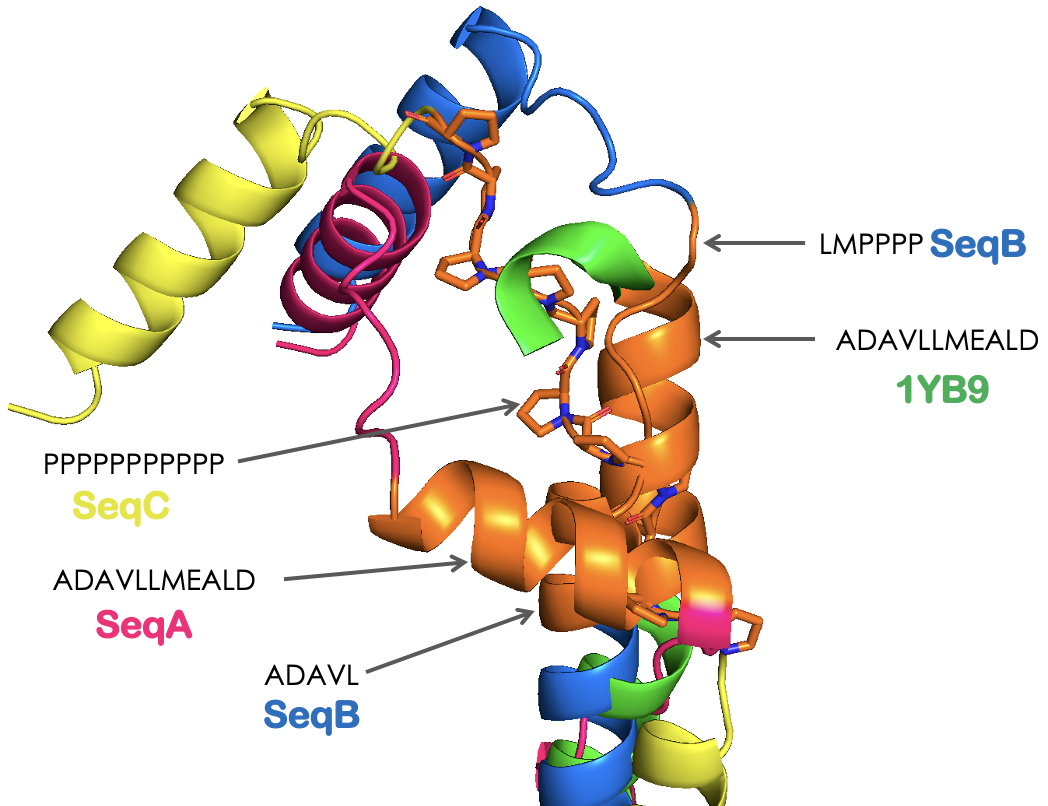
На рисунках 6-7 показаны структуры, на которых выделены остатки 59-69, в SeqC на их месте стоят Pro. Видим, что в PDB и предсказании SeqA эти остатки образуют часть альфа спирали. В SeqB на остатках 65-69 находится disorder region (не удивительно, 66-69 - пролины), а незамененная часть тоже собралась в кусок альфа-спирали. В SeqC на месте всех этих остатков находится disorder region. Ожидаемо
общий вывод
В целом AF2 работает с вставкой пролинов в альфа-спираль ровно так, как я ожидала - так как пролин не может образовывть H-связи для удержания остова спирали, то в месте вставки AF2 предсказывает disorder region. Больше вопросов к предсказанию самой структуры последовательности, мы видим непонятно вставку disorder region посреди большой альфа-спирали. Эта область находится на остатках 52-58, последовательность Gln-Ala-Leu-Cys-Leu-Ser. Пролинов тут нет, хз почему не свернулось в спираль.
Задания 2-3: DOMAK
Ссылка на колаб тут . У меня был белок 3FRO, chain A . В колабе я запустила подсчет для деления белка на 2 части: домен А заканчивается на остатке i, домен B начинается с i+1 остатка (я так поняла, что задание было таким). У меня получился вот такой вот график:
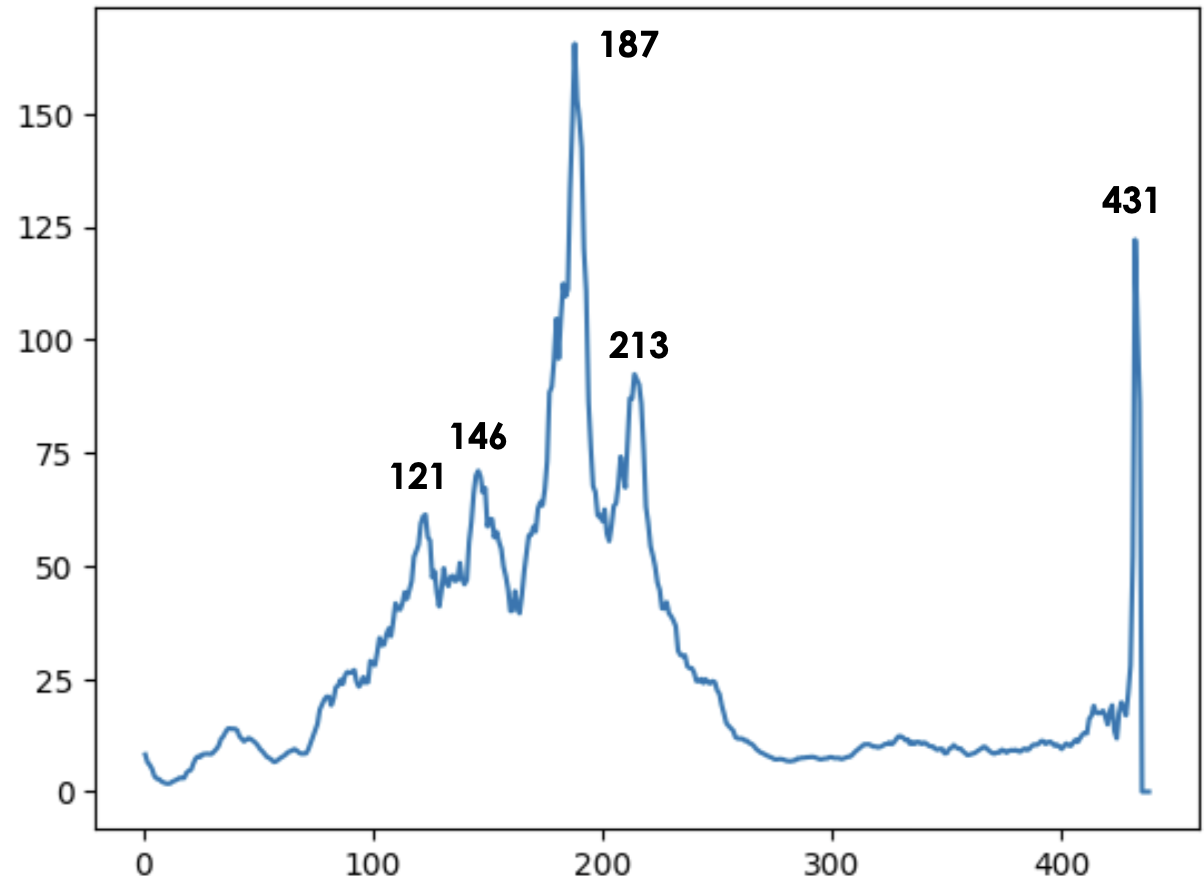
Также выведем топ позиций с максимальным предсказанным значением: 187, 188, 189, 186, 190, 185, 431, 191, 182, 184, 192, 183, 181, 179
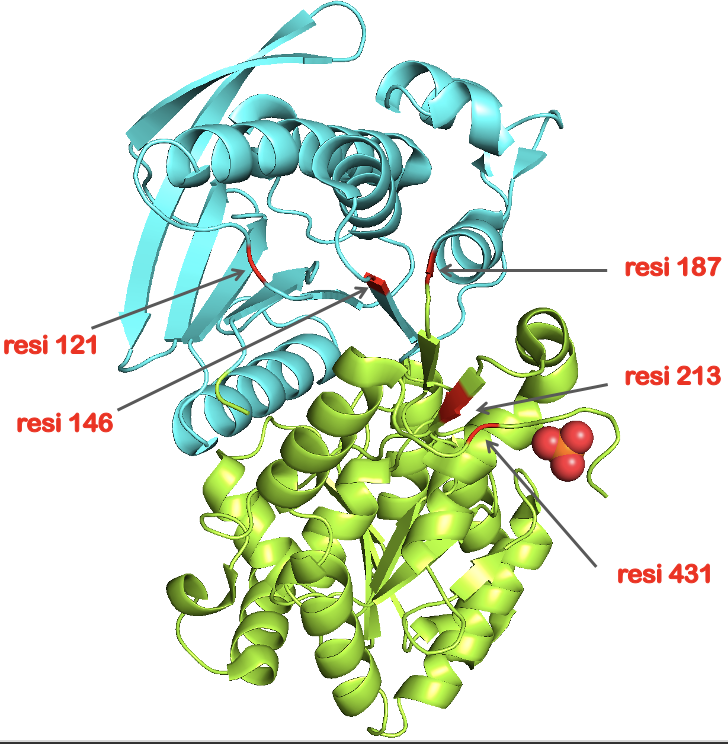
. В общем-то это просто расположение самого высоко пика на позициях 179-192, также здесь выделена позиция 431. Позиция с самым высоким split_value #187. Поищем ее в белке.
Сравним с бд структурных доменов - SCOP и CATH.
CATH
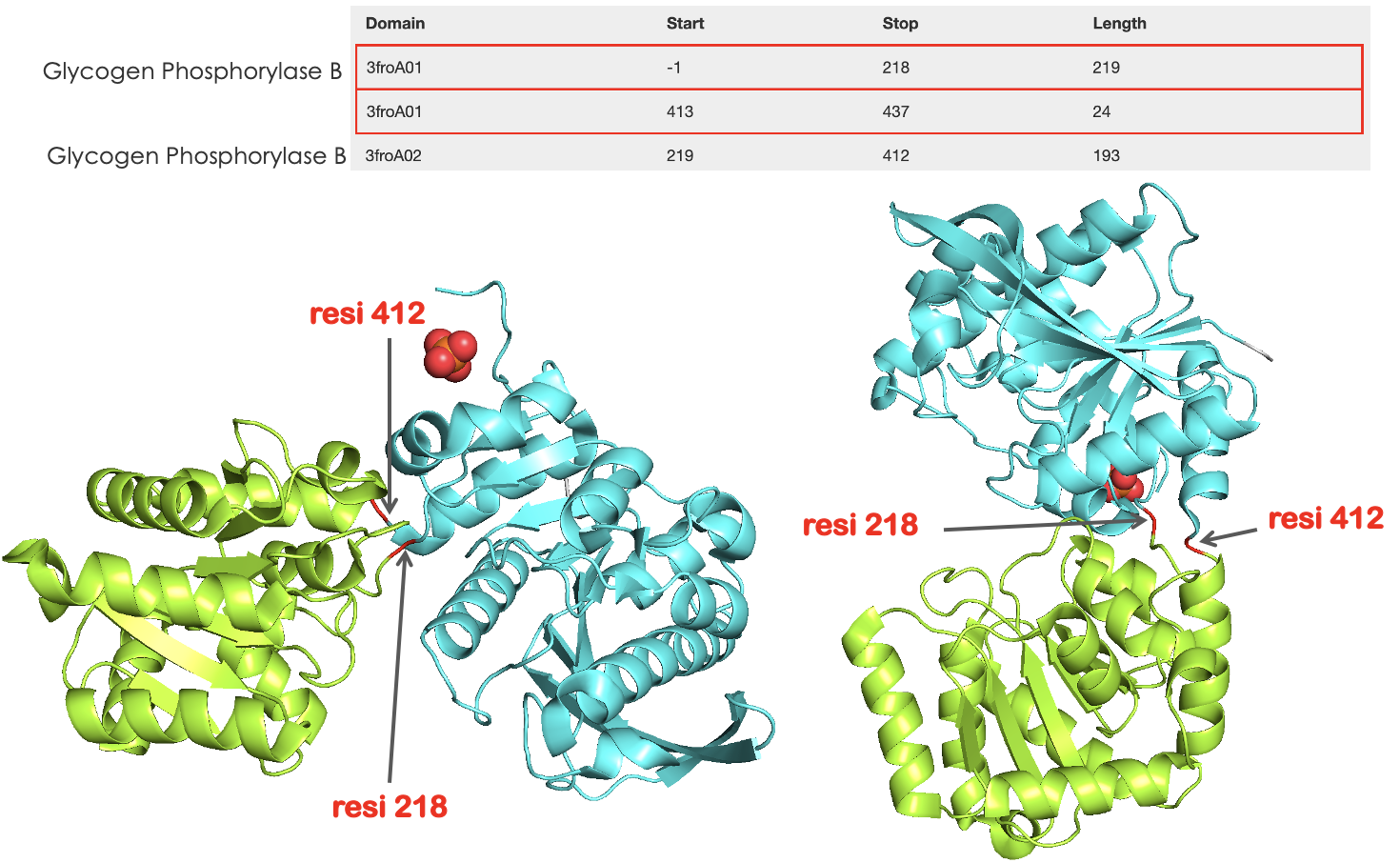
Видим, что визуально похоже, НО DOMAK относит центральные петли (сравните левый рисунок с Рис 11 с левым рисунком Рис 9), петли между доменами относятся к разным участкам. При чем DOMAK определил пик на 213 (CATH предсказывает 218, оба остатка из disorder region, так что вполне похоже), но DOMAK не предсказывает важный пик для второй спорной петли в позиции 412.
SCOP
SCOP предсказывает единственный структурный домен из суперсемейства Type B Glycogentransferase-like, домен GlgA glycogen synthase. Он находится в позициях 220-415, то есть он выделяет часть домена 3froA02 из CATH.

В общем, предсказание DOMAK получается похожим, но так мы считаем разделением между доменами единственный остаток, то в прицнипе хорошо получилось. Если бы по двум-трем остаткам предсказывали, возможно бы получилось больше похоже на CATH, но по-моему и так неплохо вышло.