Практикум 8
Нуклеотидный BLAST
Задание 1. Разные варианты BLAST для фрагмента ДНК
Участок [10434389..10448540] (14152 п.н.) 1-ой хромосомы Большой панды был взят из нуклеотидной записи NC_048218.1 . На этом фрагменте ДНК расположен ген SLC5A3 (им кодируется котранспортер натрия/мио-инозитола).
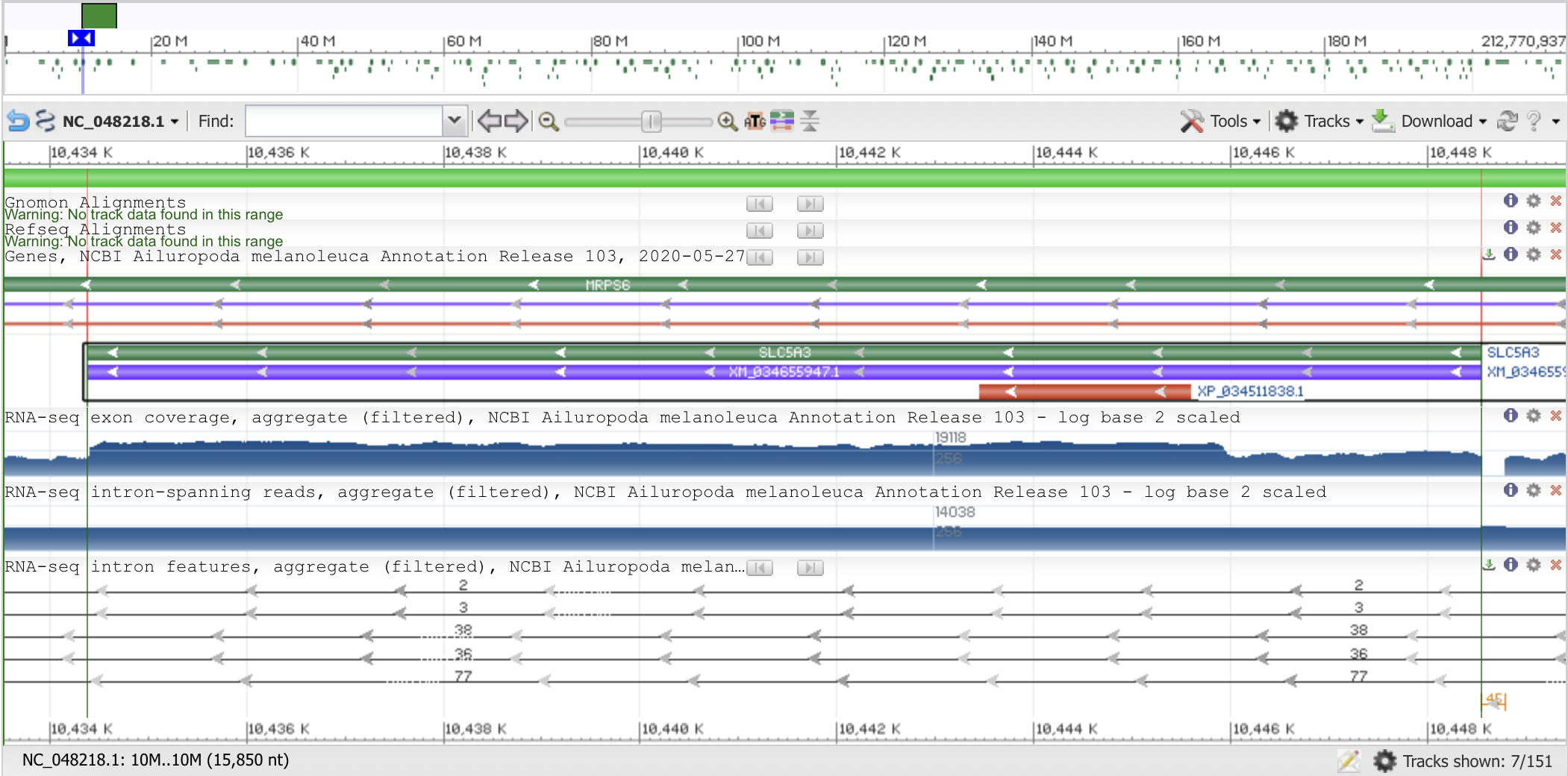
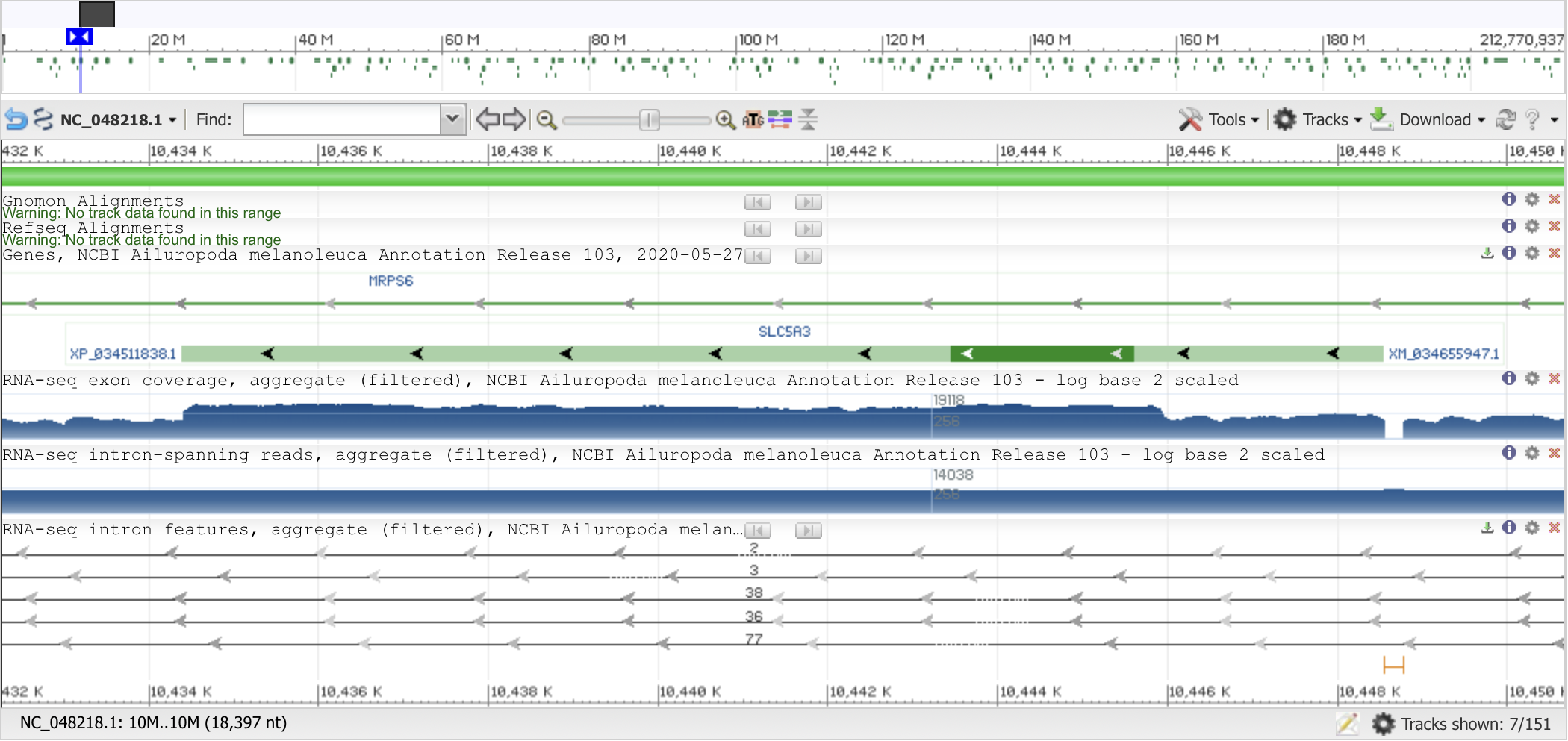
Поиск BLAST по эукариотическим последовательностям
Поиск BLAST я решила осуществить по классу Птиц (Aves), так как выбранный мной организм - млекопитающее
blastn выдал 100 находок (длина слова 11), средний процент покрытия 17%, 97 из них являются предсказанными мРНК для SLC5A3. Он используется для сравнения геномов не близкородственных организмов или поиска гомологичных последовательностей.
megablast выдал 100 находок (длина слова 28), средний процент покрытия 15%, все являются предсказанными мРНК для SLC5A3, так как он используется для поиска почти идентичных последовательностей близкородственных видов.
blastx выдал тоже 100 находок (длина слова 5), средний процент покрытия 15%, среди находок есть ген SLC5A3, SC5A3 protein и sodium/myo-inositol cotransporter. Он используется для поиска гомологичных белков, когда у нас есть только последовательность гена.
tblastx не выдал ничего (длина слова 3), даже после изменения всех параметров он всё равно выдал ошибку, потому что с помощью него мы ищем гомологи белка в последовательностях, в которых белки ещё не были предсказаны.
Задание 2. Поиск в геноме эукариота генов основных рибосомальных РНК по далекому гомологу
В состав малой субъединицы прокариотичсекой рибосомы входит 16S рРНК, в состав большой - 23S рРНК. Для выполнения задания был выбран blastn, так как мы ищем ген не белка,а рРНК и используем нуклеотидные последовательности не близкородственных организмов.
Для работы локального BLAST, была проиндексированна последовательность генома Ailuropoda Melanoleuca
makeblastdb -in GCF_002007445.2_ASM200744v3_genomic.fna -dbtype nucl
Предоставленный нам файл с последовательностями рРНК Eschericgia coli разбит на 2 файла: 16S.fasta и 23S.fasta. Далее проводился поиск по базе данных, созданной при индексации генома:
blastn -task blastn -evalue 10 -query 16S.fasta -db GCF_002007445.2_ASM200744v3_genomic.fna -out blast_16S.txt -outfmt 7
blastn -task blastn -evalue 10 -query 23S.fasta -db GCF_002007445.2_ASM200744v3_genomic.fna -out blast_23S.txt -outfmt 7
Bзначально был выставлен evalue 0.05, чтобы исключить случайные находки, но такой поиск ничего не дал, поэтому этот параметр был увеличен до 10. С помощью аргумента -outfmt данные были выведены в таблице
Для 16S количество находок 20 , в основном это ncRNA (которые принадлежат некодирующей части генома) и mRNA, но есть так же и рРНК (NC_009492.1,ген KEG54, эта находка попала в митохондриальный геном, она значимая, можно назвать гомологией, так как кодируемая митохондриально 16S РНК представляет из себя митохондриальную большую субъединицу рРНК { эту информацию можно псомотреть тут }) и 2 находки соответствовали неразрешенному геномному скаффолду - unplaced-scaffold45837, unplaced-scaffold59032 (NW_023218100.1, NW_023232762.1). Файл с находками.
Для 23S количество находок 8 - почти все мРНК и ncRNA, одна находка unplaced-scaffold10565 (NW_023178910.1) Файл с находками.