Практикум 6
Трансмембранные белки
Сравнение предсказаний трансмембранных участков в бета-листовом белке
Для выполнения этого задания я нашла в базе данных OPM трансмембранный белок, содержащий бетта-бочки, Outer membrane receptor PiuA (5fok) - рецептор внешней мембраны(PDB_ID: 5FOK, UniProt: PIUA_PSEAE ). Он был выделен из Pseudomonas aeruginosa и влияет на её устойчивость к проникновению антимикробных молекул. С использованием этого рецептора фармакологами проводятся исследования для поиска лекарственных препаратов против этих грамм-отрицательных бактерий
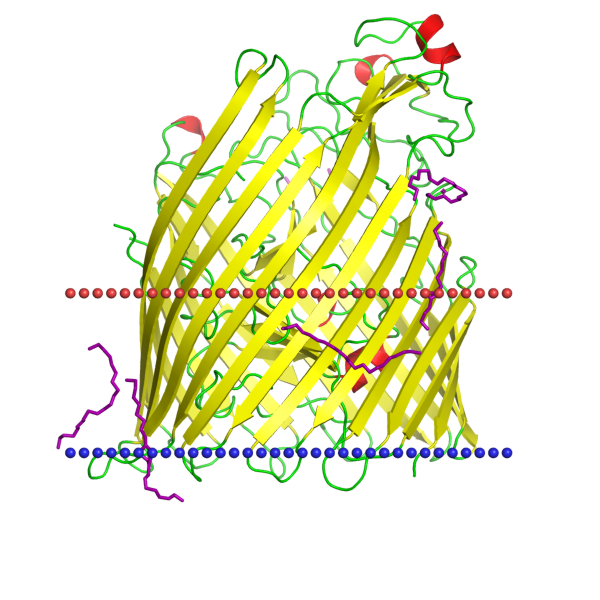
| Толщинa гидрофобной части белка в мембране | 23.4 Å |
| Координаты трансмембранных участков | 1( 158- 163), 2( 170- 178), 3( 186- 192), 4( 209- 218), 5( 222- 230), 6( 279- 286), 7( 294- 300), 8( 339- 348), 9( 355- 363),10( 426- 434),11( 441- 446),12( 478- 487),13( 494- 499),14( 530- 539),15( 544- 551),16( 579- 585),17( 594- 599),18( 626- 632),19( 640- 645),20( 666- 672),21( 680- 684),22( 710- 718) |
| Среднее количество остатков в одном β-тяже белка | 7 |
| Мембрана, в которой находится белок | Внешняя |
С сайта PDB была скачана последовательность в формате FASTA, её я подала на вход программе DeepTMHMM.
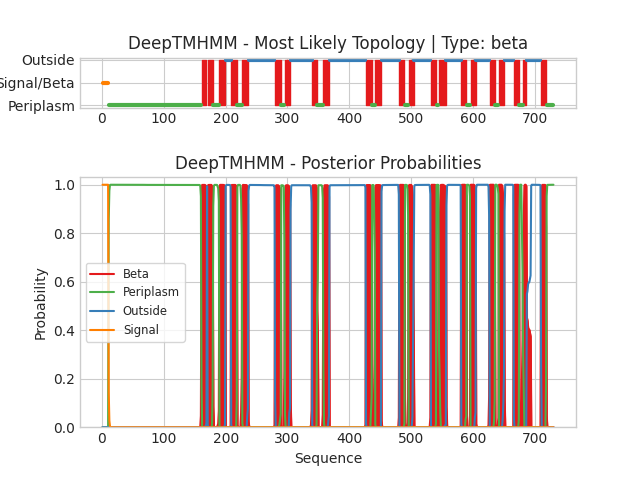
Текстовая выдача программы. Координаты трансмембранных участков белка, предсказанные DeepTMHMM: 1(161-169), 2(171-179), 3(190-199), 4(209-218), 5(227-236), 6(280-289), 7(296-304), 8(340-347), 9(358-367), 10(427-436), 11(442-451), 12(481-489), 13(496-504), 14(532-540), 15(545-556), 16(581-589), 17(596-604), 18(627-635), 19(642-650), 20(666-674), 21(681-686), 22(710-718). Предсказание DeepTMHMM получилось плохим, только два трансмембранных участка полностью совпадают (209-218) и (710-718).
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Мне был выдан белок Lipid III flippase (Флиппаза липида III) с идентефикатором UniProt: WZXE_ECOLI. Он опосредует трансслойное перемещение липида III от внутреннего к внешнему листку цитоплазматической мембраны во время сборки общего антигена энтеробактерий (ECA). Необходим для сборки фосфоглицерид-связанной формы ECA (ECA(PG)) и водорастворимой циклической формы ECA (ECA(CYC)).
Возьмём последовательность из UniProt и запустим программу DeepTMHMM. Текстовая выдача программы.
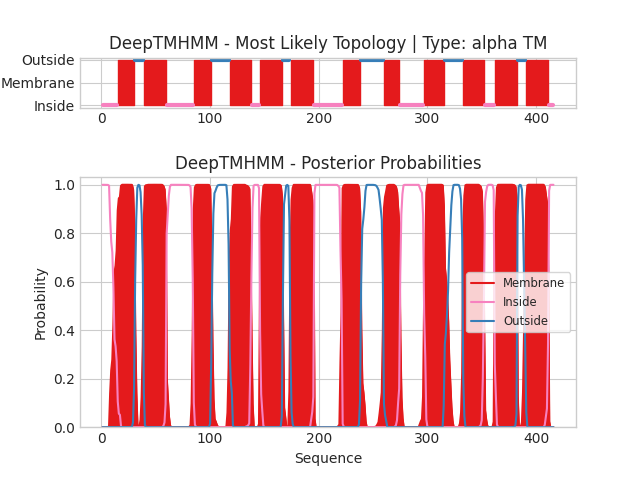
Координаты трансмембранных участков белка, предсказанные DeepTMHMM: 1(15-30), 2(39-59), 3(85-101), 4(118-138), 5(146-166), 6(174-195), 7(222-238), 8(260-274), 9(297-315), 10(333-352), 11(362-382), 12(391-411)
Потом также на сайте UniProt скачала предсказание AlphaFold трехмерной структуры выданного мне белка. Теперь предскажем топологию белка с помощью PPM и подадим ему на вход структуру предсказанную AlphaFold. Параметры: мембрана грам-отрицательных бактерий, топология N-конца - внутри клетки (по выдаче DeepTMHMM, в литературе ничего не указано). Топология, предсказанная PPM
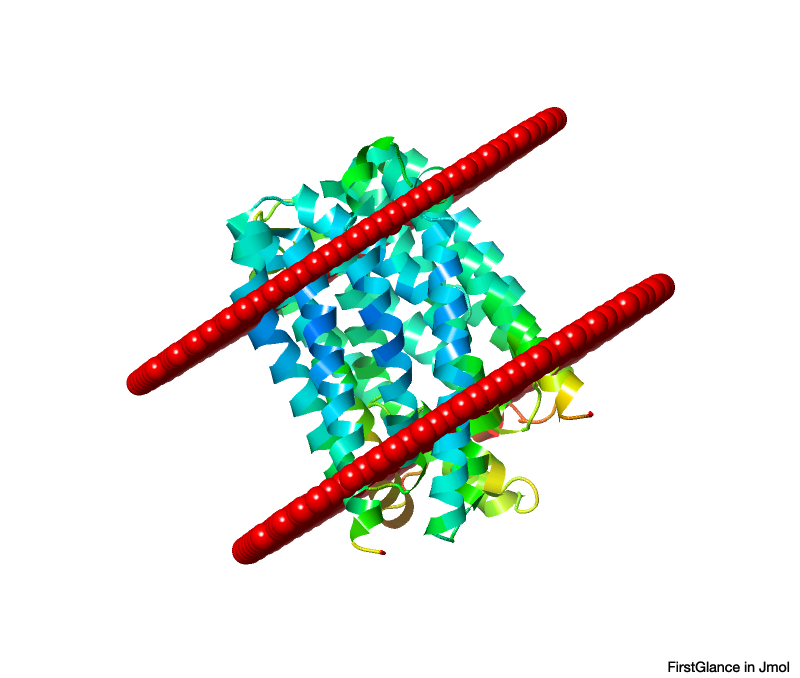
Координаты трансмембранных участков белка, предсказанные PPM: 1( 7- 25), 2( 41- 55), 3( 82- 101), 4( 119- 140), 5( 146- 163), 6( 177- 196), 7( 217- 239), 8( 254- 273), 9( 296- 315),10( 333- 352), 11( 360- 378), 12( 390- 408)
Предсказания DeepTMHMM и PPM вполне похожи, все трансмембранные участки примерно одинаковые и большей своей частью перекрываются.