Практикум 7
Базы данных Reactome и STRING
Выданный мне список белков я решила проанализировать с помощью баз Reactome и STRING, потому что они мне понравились
Reactome
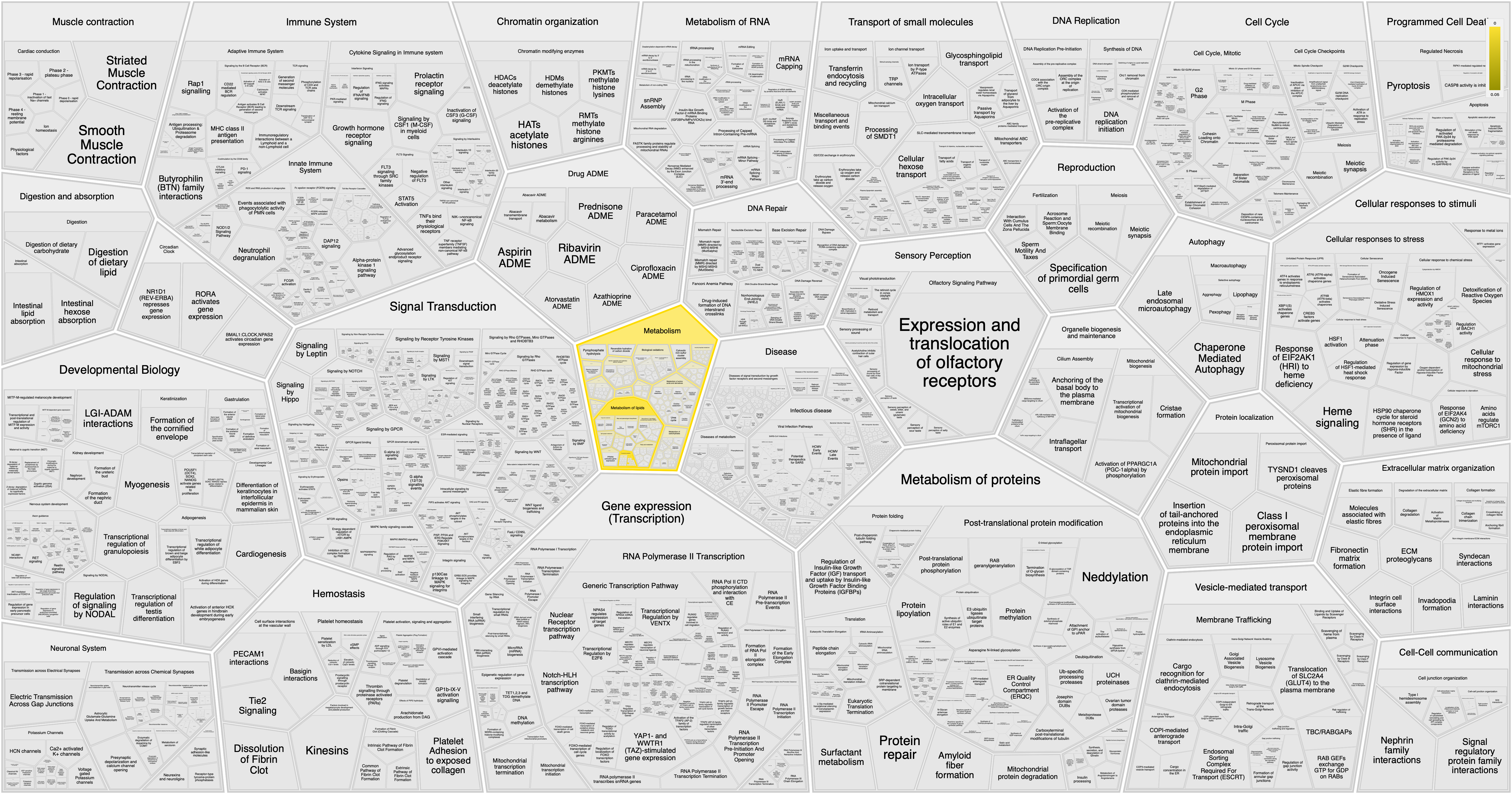
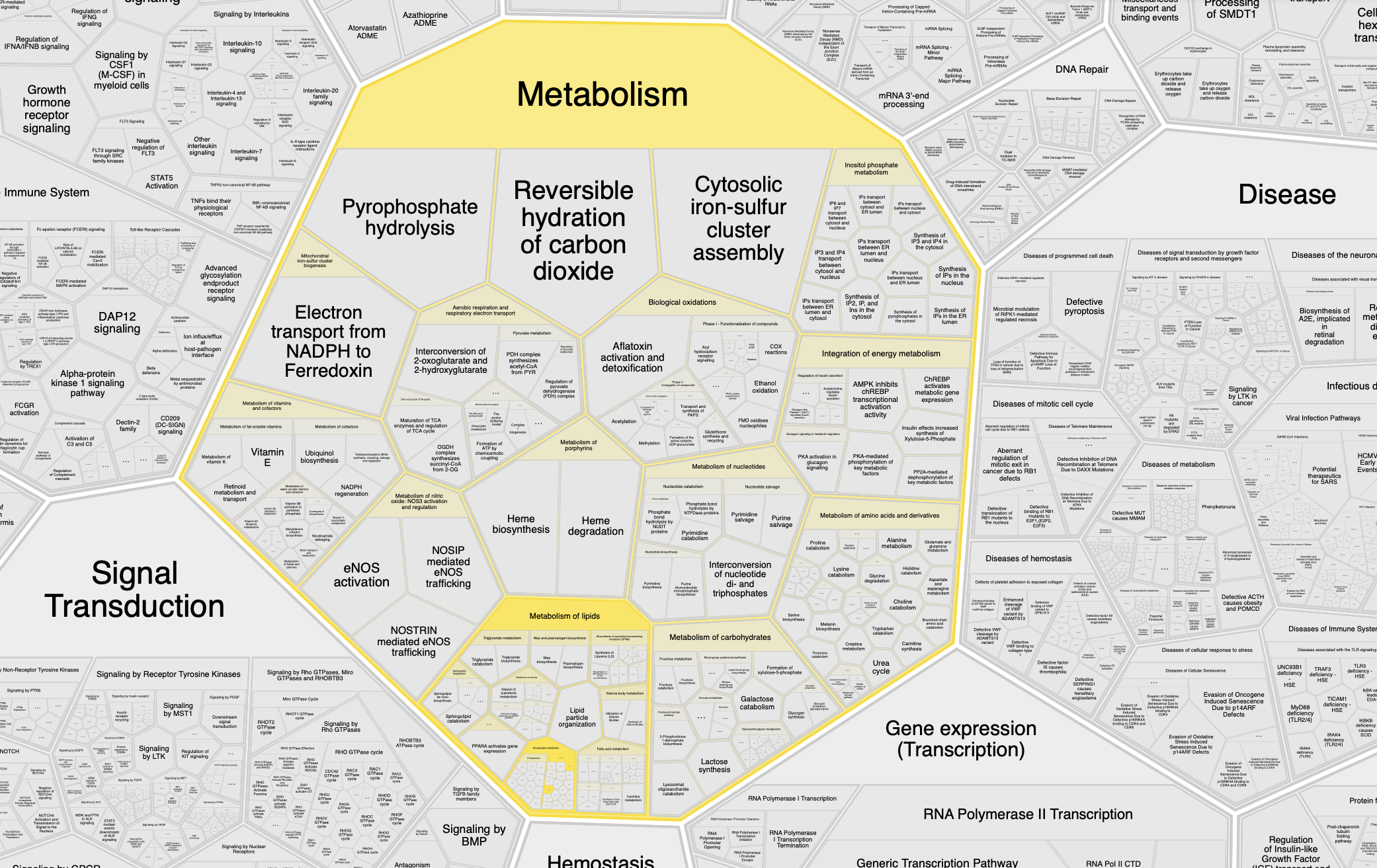
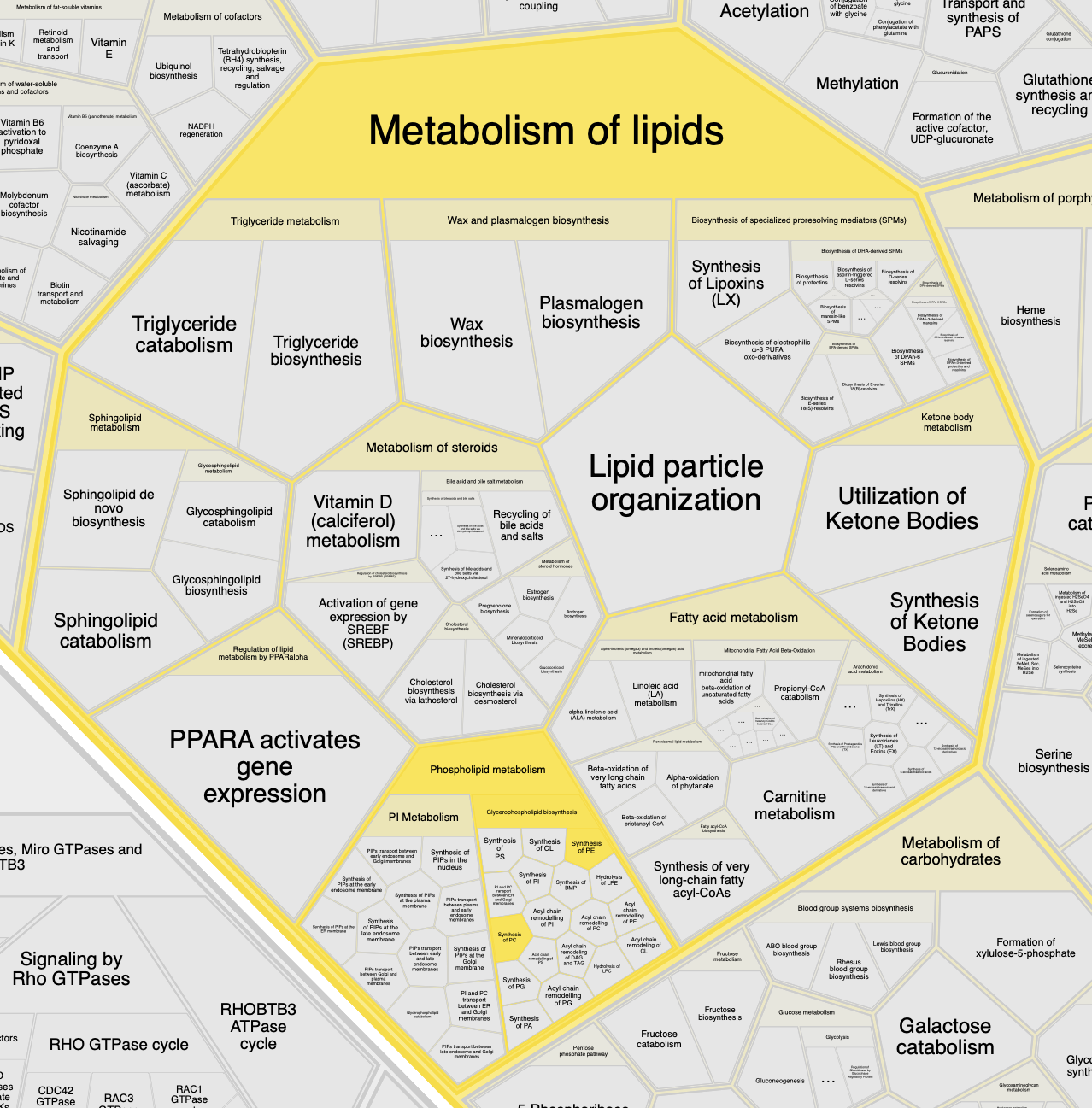
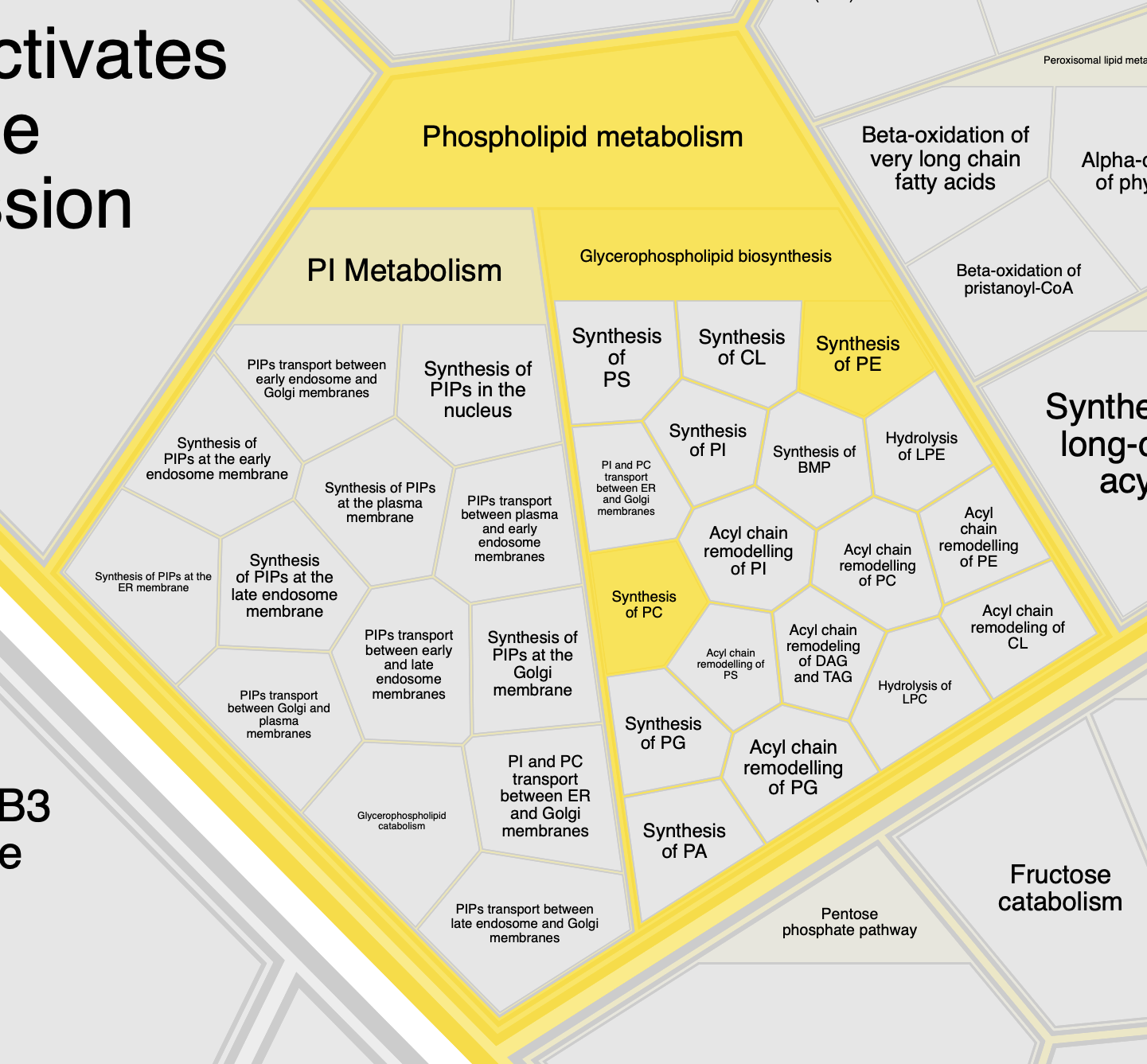
Всего в выданном мне списке 6 белков, если обогащение терминами в выдаче Reactome записать схематически, включая в схему только крупные группы, то получится:
Метаболизм 6
Метаболизм липидов 6
Метаболизм фосфолипидов 6
Биосинтез глицерофосфолипидов 6
Синтез фосфатидилхолина 4
Синтез фосфатидилэтаноламина 3
STRING
Теперь поработаем в базе данных STRING и произведем поиск по выданному списку белков. В качестве таксона выберем Homo sapiens.
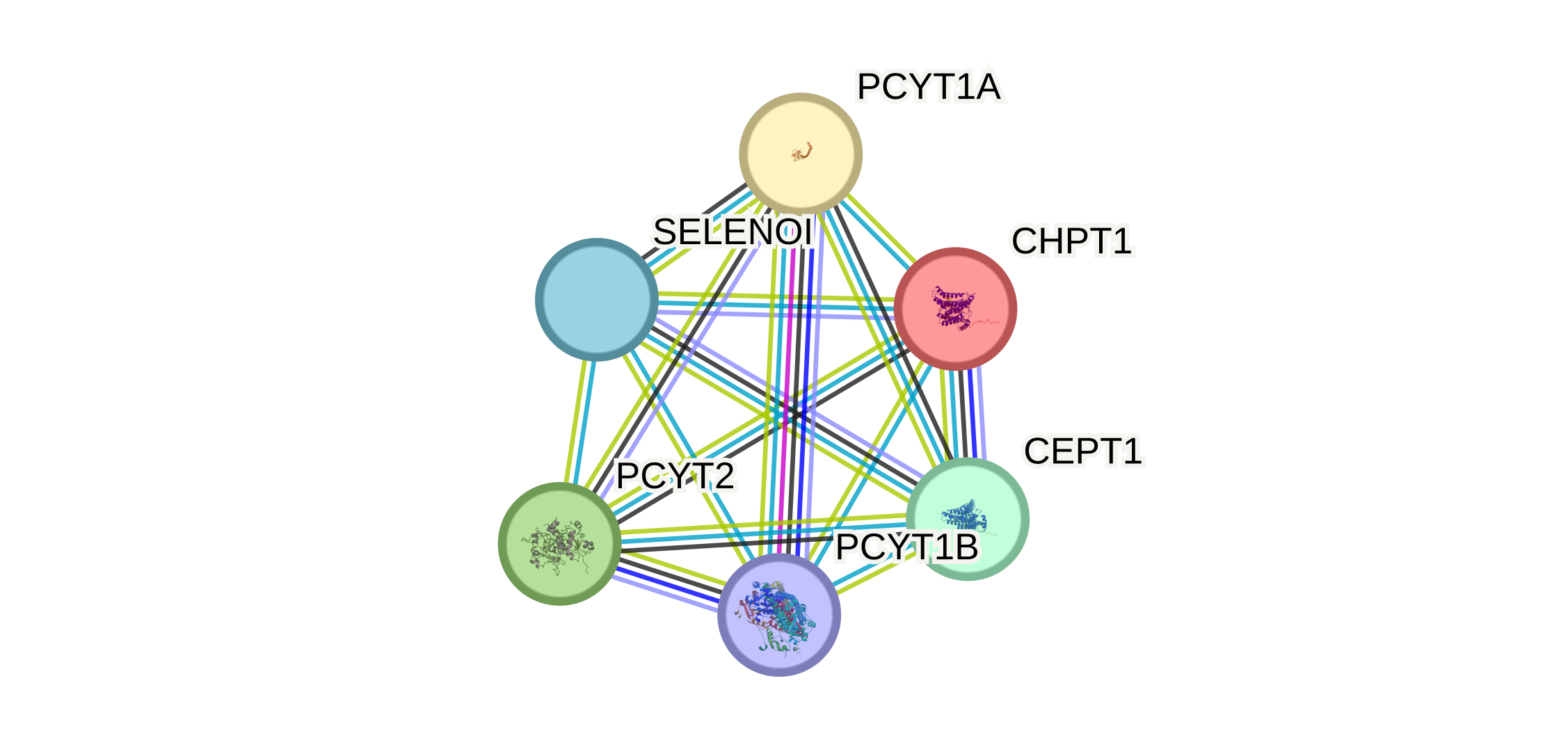
Получился граф с большим количеством ребер, PPI enrichment p-value < 1.0e-16, а значит, белки имеют больше взаимодействие между собой, чем ожидалось для случайного набора белков.
Также я решила покрасить узлы графа, в зависимости от того, в каком биологическом процессе они участвуют.
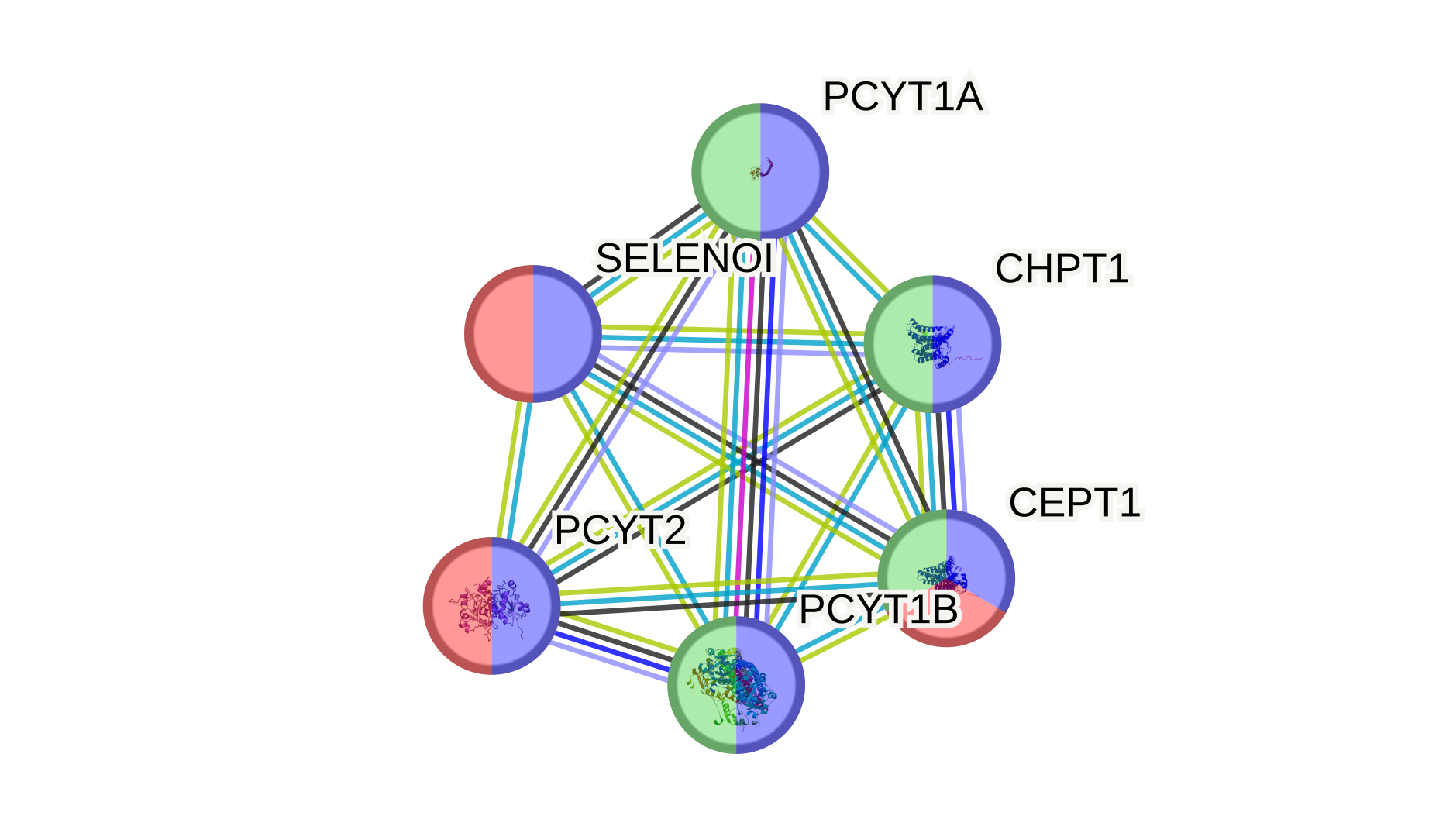
Получившиеся результаты соотносятся с теми, которые были получены в базе данных Reactome
Вывод: в выданном мне списке находятся в основном белки, которые участвуют в метаболизме липидов
