Практикум 9
Поиск de novo сигналов в ДНК
Подготовка данных
Для выполнения заданий данного практикума я решила выбрать геном бактерии Staphylococcus aureus. Был использован референсный геном GCF_000418345.1. Для получения файла со списком оперонов я использовала сервис Operon-mapper и файлы с последовательностью и аннотацией.
Далее необходимо составить список координат промоторов. Было предложено считать промотором область в 100 нуклеотидов перед началом оперона. Список оперонов и геном бактерии подавались на вход скрипту, который я позаимствовала у Георгия Муравьёва, были сгенерированы 3 файла: файл, на котором впоследствии будет обучен MEME, файл тестирования и негативного контроля.
Запуск MEME
Далее на kodomo запустиоа программу MEME, используя команду:
meme housekeeping.fasta -dna -nmotifs 3 -minw 6
На выходе получила три LOGO и HTML-отчёт, текстовую выдачу можно посмотреть тут
В результате работы программы было найдено 3 мотива. 2 из них имеют хороший E-value и похожи на SD (Рис.1 и Рис.2)


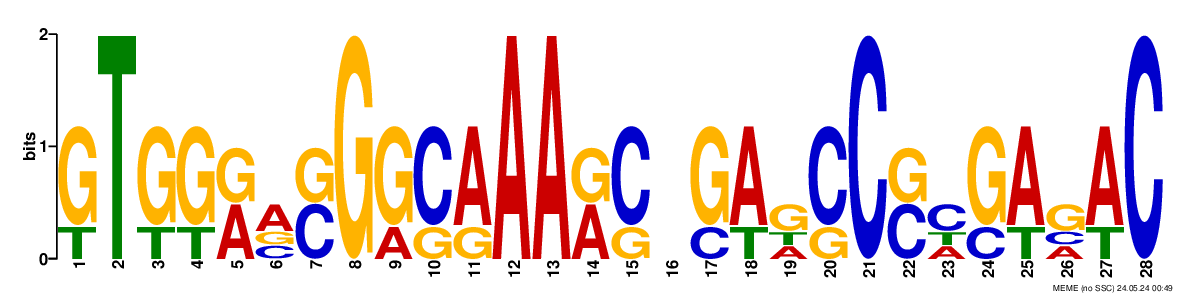
Для дальнейшей работы я решила использовать первую находку (у неё самый лучший E-value)
Поиск сигнала в материале для тестирования с помощью FIMO
Для того, чтобы найти выбранный мотив в положительном и отрицательном контроле, использовала следующие команды:
fimo --norc -motif WWWWWTTCCTCC -thresh 0.001 meme.txt promotors.fasta
fimo --norc -motif WWWWWTTCCTCC -thresh 0.001 meme.txt negative.fasta
В результате работы программы мы можем видеть, что реди всех промоторов получилось 969 находок, среди негативного контроля - 18 находок