На главную страницу четвёртого семестра
Описание базы данных SMART (Simple modular architecture research tool)
Состав базы данных, авторы и организация, предоставляющая данный сервис
SMART (Simple Modular Architectre Research Tool — простое средство поиска
модельной архитектуры) позволяет идентифицировать и аннотировать генетически
мобильные домены и анализировать доменную архитектуру. Обнаружено более 500
доменов сигнальных, внеклеточных и связанных с хроматином белков. Эти домены
всё больше аннотируются с учётом распределения в различных таксонах,
функциональных классах, с учётом третичных структур и функционально важных
остатков. Кажды домен, найденный в неизбыточной базе данных, а также параметры
поиска и таксономическая информация хранятся в соответствующей системе баз
данных. Пользовательские настройки в этой базе данных позволяют проводить поиск
белков, содержащих особые комбинации доменов, в определённых таксонах.
Базу данных SMART можно использовать в двух различных режимах:
нормальном или геномном. Главное различие между ними — это основная используемая база
данных В режиме Normal SMART база данных состоит из Swiss-Prot, SP-TrEMBL и стабильных протеомов
Ensembl. В режиме Genomic SMART использованы только протеомы полностью секвенированных
геномов; Ensembl для всех остальных протеомов таксона Matazoa, и Swiss-Prot для
других. Полный список геномов в Genomic SMART доступен здесь.
Белковая база данных в Normal SMART имеет значительную избыточность, даже хотя
идентичные белки удалены. Если Вы используете SMART для исследования доменных
архитектур или хотите провести точный доменный подсчёт в различных геномах,
рассмотрите переключение на геномный режим. Числа на страницах аннотации доменов
будут более точными, в то же время не будет много белковых фрагментов, относящихся
к одному и тому же гену в результатах, выданных на запрос архитектуры. Хотя при
этом нужно помнить, что Вы исследуете ограниченный набор геномов.
Переключение между режимами можно осуществить, используя поле
SMART MODE в верхней части страницы.
Авторы: Ivica Letunic, Richard R. Copley, Birgit Pils, Stefan Pinkert,
Jorg Schultz and Peer Bork.
Организации: Wellcome Trust Centre for Human
Genetics Roosevelt Drive, Oxford, UK; Bioinformatik, Biozentrum, Am Hubland,
University of Wuerzburg, Germany.
- Судя по всему, база данных не была сгенерирована
автоматически, а составлялась экспертами.
- Стартовая страница позволяет осуществить настройку
запроса, получить основные сведения об этой базе данных, воспльзоваться словарём,
узнать новости базы данных, написать письмо её администраторам. Для начала работы
с базой данных надо выбрать один из двух режимов: normal или genomic.
Примечание: 21 мая было замечено, что ссылка на базу данных SMART, приведённая выше,
стала вести сразу на страницу для поиска в режиме Genomic SMART, однако, поскольку эта часть задания
была выполнена ранее, а ссылка, возможно, вернётся на ту же страницу, этот пункт было
решено не переделывать.
Что можно найти в базе данных?
Поиск по последовательности и её анализ
Чтобы разобраться, что же всё-таки можно найти с помощью этой базы
данных, проведём пробный запрос, например, в режиме Normal SMART
введём в поле Sequence ID or ACC левого верхнего окошка "Sequence analysis"
ID белка FHUA_ECOLI.
На странице выдачи по этому запросу можно найти следующее:
- Доменную архитектуру белка, включающую:
- Известные домены по данным банка Pfam
- Трансмембранные сегменты, предсказанные программой TMHMM2
- Области спирально свёрнутых спиралей (coiled coil regions), предсказанные программой Coils2
- Области низкой сложности — программой SEG
- Сигнальные пептиды определённые программой SignalP
- Дополнительную информацию о белке и доменной архитектуре:
- Данные по количеству ортологов и альтернативному сплайсингу последовательности
- Данные по количеству белков с той же доменной архитектурой
- Данные по количеству белков с таким же доменным составом
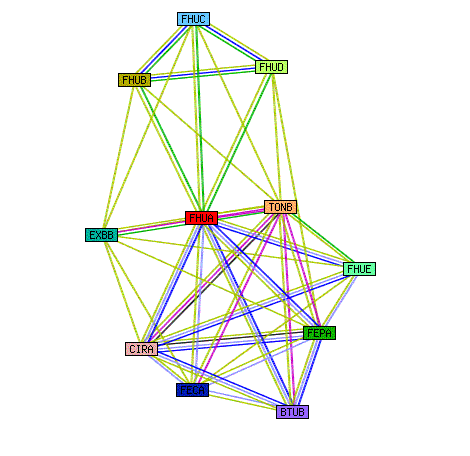
- Диаграмму, на которой можно видеть предсказанные связи белка FHUA_ECOLI с другими белками:
- Связи, отмеченные красным означают свидетельства, полученные путём сопоставления данных
- Зелёные линии означают соседство белков
- Синие линии означают совместную встречаемость белков
Некоторые области, предсказанные для данного белка, не показаны на схеме, отражающей элементы
структуры последовательности. Для этого могут быть две причины: E-value выше порогового, либо перекрывание с другими
элементами структуры; при этом приоритет определяется в следующем порядке:
SMART > PFAM > PROSPERO repeats > Signal peptide > Transmembrane > Coiled coil > Unstructured regions > Low complexity
|
Кроме того, поставив галочки в соответствующих полях страницы запроса, можно получить
ещё дополнительные предсказания:
- Далёкие, изолированные (outlier) гомологи и гомологи известной структуры: данные BLAST и PDB по данной последовательности
- Домены Pfam (эта галочка нужна для предсказания доменов Pfam, если введена последовантельность,
не являющаяся белковой и полноразмерной (аннотированной в какой-либо базе данных)
- Области, содержащие повторения, не перекрывающиеся с доменами, при помощи программы
Prospero
- Внутренние белковые нарушения (intrinsic protein disorder) — программой DisEMBL
- Позиции, соседствующие с интронами (имеет смысл для эукариот)
При попытке поиска по последовательности белка FHUA_ECOLI (747 аминокислот)
сервер смог распознать этот белок, и выдал ту же страницу. Однако, при удалении концевых аминокислот белка (оставлены первые 696),
программа перестала его узнавать, хотя выдача осталась примерно такой же: 4 области низкой
сложности с достоверным E-value, но нашлось множество новых элементов с E-value выше порогового.
Значит, по-видимому, для последовательностей, уже аннотированных SMART, с учётом аннотаций
доменов из Pfam, существуют отдельные страницы с информацией, где можно в том числе,
получить и дополнительную информацию о белке, включая связи с другими белками, а кроме того SMART позволяет делать
предсказания по вводимой последовательности.
То есть для получения, например, предсказаний доменов Pfam по вводимой последовательности
нужно поставить галочку в соответствующем месте страницы запроса. После того, как это было сделано,
выяснилось, что C-концевой домен белка — TonB-dependent receptor продолжает в нём обнаруживаться, но уже
заметно меньшей длины и E-value этого предсказания на примерно двадцать порядков выше. PDB-структура тоже по-прежнему
предсказана с очень хорошим значением E-value.
Поиск по домену или идентификатору GO
Одним из основных назначений базы данных SMART является поиск
белков, обладающих той или иной доменной архитектурой. Этот поиск можно осущетвить, используя
правое окошко поисковой страницы (Normal SMART или Genomic SMART) "Architecture analysis". Для поиска можно применять
и логические связки (AND и AND NOT). Точно такой же поиск можно произвести и по терминам GO, для этого нужно в
соответствующее поле нужно вписать его название или идентификатор GO.
Поскольку у SMART есть своя база данных доменов, отличающаася
от базы Pfam, в поле, где задаётся имя домена нужно писать именно идентификатор домена
SMART. Также пожно ограничить запрос каким-либо таксоном и составить SQL-запрос
(для продвинутых пользователей).
Провести поиск, используя сразу два поля: название домена и идентификатор GO —
нельзя. Если указать в запросе домен, то всё, что указано в поле для терминов GO, будет проигнорировано.
Если ввести какое-нибудь слово или комбинацию слов в поле для терминов GO,
то можно получить список терминов GO, в которых такая комбинация встречается. Для каждого
из этих терминов приведён список белковых доменов, с которыми этот термин ассоциируется в SMART.
Если пометить нужные домены галочкой, то выдача будет содержать объединение белков, имеющих
хотя бы один из этих доменов. В дальнейшем из найденных белков можно выбрать необходимые
и просмотреть их доменную архитектуру, щёлкнув на кнопку "Show Selected"; про любой из этих
белков можно затем получить такую же информацию, как и ранее описанную для белка FHUA_ECOLI.
Ещё можно получить последовательности в формате FASTA, сменив закладку вверху окна.
Удобство выбора сотоит в том, что белки разбиты по таксонам, и можно, например, выбрать для просмотра белки
одного таксона.
Если выбрать нужный таксон или организм в соответствующем поле или на
закладке, то поиск по доменам и по термину GO ведётся уже с учётом таксономии.
Именно для этого сервиса проявляются различия между режимами Normal SMART и
Genomic SMART. В последнем поиск ведётся только по полность секвенированным геномам, что
практически исключает и различные лишние последовательности в выдаче, а также фрагменты; однако,
и число находок заметно уменьшается: например, при поиске по домену RAS Normal SMART
находит 623 последовательности, в то время как Genomic SMART выдаёт 309 находок.
Поиск аннотированных доменов в SMART
Окно для такого поиска "Domains detected by SMART" располагается в нижней части страницы запроса.
Искать домен можно по названию домену или его идентификатору ACC, например, SM00155.
В этом случае пользователь сразу попадает на страницу аннотации данного домена. Если же
поиск вёлся по ключевому слову, то выдаётся список доменов SMART, найденных по нему, и пройдя
по ссылке, соответствующей одному из них, можно также попасть на страницу с аннотацией.
Эта страница содержит:
- Таблицу, в которой представлены:
- Идентификатор SMART (SMART accession number)
- Описание домена в SMART
- Описание домена в Interpro со ссылкой на эту базу данных
- Ссылка на соответствующие термины GO
- Выравнивание выборки белков, содержащих этот домен, и консенсусную последовательность
в различных форматах
- Количество таких доменов в белках базы данных SMART вместе с распределением по
таксонам (числа отличаются между режимами Normal SMART и Genomic SMART.
- Предсказание роли в клетке
- Ссылки на статьи об этом домене, в том числе "Primary literature" — статьи, выбранные
экспертами SMART, и "Secondary literature" — список, сгенерированный автоматически.
Для каждой статьи представлена аннотация (abstract)
- Ссылли на 3D структуры, содержащие этот домен
- Ссылки на другие ресурсы, описывающие этот домен
Среди доменов SMART можно выделить четыре группы: сигнальные, ядерные,
внеклеточные и другие.
Доступна возможность просмотреть описания всех доменов в одном
текстовом файле, а также список последних обновлений доменов.
Кроме того, можно присылать свои варианты доменов, которые, по Вашему
мнению, должны быть добавлены в базу данных SMART.
Наконец, можно оставить запрос на информирование о новых белках
с определённым доменным составом, появившихся в базе данных SMART. Для этого нужно
пройти по ссылке "Alert SMART" в одноимённом окошке на главной странице поиска.
Оценка базы данных SMART
Итак, база данных SMART содержит аннотации некоторых белков, включая их
функциональные связи с другими белками, позволяет вести поиск различных доменов и элементов структуры
введённых последовательностей. Помимо этого, существует возможность определения количества
белков с данным доменам или терминам GO как по всем последовательностям банка данных, так и
белкам из организмов с полностью секвенированным геномом (Genomic SMART), что иногда бывает удобнее.
Для найденных белков можно просмотреть информацию о них, как и информацию о доменах, содержащихся в
этих белках.
SMART содержит собственную базу данных доменов, о которых
предоставлена детальная информация. Единственная сложность в том, что эти домены зачастую
не соответствуют доменам из более привычной для использования базы данных Pfam, однако
SMART обладает большим количеством сигнальных, внеклеточных и ядерных, ассоциированных с хроматином
доменов, что очень привлекательно для исследователей данных областей науки.
Наконец, нельзя не отметить, что база данных имеет довольно привлекательный
и удобный для работы интерфейс.
На главную страницу четвёртого семестра
©
Бурков Борис, Лохматиков Алексей и Лукьянов Михаил,2006