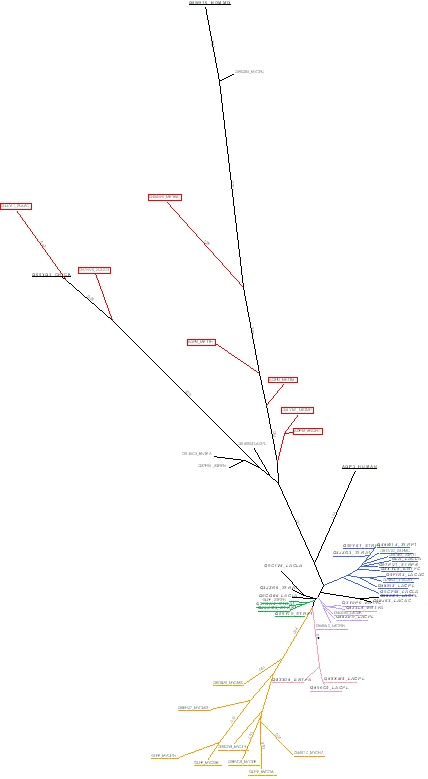
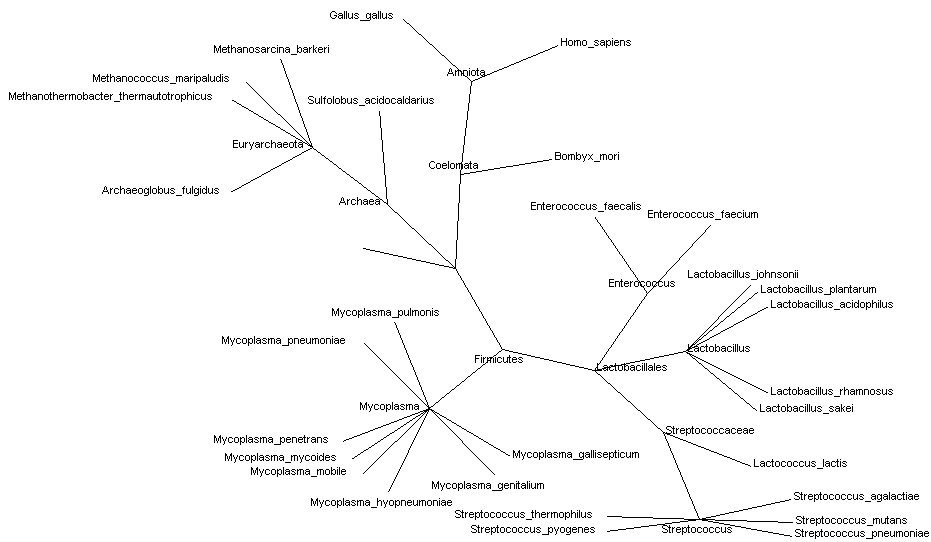
Для более удобного проведения анализа потребовалось вручную восстановить на
дереве полные названия последовательностей, поскольку некоторые из них были обрезаны программой
eprotdist.
Сразу бросается в глаза то, что последовательности из внешней группы (эукариотические), а
также последовательнысти из архей не собираются в два отдельных кластера, как следовало ожидать, а
несколько более разобщены на дереве. Это объясняется описанной выше несогласованностью понимания обозначений
использованных программ. Всё это обязывает к весьма осторожному поиску ортологов и паралогов на
исследуемом дереве и к использованию в целях этого поиска только наиболее достоверные
участки этого дерева.
Больше всего согласуется с таксономическим деревом нижняя часть филогенетического
дерева (по рисунку), представленная бактериальными белками; верхняя часть дерева, ввиду её
малой достоверности и даже явной ошибочности, не исследовалась.
Ортологи — последовательности, возникшие из одного общего предшественника в процессе
видообразования и, как правило, выполняющие одну и ту же функцию, поэтому их следует искать среди белков из
разных организмов, расположенных близко друг от друга по дереву (в одном кластере).
Паралоги — последовательности, возникшие из одного общего предшественника в результате
дупликации одного гена в одном организме и, как правило, выполняющие разные функции. Часто таким организмом
бывает предшественник целой группы организмов (таксона), поэтому паралоги нужно искать среди белков одного организма,
но расположенных в разных, иногда достаточно удалённых друг от друга кластерах.
Ортологами можно считать белки, находящиеся в кластере, помеченном синим цветом.
В их описании из БД Uniprot указано, что это либо аквапорины, либо канальные белки для глицерина. На родство
функции, выполняемой этими белками указывает их близость на филогенетическом дереве, а дальнейшее ветвление
этого кластера во многом соответствует таксономическому ветвлению. Два близкорасположенных белка из организма
Streptococcus pneumoniae по сути, можно считать, выполняют одну функцию, но, поскольку последовательности
их немного различаются, не исключено, что они произошли в результате дупликации гена, а значит,
являются паралогами. Также паралогами могут считаться белки из Lactococcus lactis.
Зелёным и фиолетовым цветами выделены другие кластеры предполагаемых ортологов среди
белков семейства Lactobacillales. Поле Description Uniprot содержит такие же описания для этих
последовательностей, что и для предыдущей группы ортологов (это неудивительно, ведь искались белки одного семейства),
но, видимо, их функции всё-таки немного отличаются, поэтому они оказались в небольшом отдалении от предыдущей группы
ортологов. Возможно даже, что две последние группы являются ортологами между собой, просто
принадлежат к организмам разных таксономических ветвей, из-за чего немного удалены друг от друга. Нельзя
сбрасывать со счетов и возможное ортологическое сходство и с группой из белков Q9CIV5_LACLA,
Q3JZN6_STRA1 и Q9CIV5_LACLA; может быть на этот вопрос смогло бы ответить дерево, построенное
чуть точнее. А вот пары белков Q3JZN6_STRA1 и Q3K3A4_STRA1, Q9CIV5_LACLA и Q9CG66_LACLA,
Q88ZE9_LACPL, Q88ZX3_LACPL являются паралогичными друг другу (последняя пара — более далёкие),
паралогами приходятся и белки из кластера, выделенного зелёным цветом, взятые из организма
Streptococcus pneumoniae, хотя возможна и ошибка в секвенировании, ведь они отличаются всего на одну аминокислоту.
Кроме того, в последних двух группах ортологов немало последоватедьностей, паралогичных белкам из
группы, выделенной синим цветом.
В кластере, выделенном розовым цветом, также присутствуют 2 возможных паралога и их ортолог
из организма родственной группы. Эти белки также являются паралогами для некоторых других из
рассмотренных выше групп.
Наконец, есть целая группа ортологов среди белков рода Mycoplasma (выделена оранжевым цветом).
Возможно, что эти белки ортологичны одной из групп ортологов из семейства Lactobacillales, но, в любом случае,
определение среди всех рассмотренных выше групп явных ортологов исходя из имеющегося дерева представляется затруднительным.
Интересно бы было посмотреть связь этой группы с белком Q98Q85_MYCPU, которая, безусловно, должна
присутствовать, но, к сожалению, из-за ошибок в реконструкции дерева этот белок оказался на неправильном месте.