С помощью SRS получили из UniProt последовательности заданного рибосомального белка из митохондрий разных эукариот. Таких белков оказалось достаточно много (129), поэтому решено было ограничится теми из них, которые входят в БД SwissProt. Таким образом, было получено 25 белковых последовательностей. Выборка содержит главным образом белки растений, простейших и грибов. Запрос:
- Description ribosomal protein s12
- Taxonomy eukaryota
- Organelle mitochondrion
Так как у многих эукариот гены рибосомных белков митохондрий "перешли" в ядерный геном и закодированы в различных хромосомах, то решено было дополнительно провести ещё поиск в БД SwissProt со следующим запросом:
- Description 28 ribosomal protein s12, mitochondrial
- Taxonomy eukaryota
В результате к выборке добавилось ещё 4 последовательности, это белки млекопитающих. Сохранили эти 29 B следовательностей в одном файле в формате FASTA. Сохранили коды доступа и идентификаторы в двух отдельных файлах. Согласно Pfam всего может быть 7 различных доменных структур у данных белков. Последовательность белка человека была рассмотрена подробнее. С помощью Pfam установили доменную структуру белка (ID - RT12_HUMAN)
Выравнивание доменов было взято из Pfam с идентификатором PF00164, с помощью скрипта на bash в нем были оставлены только найденные в SRS последовательности. Колонки состоящие из одних гэпов удалялись. Выяснилось, что 5 из29-ти найденных последовательностей не присутствуют в выравнивании Pfam, хотя на странице SRS этих белков есть ссылка на страницу Pfam с идентификатором PF00164. Данные последовательности были удалены из общей выборки (4 из первых 25-ти последовательностей и 1 из выборки с 4-мя последовательностями).
С помощью программы muscle на сервере kodomo для оставшихся 24 последовательностей было построено множественное выравнивание, к которому затем были добавлены веса с помощью pwf из пакета PFTOOLs.
Сравнение полного выравнивания и выравнивания доменов показало, что они практически идентичны. Следовательно, полное выравнивание можно смело использовать для дальнейших целей.
По взвешенному выравниванию построили профиль с помощью pfmake. Затем нормировать профиль с помощью autoscale. Простой и нормированный профили отличаются только записями R1, R2 (коэфициенты функции нормирования) и SCORE(порог для поиска по умолчанию) в поле MA.
После построения профиля по нему были проведены поиски среди альфапротеобактерий и гаммапротеобактерий с различными значениями порога для определения оптимального порогового значения.
Результаты поиска по двум группам бактерий с разными значениями порога
| гаммапротеобактении | альфапротеобактерии | |||||
| порог | всего | Онтология "клеточный компонент" | GO "малая субчастица рибосомы" | всего | Онтология "клеточный компонент" | GO "малая субчастица рибосомы" |
| 5.0 | 601 | 517 | 158 | 327 | 279 | 82 |
| 10.0 | 141 | 141 | 141 | 74 | 74 | 74 |
| 30.0 | 141 | 141 | 141 | 74 | 74 | 74 |
По результатам поиска было выбрано значение порога, равное 10.0, так как при этом пороговом значении все найденные белки как для альфапротеобактерий, так и для гаммапротеобактерий имели GO "малая субчастица рибосомы". Как видно, при таком пороге мы теряем 17 и 12 белков из гаммапротеобактерий и альфапротеобактерий соответственно, которые находятся при пороге, равном 5. Но при этом пороге много "лишних" белков; при порогах 6, 7, 8, 9 число находок с GO "малая субчастица рибосомы" становится равной таковому при поиске с порогом 10.0.
Ниже представлена гистограммыа распределения долей нормированных весов находок для
альфапротеобактерий и гаммапротеобактерий
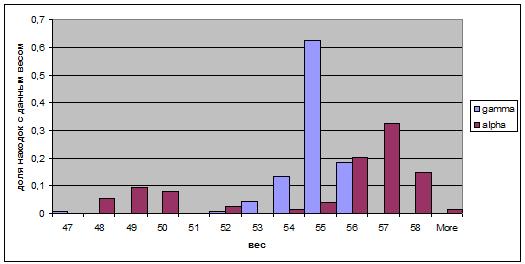
Гистограмма была построена средствами Excel по результатам поиска по построенному выше профилю для альфа- и гаммапротеобактерий с порогом 10.
Из гистограммы распределения видно, что вес
наибольшего числа находок среди гаммапротеобактений меньше, нежели вес
наибольшей части находок среди альфапротеобактерий.
Наибольшим весом обладает последовательность из альфапротеобактерии
Maricaulis maris (AC - Q0ANP5)
По распрделениям весов находок посредством программы STADIA был выполнен тест Уилкоксона. Тест показал, что различия в распределениях между выборками есть.
Вилкоксон=1.301E4, Z=5.125, Значимость=0, степ.своб = 141,74 Гипотеза 1: <Есть различия между медианами выборок>
С помощью функции "Описательная статистика" для обеих выборок были установлены выборочное среднее, а так же медиана.
альфапротеобактерии
Среднее Медиана 54.35 55.81
гаммапротеобактерии
Среднее Медиана 54.28 54.43Как видно, оба показателя для альфапротеобактерий выше таковых у гаммапротеобактерий. Это согласуется с предыдущим результатом. То есть, можно сделать заключение, что находки среди альфапротеобактерий лучше. Это значит, что профилю, построенному по митохондриальным белкам эукариот, более соответствуют белки альфапротеобактерий, нежели гаммапротеобактерий. Это может в какой-то степени свидетельствует о большей близости белка s12 митохондрий к альфапротеобактериям.
Получили 6 последовательностей рибосомальных белков s12 из разных родов Firmicutes. Построили объединенное выравнивание митохондриальных белков и белков из рибосом альфа-, гаммапротеобактерий и фирмикут посредством программы muscle на сервере kodomo.
С помощью программы proml пакета PHYLIP по полученному выравниванию было построено дерево. Out-группой была выбрана одна из последовательностей фирмикут.
Ниже представлено изображение этого дерева, полученное с помощью программы GeneMaster. Листья, отвечающие последовательностям альфапротеобактерий покрашены красным цветом, гаммапротеобактериям - синим цветом, последовательностям митохондриальных белков эукариот зелёным цветом и формикутам фиолетовым цветом.
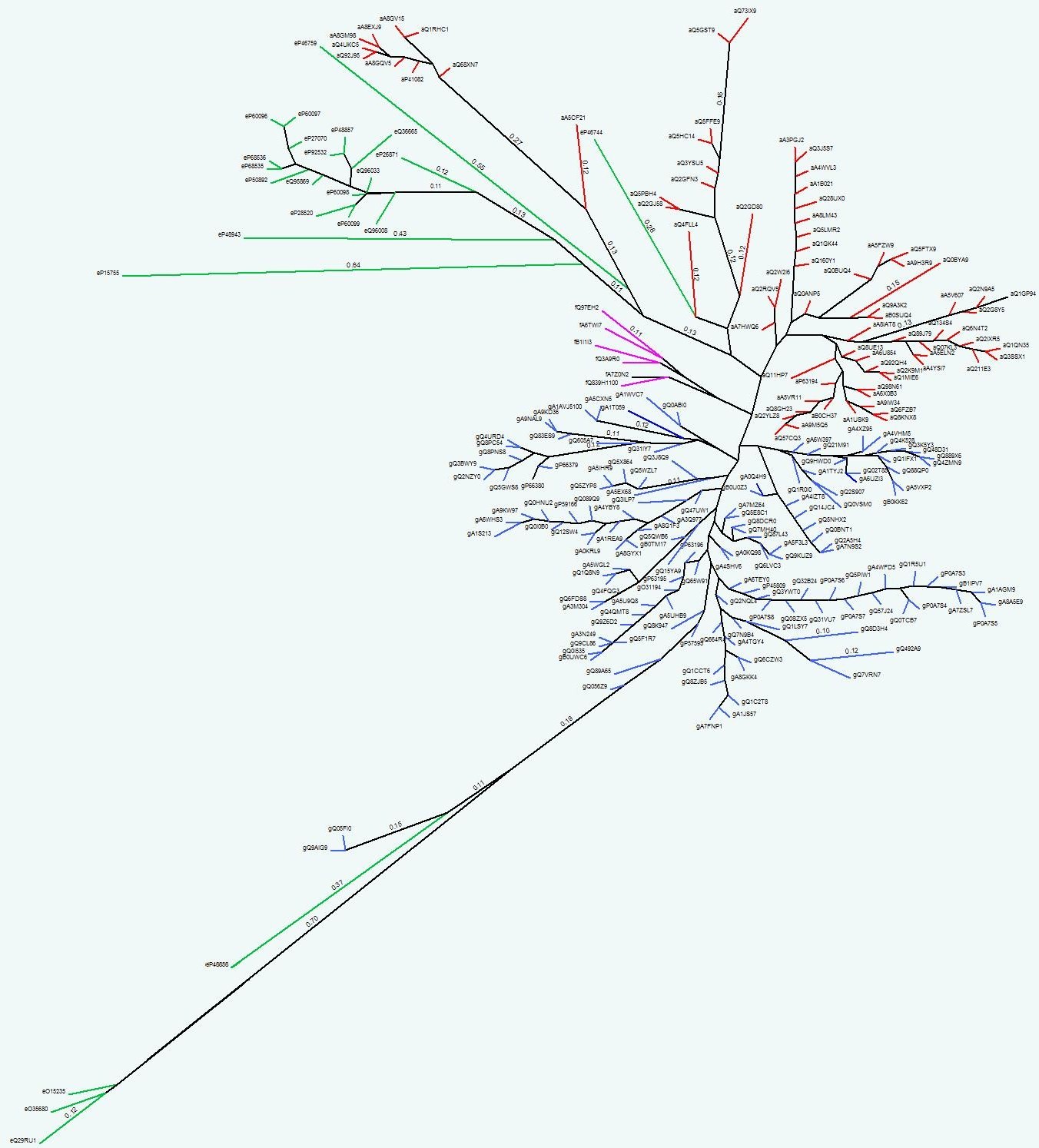
Видно, что эукариотические последовательности разделились на 2 неравные группы.
Первая (большая) ближе к альфапротеобактериям. Все последовательности этой группы принадлежат к растениям, грибам и простейшим. Наиболее близкий организм к ним из альфапротеобактеиям, как я считаю, это Orientia tsutsugamushi (Rickettsia tsutsugamushi) (AC - A5CF21).
Вторая (меньшая) группа содержит всего 4 последовательности, 3 из которых принадлежат млекопитающим. Эта группа явно ближе к гаммапротеобактериям, ближайшие представиетели Candidatus Carsonella (AC - Q9AIG9).
Для исследуемых последовательностей были определены попарные эволюционные расстояния с помощью программы protdist пакета PHYLIP. Полученную матрицу импoртировали в Exel и построили гистограмму распределения попарных расстояний между рибосомальными белками митохондрий эукариот и белками из альфа- и гаммапротеобактерий.
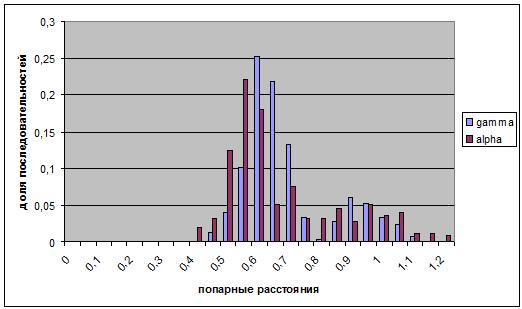
Расстояние, на которое приходится максимум находок для альфапротеобактерий немного меньше такового для гаммапротеобактерий, что в первом приближении говорит о большей эволюционной близости рибосомального белка s12 альфапротеобактерий к рибосомальному белку митохондрий эукариот, нежели гаммапротеобактерий.
