| Предсказание парных выравниваний. | |||||||||||||||||||||||||||||||||||||
| Главная |
Выравнивание последовательностей и поиск гомологичных блоков. Было произведено множественное выравнивание последовательностей из файла aln_11.fasta . Из него было выбрано парное выравнвиание двух последних последовательностей (рис. 1). Построение глобального выравнивания последовательностей в формате fasta Выравнивание, полученное с помощью приложения needle пакета EMBOSS (gap opening penalty 10.0, gap extension penalty 0.5) 3stk_a.needle :
/*>3STK_A
MAGSSFSGKYQLQSQENFEAFMKAIGLPEELIQKGKDIKGVSEIVQNGKHFKFTITAGS-
-KVIQNEFTVGEECELETMTGEKVKTVVQLEGDNKLVTTFKNIK------SVTELNGDII
TNTMTLGDIVFKRISKRIGT
/*>1SA8_A
----------------AFDGTWKVGGLKLTITQEGN---------------KFTVKESSN
FRNIDVVFELGVDFAYSLADGTELTGTWTMEG-NKLVGKFKRVDNGKELIAVREISGNEL
IQTYTYEGVEAKRIFKKE—
Построение локального выравнивания Наилучшее локальное выравнивание, полученное с помощью приложения water пакета EMBOSS (gap opening penalty 10.0, gap extension penalty 0.5) 3stk_a.water :
>3STK_A
FEAFMKAIGLPEELIQKGKDIKGVSEIVQNGKHFKFTITAGS--KVIQNEFTVGEECELE
TMTGEKVKTVVQLEGDNKLVTTFKNIK------SVTELNGDIITNTMTLGDIVFKRISKR
>1SA8_A
FDGTWKVGGLKLTITQEGN---------------KFTVKESSNFRNIDVVFELGVDFAYS
LADGTELTGTWTMEG-NKLVGKFKRVDNGKELIAVREISGNELIQTYTYEGVEAKRIFKK
Выравнивание двух заведомо негомологичных последовательностей Выравнивание, полученное с помощью приложения needle пакета EMBOSS needle (gap opening penalty 10.0, gap extension penalty 0.5) D8HZ92.needle: >D8HZ92_AMYMU D8HZ92 AHBA synthase OS=Amycolatopsis mediterranei (strain U-32) GN=rifK PE=1 SV=1 MNARKAPEFPAWPQYDDAERNGLVRALEQGQWWRMGGDEVNSFEREFAAHHGAAHALAVT NGTHALELALQVMGVGP-GTEVIVPAFTFISSSQAAQRL------GAVTVPVD-----VD AATYNLDPEAVAA-------AVTPR------TKVIMPVHMAGLMADMDALAKISADTGVP LLQDAAHAHGARWQGKRVGELDSIATFSFQNGKLMTAGEGGAVVFPDGETEKYETAFLRH SCGRPRDDRRYFHKIAGSNMRLNEFSASVLRAQLARL-----DEQIAVRDERWTLLSRLL GAIDGVVPQGGDVRADRNSHYMAMFRIP--GLT--------------EER-RNALVDRLV EAGLPAFAAFRAIY-RTD------AFWELGAPDESVDAIARRCP----NTDAISSDCVWL HHRVLLAGEPELHATAEIIADAVARA >YP_909930.1 chorismate mutase ------------------------------------------------------------ ------MSARKLFYLGPQGT------FTHQAAVNAAQELARFEPQGFDLMPMDDVPQILD AAQHG-DGWGIVAWENNVEGYVVPNLDALIDAKDLVGFARVGVNVEFDAYVAQGADPAEA RIA-TAHPHGLA-QCKRF-----IAEHRLSTQPATSNAAACRDLIPG------EIAFGPA ICGELYDITRIGTAIQDYQGAATDFLVLSPRAEVARLLAKPRAEANVEYESVLTLIPLVT G--PGVLANLLDVFRDAGLNMTSFISRPIKGRTGTYSFIVTLDAAPWEERFRDALVE-IA EHG--DWAKTLAVYPRREHPNPPVTSWML--PQGGVRLDDSHLPDDWQNDETVRRELMW- -------------------------- Выравнивание, полученное с помощью приложения water пакета EMBOSS needle (gap opening penalty 10.0, gap extension penalty 0.5) D8HZ92.water: >D8HZ92_AMYMU D8HZ92 AHBA synthase OS=Amycolatopsis mediterranei (strain U-32) GN=rifK PE=1 SV=1
ALQVMGVGP-GTEVIVPAFTFISSSQAAQRL------GAVTVPVD-----VDAATYNLDP
EAVAA-------AVTPR------TKVIMPVHMAGLMADMDALAKISADTGVPLLQDAAHA
HGARWQGKRVGELDSIATFSFQNGKLMTAGEGGAVVFPDGETEKYETAFLRHSCGRPRDD
RRYFHKIAGSNMRLNEFSASVLRAQLARL-----DEQIAVRDERWTLLSRLLGAIDGVVP
QGGDVRADRNSHYMAMFRIP--GLT--------------EER-RNALVDRLVEAGLPAFA
AFRAIY
>YP_909930.1 chorismate mutase
ARKLFYLGPQGT------FTHQAAVNAAQELARFEPQGFDLMPMDDVPQILDAAQHG-DG
WGIVAWENNVEGYVVPNLDALIDAKDLVGFARVGVNVEFDAYVAQGADPAEARIA-TAHP
HGLA-QCKRF-----IAEHRLSTQPATSNAAACRDLIPG------EIAFGPAICGELYDI
TRIGTAIQDYQGAATDFLVLSPRAEVARLLAKPRAEANVEYESVLTLIPLVTG--PGVLA
NLLDVFRDAGLNMTSFISRPIKGRTGTYSFIVTLDAAPWEERFRDALVE-IAEHG--DWA
KTLAVY
Сравнение различных выравниваний Информация о различных выравниваниях представлена в таблице 1.
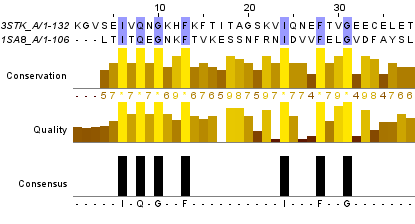
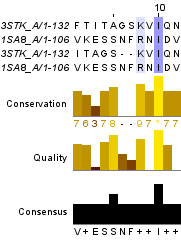
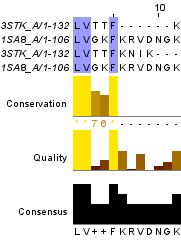
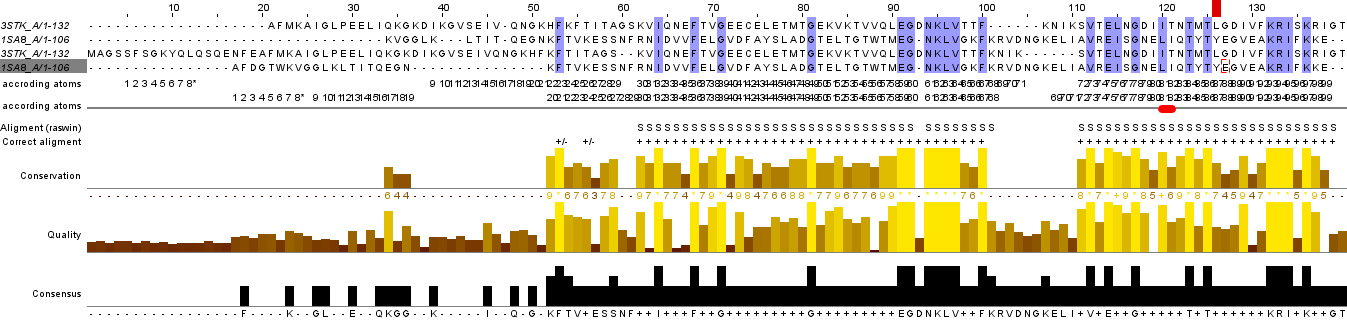
Как видно из таблицы 1, выравнивания гомологичных и негомологичных позиций не сильно отличаются по числу идентичных и сходных остатков. Но заметна разница при переходе от глобального выравнивания к локальному. Это указывает на отсутствие протяжённого участка гомологии, как в случае выравнивания гомологичных последовательностей. Сравним парное выравнивание, полученное из множественного, с глобальным выравниванием, полученным с помощью needle. В первом выравнивании по сравнению со вторым больше число идентичных остатков, но меньше число похожим остатков. Также меньше число гэпов. Возможно, первое выравнивание более корректно по той причине, что оно являлось частью множественного выравнивания и участки, выявленные алгоритмом как гомологичные у всех последовательностей, с большей вероятностью являются таковыми. Выравнивание совпадают на 62-100, 111-140 позициях. Фрагмент выравнивания слева от участка несоответствия выравниваний (1-61 позиции включительно) изображён на рис. 2. Аналогичный фрагмент справа от другого участка несоответствия выравниваний (101-111 позиции включительно) изображён на рис. 3. Проверка правильности парных выравниваний гомологичных последовательностей С помощью программ SupCheck и RasMol была проверена правильность выравнивания путём совмещения пространственной структуры белковых последовательностей. Для второго выравнивания было выявлено 32 ошибки второго рода (остатки совмещаются в пространстве, но не находятся в одной колонке, либо эта колонка не была признана достоверной до пространственного выравнивания) и 31 ошибка первого рода (аинокислотные остатки стоят в одной колонке в выравнивании, но не совмещаются в пространстве). Для первого выравнивания, соответственно 27 ошибок второго рода и 8 первого. Выравнивание изображено на рис. 4. Проект можно скачать по следующей ссылке . |
||||||||||||||||||||||||||||||||||||
| Обо мне | |||||||||||||||||||||||||||||||||||||
| Ссылки | |||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||
© Denis Moshensky, 2013
Дата последнего изменения: 26.05.2014
Задавайте вопросы по электронной почте