| BLAST | |||||||||||||||
| Главная |
Поиск гомологов белка AHBA synthase (идентификатор UniProt D8HZ92_AMYMU) с помощью программы protein blast Параметры поиска гомологичных последовательностей были выбраны по умолчанию. Среди результатов поиска была выбрана последовательность белка аминотрансферазы организма Pseudomonas aeruginosa PAO1. Информация о сходстве найденного белка с данным представлена в таблице 1. Расшифровка столбцов таблицы приведена ниже.
Descriprion - описание найденной белковой последовательности; Выравнивание для данного белка и найденного гомолога:
Query 22 GLVRALEQGQWWRMGGDEVNSFEREFAAHHGAAHALAVTNGTHALELALQVMGVGPGTEV 81
G+ R L GQ+ + G EV E A GA + ++ NGT AL++ +GVGPG EV
Sbjct 21 GIQRVLRHGQY--ILGPEVTELEDRLADFVGAKYCISCANGTDALQIVQMALGVGPGDEV 78
Query 82 IVPAFTFISSSQAAQRLGAVTVPVDVDAATYNLDPEAVAAAVTPRTKVIMPVHMAGLMAD 141
I P FT++++++ LGA V VD+D TYNLDP+ + AA+TPRTK I+PV + G AD
Sbjct 79 ITPGFTYVATAETVALLGAKPVYVDIDPRTYNLDPQLLEAAITPRTKAIIPVSLYGQCAD 138
Query 142 MDALAKISADTGVPLLQDAAHAHGARWQGKRVGELDSIATFSFQNGK-LMTAGEGGAVVF 200
DA+ I++ G+P+++DAA + GA ++GKR L ++A SF K L G+GGA+
Sbjct 139 FDAINAIASKYGIPVIEDAAQSFGASYKGKRSCNLSTVACTSFFPSKPLGCYGDGGAIFT 198
Query 201 PDGETEKYETAFLRHSCGRPRDDRRYFHKIAGSNMRLNEFSASVLRAQLARLDEQIAVRD 260
D E RH DRRY H G N RL+ A++L +L +E+IA+R
Sbjct 199 NDDELATAIRQIARHG-----QDRRYHHIRVGVNSRLDTLQAAILLPKLEIFEEEIALRQ 253
Query 261 E 261
+
Sbjct 254 K 254
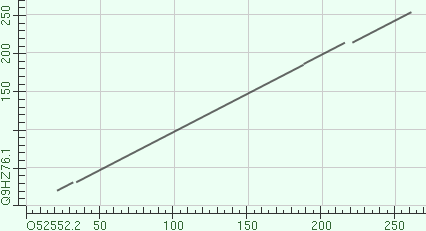
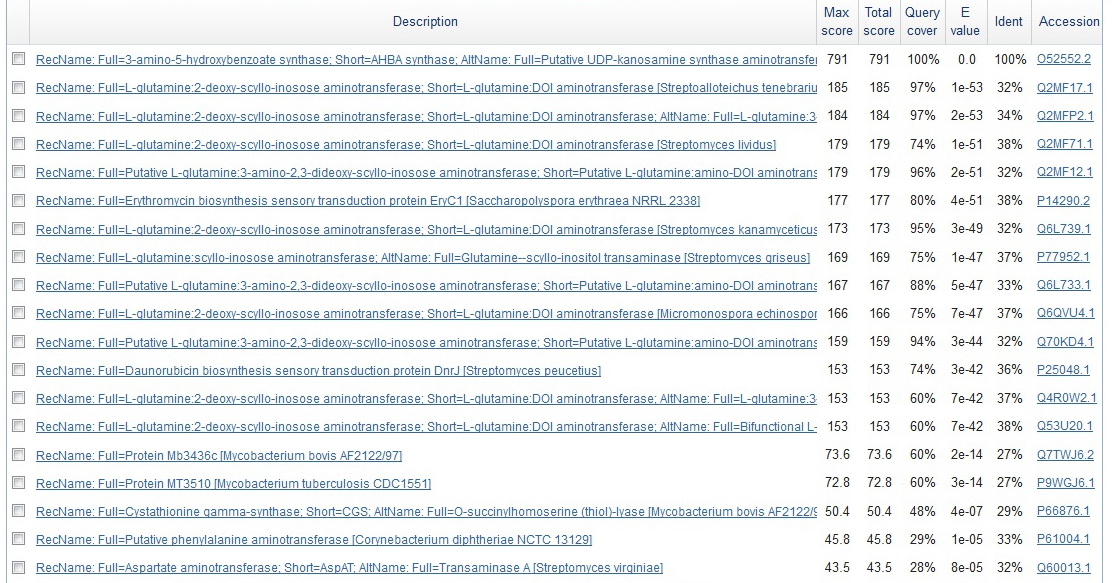
Участок сходства имеет координаты 22-261 для последовательности AHBA synthase A. mediterranei и 21-254 для найденной. Найденная последовательность имеет длину 359 аминокислотных остатков. Протяжённость выравнивания 241 остаток. Примерно 67% длины найденной последовательности было выровнено с участком query последовательности AHBA synthase A. mediterranei. 97 (40% от числа позиций выравнивания) остатков найденной последовательности идентичны остаткам последовательности AHBA synthase A. mediterranei, сходных остатков 43 (18% от числа позиций выравнивания). Карта локального сходства между query и выбранной находкой С помощью одного из сервисов BLAST была построена карта локального сходства участка query и участка найденной последовательности (рис. 1). Из dot-матрицы видно, что последовательности обладают высоким уровнем сходства, за исключением небольших участков в началае и конце выравнивания. Поиск гомологов последовательности AHBA synthase A. mediterranei в Uniprot/SwissProt Среди белков представителей эукариот гомологов данного белка найдено не было. Дальнейший поиск гомологов был произведён среди белков представителей типа Actinobacteria. Было найдено 18 последовательностей, для которых величина E-value имеет значение менее 0.001. Скриншот с результатами поиска представлен на рис. 2. Было сохранено выравнивание 11 найденных последовательностей. Выравнивание представлено на рис. 3. Всего было найдено 40 (9.6% от длины выравнвиания) абсолютно консервативных позиций. Некоторые колонки были выделены программой Jalview, несмотря на то, что не все аминокислоты в этой позиции совпадают. Такие колонки не учитывались. Функционально консервативных позиций был найдено 43 (10% от длины выравнвиания). Количество консервативных позиций достаточно для того, чтобы сделать вывод о гомологичности последовательностей. Файл с выравнивание можно скачать по ссылке. |
||||||||||||||
| Обо мне | |||||||||||||||
| Ссылки | |||||||||||||||
|
|
|||||||||||||||
© Denis Moshensky, 2013
Дата последнего изменения: 18.09.2014
Задавайте вопросы по электронной почте