| Сравнение пространственных структур белков | |||||||||||||||
| Главная |
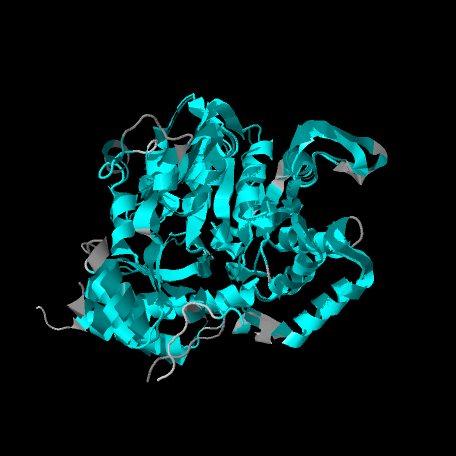
Сравнение структуры белка AHBA synthase (идентификатор PDB 1B9H) и белка DesV (идентификатор PDB 2OGA) Поиск белков со схожей структурой осуществлялся с помощью сервиса PDBeFold. Параметры поиска стандартные. Сервисом PDBeFold было проанализировал 100424 PDB-файлов и 264240 белковых цепочек. В итоге было выбрано 82 белка, чья структура наиболее сходна со структурой белка AHBA synthase (идентификатор 1B9H). Для анализа была выбрана цепь A белка с идентификатором 2OGA. В таблице 1 перечислены его основные параметры. При выборе были учтены следующие параметры: процент совпадающих аминокислотных остатков (%seq) - 33, мера сходства структур (RMSD) - 1.56 ангстрем, число сопоставленных альфа-атомов (N_align) - 344.
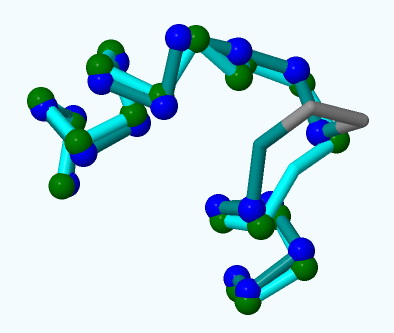
DesV также, как и AHBA synthase, является PLP (pyridoxal-5'-phosphate) зависимым ферментом (гидролазой). Выделен из бактерии Streptomyces venezuelae. На рис. 1 представлено сопоставление цепей исходного белка и находки. Рассмотрим участки цепей обоих белков для более точного сравнения структур (см. рис. 2). Я выделил в Jmol участки 22-43 цепи AHBA synthase (CHAIN1) и 29-48 цепи DesV (CHAIN2). Командой SELECT WITHIN(1.6,CHAIN1.CA) AND CHAIN2 OR WITHIN(1.6,CHAIN2.CA) AND CHAIN1 можно выделить все С-альфа атомы обеих цепей, которые находятся на расстоянии не более 1.6 ангстрем друг от друга. Для сравнения, расстояние между соседними С-альфа атомами одной цепи составляет 3,8 ангстрема. В результате было обнаружено по 18 атомов на каждой цепи, на рис. 2 они показаны шариками. Выделен также участок расхождения цепей. Внутри него цепи имеют разную длину, совпадающих аминокислот практически нет. |
||||||||||||||
| Обо мне | |||||||||||||||
| Ссылки | |||||||||||||||
|
|
|||||||||||||||
© Denis Moshensky, 2013
Дата последнего изменения: 13.09.2014
Задавайте вопросы по электронной почте