| Восстановление предкового состояния доменной структуры | |||||||||||||||||
| Главная |
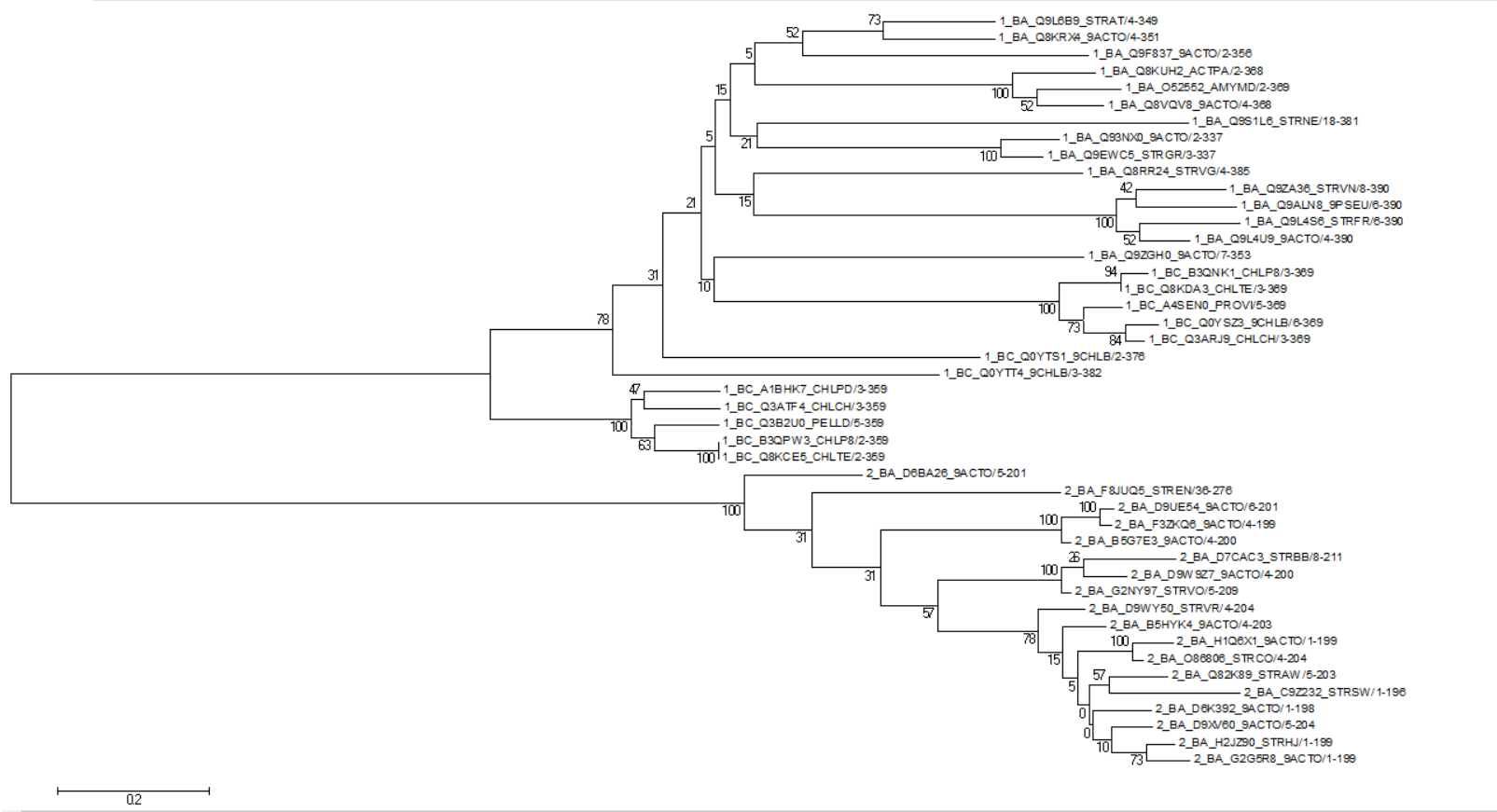
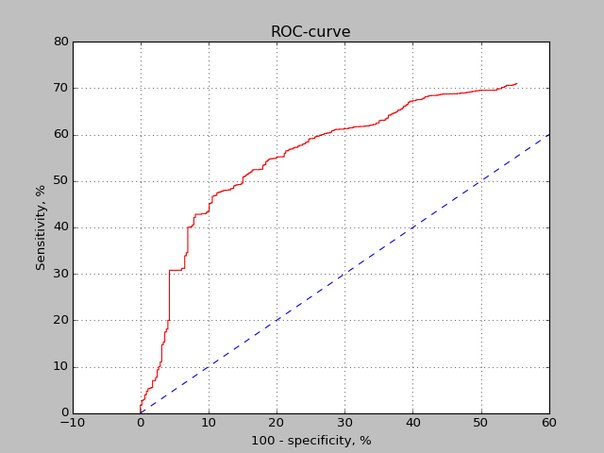
Выбор объектов для изучения и построение выравнивания Было выбрано семейство DegT_DnrJ_EryC1 (PF01041). Белки этого семейства являются пиридоксальфосфат зависимыми аминотрансферазами. В базе данных Pfam для данного семества известно 29 доменных архитектур, 8204 последовательности, одно взаимодействие, 78 структур и для 3491 организма было показано наличие в их геноме белков с доменом DegT_DnrJ_EryC1. Список доменных архитектур, включающих этот домен, представлен на странице Pfam . Можно заметить, что чаще всего в белке наблюдается лишь один домен DegT_DnrJ_EryC1 (7817 последовательностей), на порядок реже наблюдается два домена DegT_DnrJ_EryC1 (154 последовательности). Другие доменные архитектуры наблюдаются сильно реже, максимум в 18-ти последовательностях. Доменных архитектур, в которые входит не менее 20 последовательностей, оказалось две: 1. Доменная архитектура, похожая на Q8KUH2_ACTPA (доменная структура DegT_DnrJ_EryC1 из Actinosynnema pretiosum) содержит один домен DegT_DnrJ_EryC1 с координатами 15-382 аминокислотных остатков в белке длиной 388 аминокислотных остатков 2. Доменная архитектура, похожая на Q0IDI3_SYNS3 (доменная структура DegT_DnrJ_EryC1 из Synechococcus sp.) содержит два домена DegT_DnrJ_EryC1 с координатами 13-301 и 298-391 аминокислотных остатков в белке длиной 44 аминокислотных остатка С помощью скриптов была получена таблица, в которой представлена информация о структурах домена. Таблицу можно скачать ниже. Полученные в результате файлы: Таблица с идентификаторами и таксономией Список идентификаторов выбранных белков Построение филогенетического дерева последовательностей домена Далее с помощью программы MEGA и алгоритма Neighbour-joining с количеством бутстрэп-реплик 100 было построено дерево по подправленному выравниванию ( проект JalView . Выбранные одно и двухдоменные архитектуры сильно различаются, что видно на филогенетическом дереве (рис. 1). Двухдоменная архитектура встречается только у актинобактерий, а однодоменная - еще и у хлоробиевых бактерий. Поэтому, скорее всего, предковой являлась однодоменная архитектура, что логично. Дерево в формате Newick можно скачать здесь. Построение профиля подсемейства Для постороения HMM профиля были использованы последовательности , образующие отдельную ветвь на дереве (рис. 1). Далее был использован HMMER: Для построения roc-кривой положительными находками считались белки, содержащие только один домен DegT_DnrJ_EryC1. В таблице 1 приведена информация о находках при выбранном пороге по E-value.
|
||||||||||||||||
| Обо мне | |||||||||||||||||
| Ссылки | |||||||||||||||||
|
|
|||||||||||||||||
© Denis Moshensky, 2013-2015
Дата последнего изменения: 24.05.2015
Задавайте вопросы по электронной почте