
Пробные Выравнивания
| Главная |
| Первый семестр |
| Второй семестр |
Таблица 1. Результаты поиска гипотетических гомологов белка 1IHF_ECOLI
| Поиск по БД Swiss-Prot | Поиск по БД PDB | Поиск по БД "nr" | |
| 1. Лучшая находка (в принципе должна соответствовать заданному белку) | |||
| Идентификатор БД | P0A6Y1 | pdb|1IHF|B | NP_286787.1 |
| E-value | 6e-50 | 4e-51 | 1e-48 |
| Вес (в битах) | 195 | 195 | 195 |
| % идентичности | 100% | 100% | 100% |
| Найдены ли другие белки с теми же значениями E-value и веса в битах? Если найдены, то укажите общее число и приведите один идентификатор (любой, но желательно Swiss-Prot ID) |
- | pdb|1OWG|B | - |
| 2. Сколько хороших кандидатов в гомологи найдено? (число находок в списке описаний, Descriptions, с E-value < 1E-10) | 197 | 34 | 895 |
| 2. "Худшая" находка (последняя в выдаче с E-value < 1) | |||
| Номер находки в списке описаний (Descriptions) | 395 | 43 | 2430 |
| Идентификатор БД | Q90610 | pdb2Z62|A | ref|YP_303251.1 |
| E-value | 0,20 | 0,92 | 0,92 |
| Вес (в битах) | 33.9 | 28.1 | 36.2 |
| % идентичности | 30% | 30% | 26% |
| % сходства | 52% | 57% | 47% |
| Длина выравнивания | 55 | 33 | 69 |
| Координаты выравнивания (номера первых и последних а.о. выровненых участков последовательности IHFB) | 36_91 | 20_52 | 1_69 |
| % гэпов | 0% | 0% | 0% |
2. Вес в битах сравнения белка с самим собой во всех трех поисках одинаков, а e-value различны т. к. оно зависит от числа последовательностей в банке, по которому проходил поиск
3. Меньше всего потенциальных гомологов в PDB больше всего в nr т.к. всего последовательностей меньше в PDB и больше в nr.
4. "худшие" находки - различны т.к. количество последовательностей в банках различно. Я полагаю, что лучшая из них Q90610 т.к. у неё наибольшее различие в e-value со следующим за ней.
2).Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам
| Таксоны | Homo sapiens | Archea | Actinobacteria | Alteromonadales | Vibronacea |
| Идентификатор БД | no results | ref|YP_002567214.1 | ref|ZP_03821322 | sp|A1S6D6 | sp|Q6LPE4.1 |
| E-value | - | 4e-08 | 3e-13 | 4e-39 | 2e-42 |
| Вес (в битах) | - | 55.1 | 73.6 | 157 | 168 |
| % идентичности | - | 42% | 37% | 82% | 84% |
| % сходства | - | 60% | 59% | 89% | 94% |
| Длина выравнивания | - | 60 | 91 | 91 | 91 |
| Координаты выравнивания (номера первых и последних а.о.) | - | 23_71 | 1_91 | 1_91 | 1_91 |
| %гэпов | - | 14% | 1% | 0% | 0% |
Во всех исследовавшихся таксонах, кроме Homo sapiens были надены гомологи изучаемого белка(IHFB_ECOLI)
3.Поиск заданного, но неизвестного белка по его фрагменту Здесь:
| Поиск по фрагменту | Поиск по полной последовательности | |
| AC лучшей находки | Q4UKH2.2 | Q4UKH2.2(ID: DBH_RICFE) |
| E-value | 2e-15 | 8e-53 |
| вес | 79.1 | 204 |
| Найдены ли другие белки с теми же значениями веса и e-value | Q92J57.2 | - |
Q4UKH2 1 MNKTEFIAFMTDHGHNHKHASHKTLTKADAEKALNLVLDSVISAIKSHHNINITGFGSFE 60
M K+E I + H KT+ E A+ +L+ + S + I I GFGSF
IHFB_ECOLI 1 MTKSELIERLATQ---QSHIPAKTV-----EDAVKEMLEHMASTLAQGERIEIRGFGSFS 52
Q4UKH2 61 IHHRKAREGRNPKTGAKMKIDAYNQPTFRAGRKMKEACN 99
+H+R R GRNPKTG K++++ P F+ G+++++ N
IHFB_ECOLI 53 LHYRAPRTGRNPKTGDKVELEGKYVPHFKPGKELRDRAN 91
в первом задании блока выравнивались фрагменты с номерами позиций в данном выравнивании 3_26консервативные позиции в выравнивании BLAST и ручного выравнивания совпадают, но гэп я расположил иначе
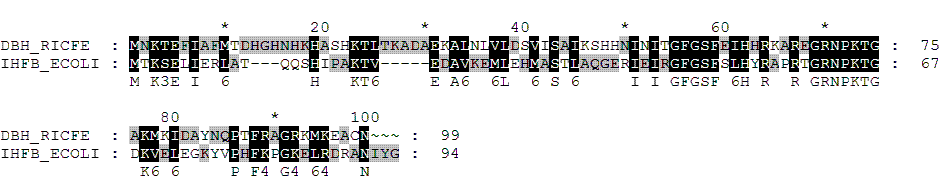
4).Глобальное выравнивание (needle)
Локальное выравнивание (water)
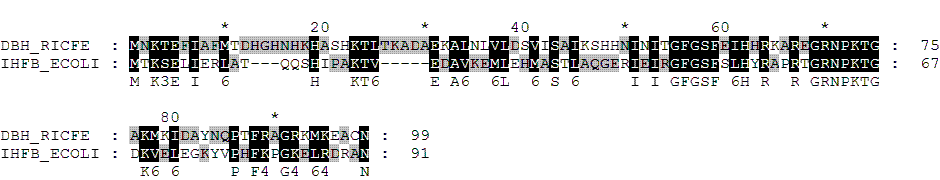
выравнивание BLAST идентично локальному выравниванию и за исключением концевого участка IYG глобальному выравниванию приштрафе за открытие гэпа 11 и штрафе за продолжение 1.
| Главная | Второй семестр |