
| Главная |
| Первый семестр |
| Второй семестр |
| Pymol |
Расположение 1-99 цепь A.
Класс: Альфаспиральные белки
Укладка IHF-like ДНК-связывающие белки
Суперсемейство IHF-like ДНК-связывающие белки
Семейство Прокариотические ДНК-связывающие белки
Это суперсемесемейство содержит два семейства
Одно суперсемейство имеет такую укладку
Домен1 Protein: Integration host factor Бета субъединица (IHFB) Организм E. Coli [TaxId: 562] [a.55.1.1]
Расположение 1-94 цепь B.
Класс: Альфаспиральные белки
Укладка IHF-like ДНК-связывающие белки
Суперсемейство IHF-like ДНК-связывающие белки
Семейство Прокариотические ДНК-связывающие белки
Это суперсемесемейство содержит два семейства
Одно суперсемейство имеет такую укладку
СATH
Домен 1IHFA 2-97 цепь A
4.10.520.10.3.1.1.1.4 архитектура Few Secondary Structures
топологиz Irregular cуперсемейство HU Protein; Chain A семейство HU Protein, subunit A Домен 1IHFA 1-94 цепь B
4.10.520.10.3.1.1.1.4 архитектура Few Secondary Structures
топология Irregular cуперсемейство HU Protein; Chain A семейство HU Protein, subunit A Это суперсемесемейство содержит два семейства
Одно суперсемейство имеет такую укладку
3. Небольшие различия в определении координат доменов, различия в номенклатуре одних и тех же таксонов. 4.
Выравнивание По данному выравниванию с помощью сервиса Geometrical core найдем геометрическое ядро с порогом 2 ангстрема.
| Pos. | 1CHD_A | 1U94_A |
| 82 | SER182 | GLY87 |
| 83 | PRO183 | LYS88 |
| 84 | ALA184 | THR89 |
| 85 | VAL185 | CYS90 |
| 86 | ILE186 | ALA91 |
| 87 | ILE187 | PHE92 |
| 88 | THR188 | ILE93 |
| 89 | GLN189 | ASP94 |
| 90 | HIS190 | ALA95 |
| 110 | SER210 | ASP110 |
| 111 | VAL211 | ILE111 |
| 161 | ASP261 | LEU126 |
| 164 | PHE264 | CYS129 |
| 165 | HIS265 | ASP130 |
| 166 | SER266 | ALA131 |
| 167 | VAL267 | LEU132 |
| 170 | HIS270 | SER135 |
| 171 | ALA271 | GLY136 |
| 183 | MET283 | ALA147 |
| 209 | TYR295 | ALA179 |
| 229 | VAL311 | GLN194 |
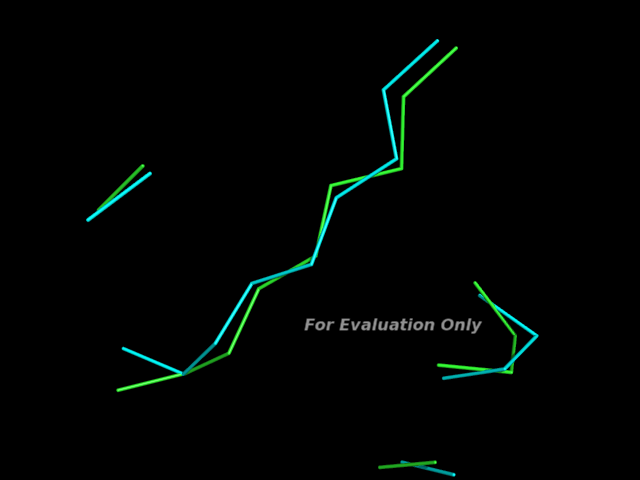
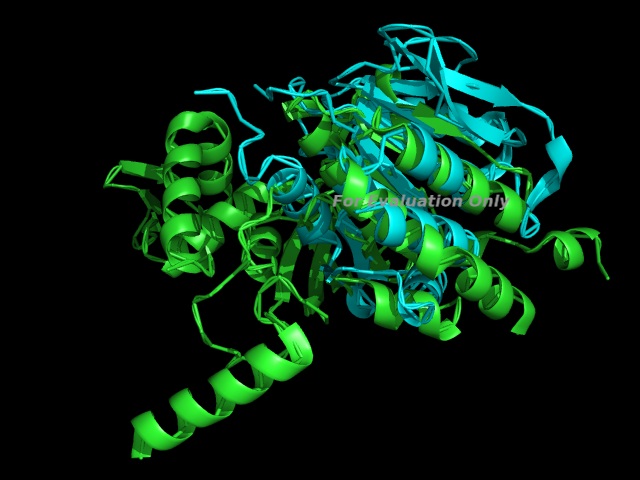
Домен белка 1u94 больше и часть его ни с чем не совместитась.
RMSD достаточно высоко(1.231).Т.е. домены, имеющие одинаковую топологию, но входящие в разные суперсемейства,имеют слабо выраженную сходную пространственную структуру.
| Главная | Второй Семестр |