
Третий семестр
| Главная |
| Первый семестр |
| Второй семестр |
| Pymol |
- Проанализируем выдачу программы PROCHECK для
своей записи PDB 1IHF.
Для этого воспользуемся базой данных
PDBSum и
со страницы своей структуры пройдем по гиперссылке
"PROCHECK" (слева).
Скопируем в отчёт карту Рамачандрана своей структуры.
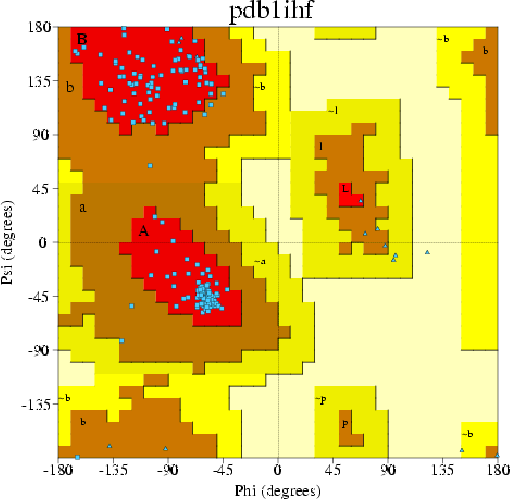
No. of residues %-tage ------ ------ Most favoured regions [A,B,L] 155 95.7% Additional allowed regions [a,b,l,p] 7 4.3% Generously allowed regions [~a,~b,~l,~p] 0 0.0% Disallowed regions [XX] 0 0.0% ---- ------ Non-glycine and non-proline residues 162 100.0% End-residues (excl. Gly and Pro) 73 Glycine residues 15 Proline residues 10 ---- Total number of residues 260
Ни один остаток не попал в разрешенную или запрещенную области.
Приведем процент остатков (отличных от Gly и Pro, они обозначены голубими треугольничками), попавших в предпочитаемые области на карте (красная область). 7 остатков, отличных от Gly и Pro, попали в допустимую область, два - в запрещенную. В предпочитаемой - 155 остатков. Таким образом, процент попавших в предпочитаемую область остатков 95,7% Это значение больше 90%, что является хорошим показателем структуры. - На сервере EDS
зайдем на страницу своей структуры. Пройдем по гиперссылке "Significant regions"
Большой Z-score по RSR имеет только три остатока цепи B: 92 ILE(изолейцин) RSR=0,262;
93 TYr(тирозин) RSR=0,216;
94 GLY(глицин) RSR=0,441; . Создадим изображение этих остатков, на котором был бы видна его модель и электронная плотность вокруг него (поверхность уровня 1 электр. плот.).
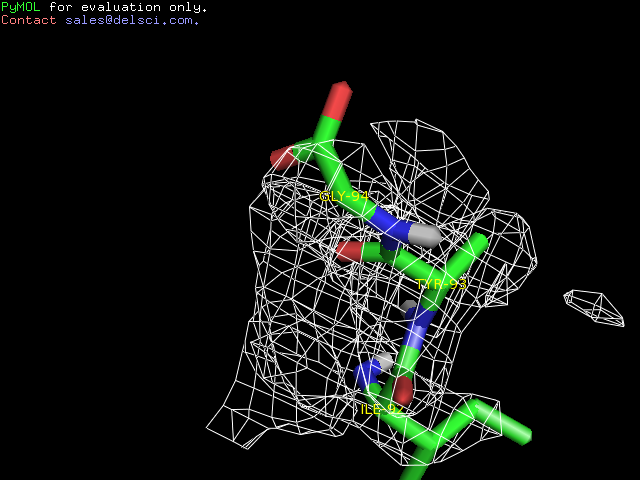
Наблюдаем отсутствие у остатка Тирозина всех атомов кроме С1.
Значения:пространственного R-фактора (RSR)=0,262; 0,216; 0,441; коэффициента корреляции для электронной плотности ("real-space correlation coefficient") =0,873; 0,651; 0,632 Z-score пространственного R-фактора для этого остатка=2,918; 2,312; 5,75.Простр. R-фактор >10%, а Z-score >0. Это говорит о том, что действительно расположение остатков плохое.Как видно из картинки, остаток очень плохо вписался в электронную плотностьУлучшились показатели: R, R-free, 1st generation packing quality, 2nd generation packing quality, Ramachandran plot appearance, Chi-1/Chi-2 rotamer normality, Backbone conformation, Bond length RMS Z-score, Bond angle RMS Z-score, Total number of bumps. Показатель Z-score ухудшился(он стал ближе к нулю по значению) и Unsatisfied H-bond donors/acceptors. В Full WHAT_CHECK reports: Error: Matthews Coefficient (Vm) very high
Главная Второй Семестр