Практикум 11.
1. Выбор и описание семействa доменов из Pfam.
Для анализа я выбрала семейство доменов tRNA ribose 2'-O-methyltransferase, aTrm56. По данным из таблицы:
- AC: PF01994
- ID: Trm56
- Seed: 81
- Full: 271
- 3D-structure: 4
- Domain Architectures: 2
Данные таблицы и Pfam немного разнятся. Например на Pfam seed: доменных архитектур 4, 3-d структуры 2.
Функция.
Данный домен катализирует 2'-O-метилирование остатка цитидина в тРНК архей. Этот фермент обнаружен во всех секвенированных на данный момент геномах архей, кроме одного, и принадлежит к семейству SPOUT (класс IV) РНК-метилтрансфераз.
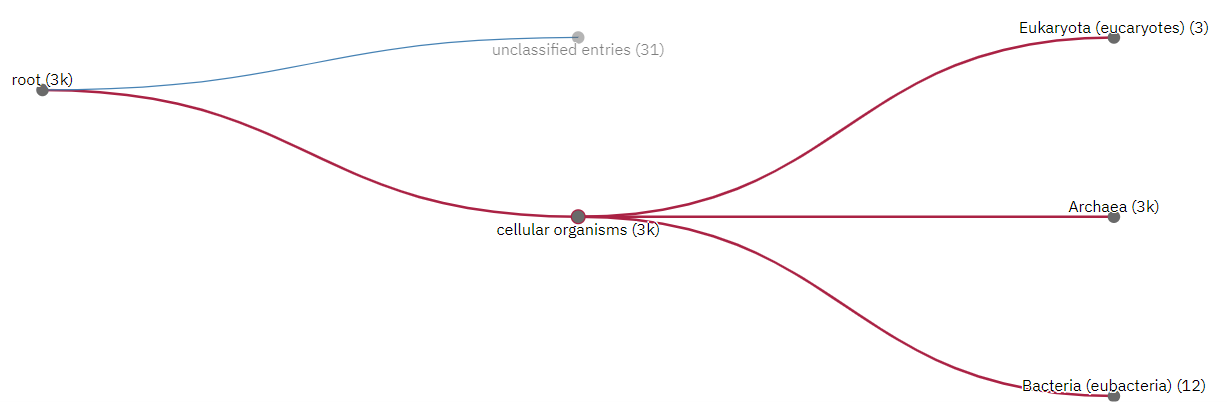
Таксономическая распространенность.
Всего в графе Taxonomy представлено 2000 таксонов. Среди них абсолютное большинство археи. В меньшей степени у бактерий и совсем чуть-чуть у эукариот.
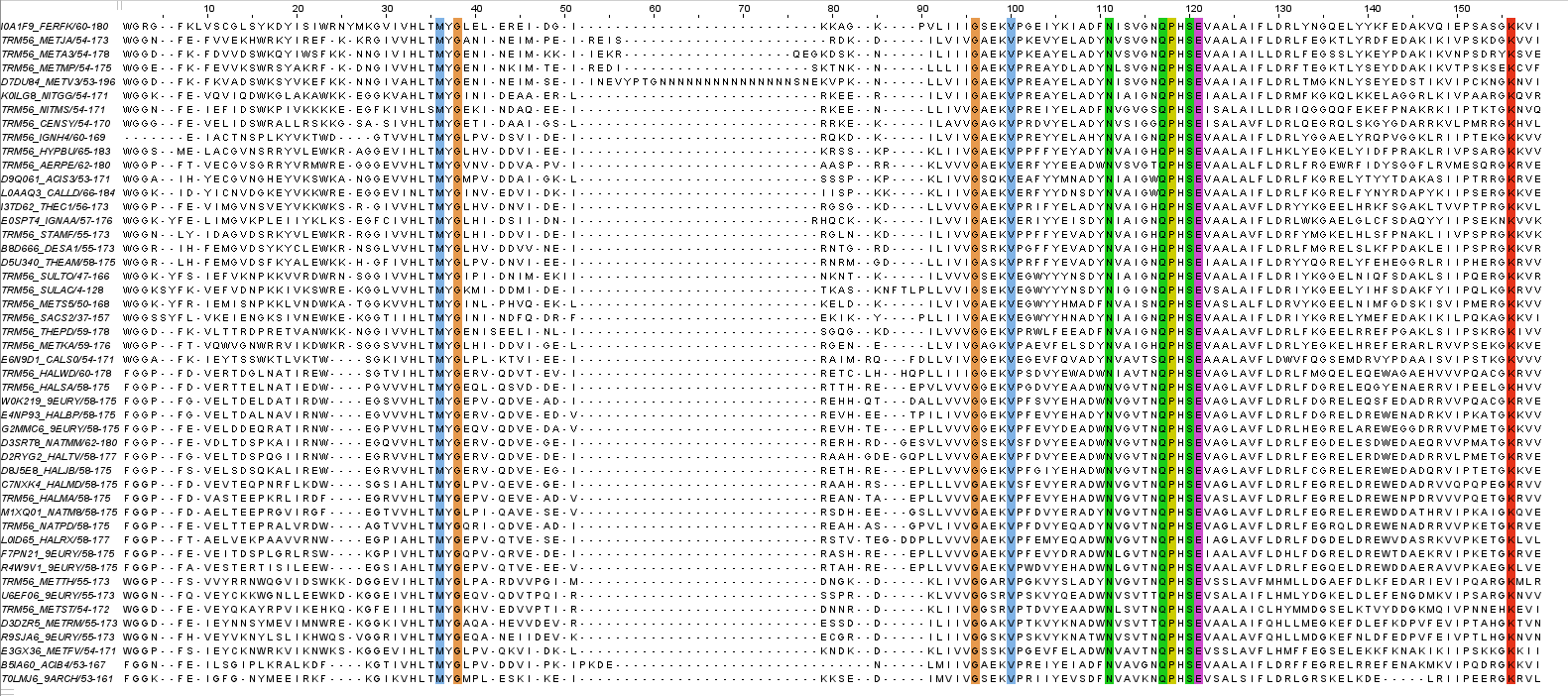
3. Выравнивание seed с точки зрения гомологичности всех последовательностей или их подмножества.
1.Максимально достоверный блок, включающий фрагменты из всех последовательностей.
Для этого задания я перевыровняла последовательности edit => remove all gaps; web service => alignment => Mafft with Defaults. Затем при окрашивании выравнивания через параметр Above identity threshold (100%) я получила следующее: максимальные достоверные блоки - 117-118,120-121 Также встречаются единичные аминокислоты, консервативные во всех последовательностях - 36, 38, 96, 100, 111, 156.
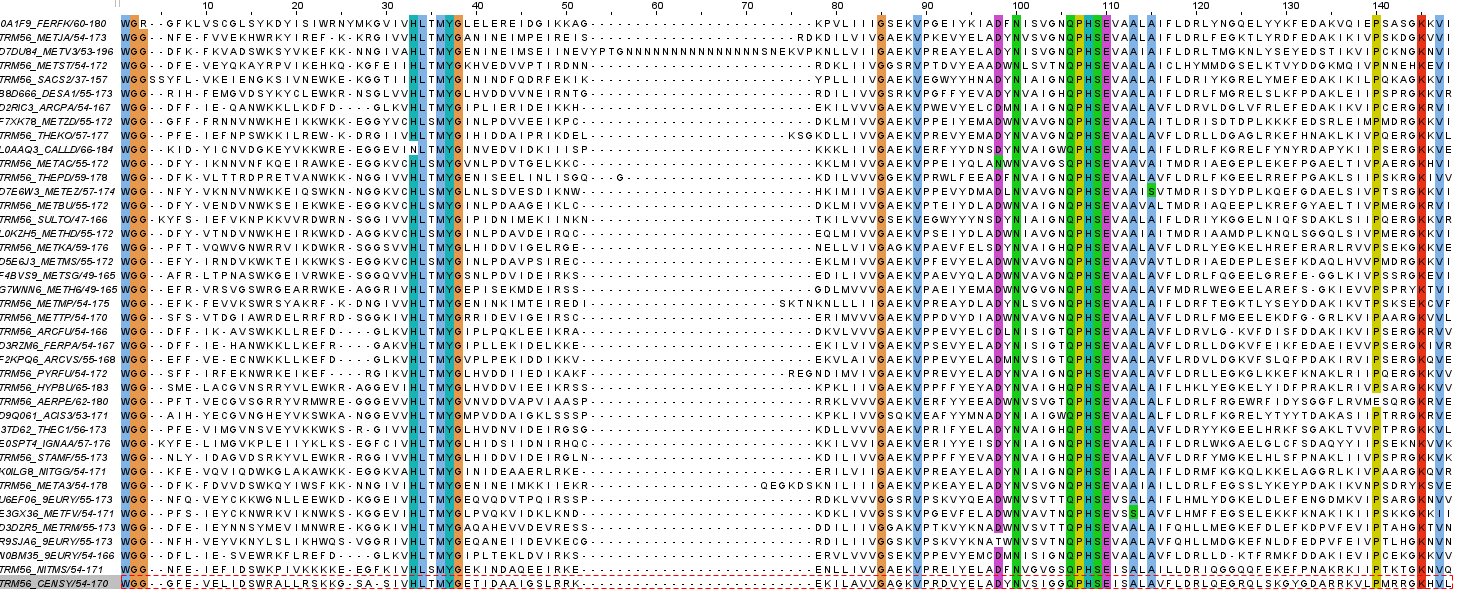
2.Максимально достоверный блок, включающий фрагменты НЕ из всех последовательностей.
Я выбрала наиболее схожие последовательности(41): таким же способом перевыровняла в новом окне. Максимально достоверные блоки - 106-110, 36-38, 1-2. Единичные аминокислоты, консервативные во всех последовательностях - 34, 85, 89, 100, 103, 105, 145, 147.
3.Участок выравнивания, на котором нет основания считать, что выравнивание не отражает ход эволюции.
Блоки 45-72 и 81-84 не отражают ход эволюции, на них нельзя выделить достоверных подблоков так как, в большинстве последовательностей стоят гэпы.
5. Карту локального сходства (dotplot) двух последовательностей с разной доменной архитектурой.
Для этого задания я выбрала домены PF01994 (белок AC:O29507) и PF01994 - PF01966 (белок AC:Q9HJN6), с параметром E-Value 0.05.
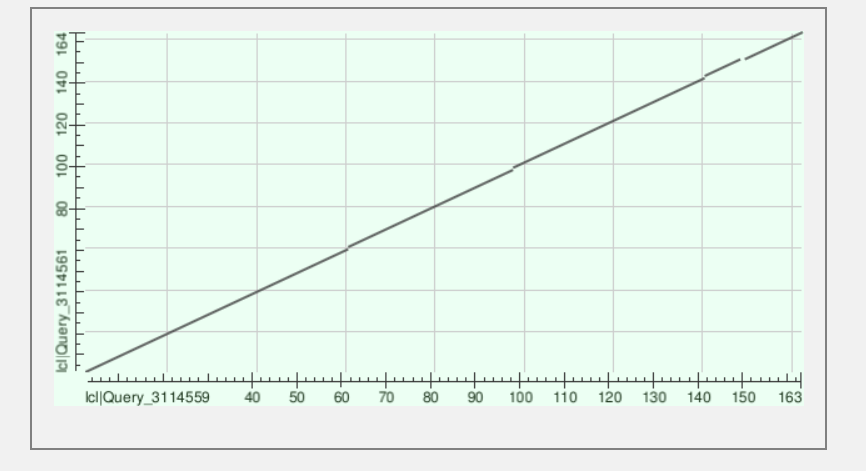
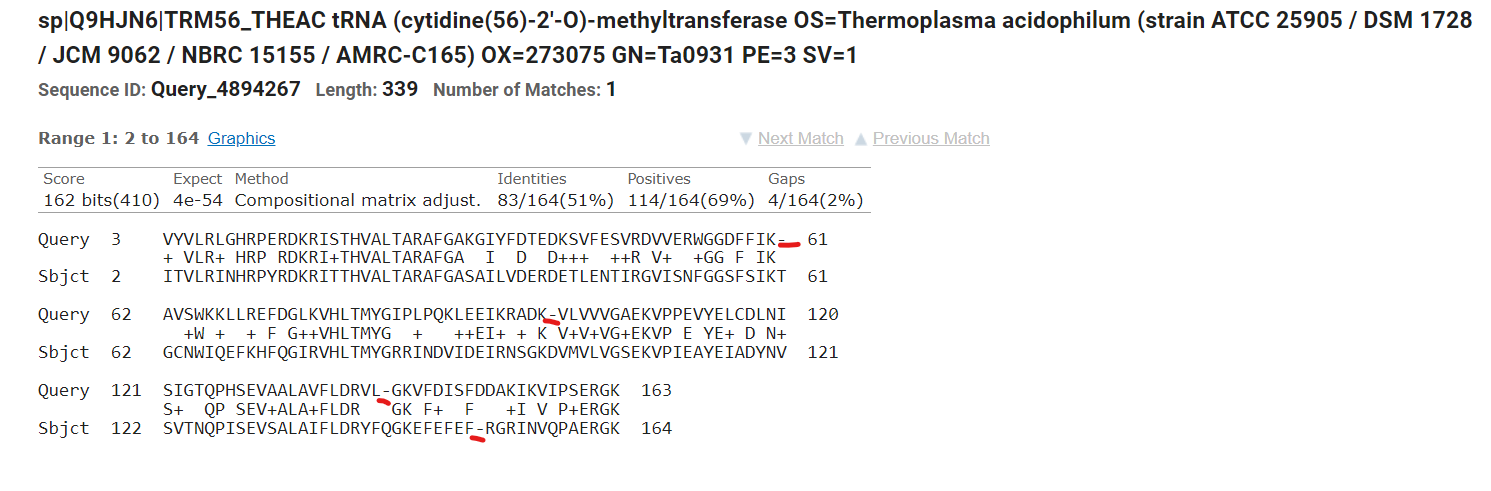
На диаграмме видны 4 разрыва, которые соответствуют инделям в выравнивании (подчеркнуты красным).